УДК 633.854.78:632.4.01/.08:575.174.015.3:577.2.08
ВНУТРИВИДОВОЙ ПОЛИМОРФИЗМ ВОЗБУДИТЕЛЯ ЛОЖНОЙ МУЧНИСТОЙ РОСЫ ПОДСОЛНЕЧНИКА, ВЫЯВЛЯЕМЫЙ МОЛЕКУЛЯРНЫМИ МАРКЕРАМИ
С.З. ГУЧЕТЛЬ, Т.С. АНТОНОВА, Т.А. ЧЕЛЮСТНИКОВА, С.А. РАМАЗАНОВА, Н.М. АРАСЛАНОВА
У 42 изолятов шести рас возбудителя ложной мучнистой росы Plasmopara halstedii (Farl.) Berl. et de Toni, поражающих подсолнечник в Краснодарском крае, с использованием 36 праймеров и их комбинаций в ПЦР изучали внутривидовой полиморфизм, выявляемый молекулярными маркерами. Степень полиморфизма оценена как слабая, поскольку лишь 10 праймеров и праймерных комбинаций показали наличие полиморфизма.
Ключевые слова: ложная мучнистая роса, подсолнечник, Plasmopara halstedii, расы, ПЦР, RAPD, SSR, маркеры, полиморфизм, дифференциация.
Ложная мучнистая роса подсолнечника (ЛМР), вызываемая облигатным грибным патогеном Plasmopara halstedii (Farl.) Berl. et de Toni из класса Oomycetes, — одно из наиболее вредоносных заболеваний этой культуры в мире. Практически во всех странах, занимающихся возделыванием подсолнечника, ежегодно возникают новые патогенные расы гриба. Если в 2002-2003 годах, по разным сведениям, насчитывалось 11-17 рас (1, 2), то в 2007 году описано 36 патотипов патогена (3).
Традиционно дифференциацию рас P. halstedii (подобно расам других патогенов) осуществляют по их способности поражать растения линий-дифференциаторов. Кроме того, достаточно эффективно используются ПЦР-методы — для установления степени генетической близости между расами и изолятами внутри вида, филогенетических взаимоотношений между видами (4, 5). Однако физиологические расы грибов в силу своего общего происхождения часто бывают сходны по молекулярно-генетиче-ским характеристикам и при анализе методами полимеразной цепной реакции, например с RAPD (random amplified polymorphic DNA) и SSR (simple sequence repeat) праймерами, проявляют высокую генетическую однородность (6, 7).
Среди изолятов P. halstedii, собранных в районах Германии, обнаружен полиморфизм по микро- и минисателлитам, не коррелирующий ни с географическим происхождением, ни с принадлежностью к физиологической расе. Вместе с тем праймерные комбинации генерировали амплифицированные фракции, позволяющие отличить один изолят от другого (8).
С помощью 30 RAPD-праймеров были дифференцированы 58 изолятов пяти патотипов P. halstedii, собранных во Франции, при использовании 21 RAPD-праймера изучена взаимосвязь между расами 100, 300, 700, 330, 710, 703, 730, 770 из разных стран и обнаружен слабый полиморфизм, не зависящий от расы и географического происхождения (7). При испытании 12 EST-маркеров (expressed sequence tags-derived marker) выявлена высокая степень генетической разнородности среди 32 изолятов возбудителя ложной мучнистой росы подсолнечника из Франции и России (9). Мы, применив RAPD-ПЦР (10), показали, что четыре расы (330, 700, 710 и 730) генетически близки при четком отличии расы 300 от остальных.
Целью нашей работы было изучение выявляемого молекулярными маркерами внутривидового полиморфизма возбудителя ложной мучнистой росы Plasmopara halstedii, который поражает подсолнечник в Краснодарском крае.
Методика. Объектом исследований служили 42 изолята шести рас возбудителя ложной мучнистой росы — 300, 310, 330, 700, 710 и 730 (соответственно 2, 1, 12, 3, 13 и 11 изолятов), собранные с пораженных растений подсолнечника в разных районах Краснодарского края в 2005-2007 годах (различия в числе изолятов по расам связаны с частотой их обнаружения в агроценозах Северного Кавказа) (11). Расовую принадлежность изолятов определяли в соответствии с предложенной номенклатурой (12). Искусственное заражение линий-дифференциаторов и сортов подсолнечника осуществляли по общепринятой методике погружением корней проростков в инокулюм зооспор (13).
ДНК выделяли из конидиального спороношения P. halstedii, используя модифицированный метод (14). Материал был собран с семядольных листьев проростков восприимчивого сорта подсолнечника ВНИИМК 8883, искусственно зараженных зооспорами каждого изолята отдельно.
Для ПЦР-анализа использовали 21 праймер: два минисателлита и восемь SSR-праймеров — M13, T3B, GATA, GTG5, TCC5, GAA6, GACA4, CA 8, CAC 5, GGAT4 (8), а также 15 их парных комбинаций — М13 + Т3B, GAA6 + GACA4, TCC5 + GAA6, T3B + GTG5, M13 + GAA6, GAA6 + GTG5, T3B + GAA6, TCC5 + GTG5, GACA4 + TCC5, GACA4 + M13, GACA4 + T3B, GACA4 + GTG5, TCC5 + M13, TCC5 + T3B, M13 + GTG5; два праймера ISSR (inter simple sequence repeat) — (СА)10G и (САС)7Т (15); SSR-праймеры Pv 39 и Pv 17, выявившие полиморфизм между физиологическими расами возбудителя ЛМР винограда (16); ITS 1, ITS 2 (internal transcribed spacer) и G1, G2, G3 (17); два RAPD-праймера — РТО и OPG 06 (Operon Technologies, Inc., США), выявившие ранее полиморфизм между физиологическими расами возбудителя ЛМР подсолнечника (18) (общее число праймеров и их комбинаций — 36).
ПЦР выполняли в реакционной смеси (25 мкл) следующего состава: 67 мМ Трис-HCl, рН 8,8; 16,6 мM сульфат аммония; 1,5-3,0 мM MgCl2; 0,01 % Твин 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК-полимеразы («Госниигенетика», Россия). Амплификатор Терцик («АО ДНК-технология», Россия), режим — начальная денатурация ДНК в течение 2 мин при 94 °С; 30-35 циклов с последовательной сменой температур: 60 с, 94 °С (денатурация), 60 с, 36-60 °С (в зависимости от праймера) (отжиг), 120 с, 72 °С (элонгация); финальная элонгация 4 мин при 72 °С.
Электрофорез продуктов амплификации проводили в агарозном геле (1,5 % агароза, 1,5xТАЕ-буфер, камера для горизонтального электрофореза) в течение 1,5-2,0 ч при I = 50 мА, U = 70-90 В. Реакционную смесь (10 мкл) наносили на гель вместе с красителем бромфеноловым синим. В качестве маркеров длины фрагментов ДНК использовали GeneRuler 1 kb DNA Ladder (UAB «Fermentas», Литва). Последующее окрашивание осуществляли бромистым этидием. Результаты электрофореза документировали при помощи трансиллюминатора и видеосистемы («АО ДНК-технология», Россия) с программным обеспечением Gel Imager 2. Реакции повторяли 2-3 раза, принимая за достоверные только воспроизводимые фракции. Изоляты различали по наличию или отсутствию в геле полос, соответствующих фрагментам ДНК определенной длины. Длину фрагментов оценивали с помощью программы Gel-Pro.
Кластерный анализ выполняли с использованием пакета программ Statistica 6.0 (для кластеризации применили метод Ward).
Результаты. Изученные изоляты перечислены в таблице.
| Характеристика изолятов Plasmopara halstedii, проанализированных с применением ПЦР-маркеров (Краснодарский край, 2005-2007 годы) | |||
Раса |
Название изолята |
Место сбора изолята |
Кластер, |
300 |
D8_5_05A |
Поля ВНИИМК |
Ia |
300 |
L_05_1A |
Лабинский район |
Ia |
310 |
A310_05B |
Поля ВНИИМК |
Ic |
330 |
В_05_8C |
Поля ВНИИМК |
IIe |
330 |
D116_05C |
Поля ВНИИМК |
IIe |
330 |
ВК_2_06C |
Поля ВНИИМК |
IIe |
330 |
D311_05C |
Поля ВНИИМК |
Ic |
330 |
А_64_05C |
Каневской район, станица Новоминская |
IIe |
330 |
А131_05C |
Каневской район, станица Новоминская |
IIe |
330 |
F1_2_05C |
Каневской район, фермерское поле |
IIe |
330 |
F110_05C |
Каневской район, фермерское поле |
IIe |
330 |
G_11_05C |
Каневской район |
IIe |
330 |
G_8_05C |
Каневской район |
IIe |
330 |
В_61_05C |
Станица Выселки |
Ic |
330 |
В_26_05C |
Станица Выселки |
IIe |
700 |
В_P2_05D |
Станица Выселки, поле 2 |
Ic |
700 |
NP94_06D |
Крыловской район, станица Новопашковская, поле 1 |
Ic |
700 |
L_4_05D |
Лабинский район |
Ib |
710 |
NP32_06E |
Крыловской район, станица Новопашковская, поле 3 |
Ib |
710 |
KG43_07E |
Крыловской район |
Ib |
710 |
GL_2_05E |
Поля ВНИИМК |
Ic |
710 |
В_05_13E |
Поля ВНИИМК |
Ic |
710 |
В_19_05E |
Станица Выселки |
Ic |
710 |
В_11_05E |
Станица Выселки |
Ic |
710 |
В_09_05E |
Станица Выселки |
Ic |
710 |
В08_05E |
Станица Выселки |
Ic |
710 |
В_89_05E |
Станица Выселки |
Ic |
710 |
В_85_13E |
Станица Выселки |
Ib |
710 |
В_07_05E |
Станица Выселки |
Ib |
710 |
L_2_05E |
Лабинский район |
Ib |
710 |
L_3_05E |
Лабинский район |
Ib |
730 |
D216_05F |
Поля ВНИИМК |
IId |
730 |
GL_3_05F |
Поля ВНИИМК |
IIe |
730 |
ВК_2_05F |
Поля ВНИИМК |
Ic |
730 |
В_25_05F |
Станица Выселки |
Ib |
730 |
В_32_05F |
Станица Выселки |
Ib |
730 |
В_28_05F |
Станица Выселки |
Ic |
730 |
В_43_05F |
Станица Выселки |
Ic |
730 |
В_33_05F |
Станица Выселки |
Ic |
730 |
В_18_05F |
Станица Выселки |
Ic |
730 |
В_46_05F |
Станица Выселки |
IId |
730 |
В_29_05F |
Станица Выселки |
IIe |
П р и м е ч а н и е. Последние буквы в названии изолятов (A, B, C, D, E, F) — обозначение рас (соответственно 300, 310, 330, 700, 710 и 730); I, II — кластеры; a, b, c, d, e — субкластеры. |
|||
При скрининге праймеров для последующей дифференциации между пятью случайно отобранными изолятами, представляющими наиболее распространенные расы (300, 700, 330, 710 и 730, рис. 1), GATA, (CA)10G, (CAC)7T и ITS 1 не гибридизовались с матричной ДНК; 24 (праймеры и праймерные комбинации) полиморфизма не выявили; 10, или всего 28 % от испытанных (PTO, OPG 06, T3B, М13, TCC5, GTG5, GAA6, GAA6 + GACA4, T3B + GAA6, TCC5 + T3B), выявили полиморфизм, что свидетельствует о слабой внутривидовой изменчивости P. halstedii, поражающей подсолнечник в Краснодарском крае. С использованием праймеров, выявленных в результате скрининга, анализировали все остальные изоляты гриба. При этом обнаружили 58 фракций ДНК, в том числе 40 полиморфных, характеризующихся наличием/отсутствием фрагментов определенной длины (от 2 до 11 полиморфных локусов на праймер). Уровень полиморфизма составил 0,68, амплифицированные фрагменты имели (в зависимости от праймера) размер от 80 п.н. до 2500 п.н.
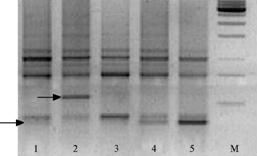 |
Рис. 1. Электрофоретические спектры продуктов амплификации ДНК с праймером Т3В у рас Plas-mopara halstedii: 1-5 — соответственно расы 300, 330, 700, 710 и 730; стрелками отмечены полиморфные фрагменты ДНК длиной 560 п.н. и 360 п.н. (сверху вниз); М — маркер молекулярной массы ДНК 1 kb (UAB «Fermentas», Литва) (Краснодарский край, 2005-2007 годы). |
Мы не выявили локусов ДНК, позволяющих четко различать расы между собой. Более того, некоторые изоляты рас 330, 710 и 730 оказались сходными (рис. 1, 2). Из них идентичными, в основном, были изоляты, принадлежащие к одной расе. Лишь в одном случае сходный спектр ампликонов показали два изолята, принадлежащие к разным расам, — В-19-05E (раса 710) и В-Р2-05D (раса 700). Генетическое сходство этих изолятов нельзя объяснить общей локализацией, поскольку они собраны с разных полей Выселковского района. Воз-можно, изолят расы 700 на самом деле принадлежал к расе 710, так как индикация с помощью линий-дифференциаторов не всегда надежна.
В то же время выявлялись фрагменты амплифицированной ДНК, уникальные для некоторых изолятов. Так, фрагменты G061135, G061225, (T3B + GAA6)553 и T3B521 были уникальны соответственно для изолятов NP94_06D, KG43_07E, B_85_15E и B_43_05F, причем не наблюдалось связи между уникальностью генотипа и его расовой принадлежностью.
|
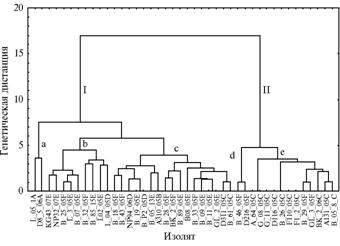
Рис. 2. Дендрограмма генетического сходства изолятов Plasmopara halstedii на основе полиморфных амплифицированных фрагментов ДНК: I и II — кластеры, a, b, c, d, e — субкластеры; описание изолятов см. в таблице (Краснодарский край, 2005-2007 годы). |
Представление данных ПЦР-анализа в виде матрицы состояний би-нарных признаков (наличие в электрофоретических спектрах одинаковых по размеру фрагментов ДНК — 1 и отсутствие — 0), выполнение на основе мат-рицы кластерного анализа и построение дендрограммы гене-тических взаимоотношений между изученными изолятами (см. рис. 2) показало, что они разделились на два кластера — I и II с генетической дистанцией между ними 17,50. Кластер I включал три субкластера (a, b и c), кластер II — два (d и e). Кластер I объединил изоляты рас 300, 310, 700, 710, пять изолятов расы 730 и два изолята расы 330. Оба изолята расы 300 выделились в отдельный субкластер а со степенью дистанцирования 7,80 от объединенного кластера, включающего остальные генотипы. Этот факт также подтверждает наше мнение, что раса 300 генетически удалена от остальных в силу происхождения с другого континента, в то время как остальные имеют общее происхождение, вероятно от бывшей европейской расы 1 (100) (10, 18). Изоляты рас 700, 710 и 730 равномерно распределились по субкластерам b и с, генетическая дистанция между которыми составила 5,18.
Кластер II объединил в основном изоляты расы 330 (в субкластере е) и четыре изолята расы 730, не попавшие в кластер I (см. рис. 2, см. табл.). Такое распределение расы 730 может свидетельствовать о ее происхождении от гибридизации рас 710 и 330.
Объединение изолятов в кластеры в большей степени зависело от принадлежности к определенной расе, чем от места сбора изолята.
Мы показали, что мини- и микросателлитные локусы более полиморфны в геноме P. halstedii, чем локусы, выявленные с помощью RAPD-ПЦР. В наших предыдущих исследованиях из 22 использованных RAPD-праймеров 13 позволили отличить расу 300 от остальных и лишь один выявлял изменчивость у всех изученных рас возбудителя ЛМР, поражающей подсолнечник в Краснодарском крае (10, 18).
Таким образом, изученные изоляты расы 300 возбудителя ложной мучнистой росы (мера дистанцирования между изолятами в дендрограмме 3,50) и большинство изолятов расы 330 (мера дистанцирования 4,80) объединяются в отдельные субкластеры (соответственно Ia и IIe), проявляя большую степень внутрирасового сходства. Изоляты рас 310, 700 и 710 находятся в субкластерах b и с кластера I. Наибольшей генетической гетерогенностью обладают изоляты расы 730, поскольку анализ молекулярных характеристик позволил отнести их к разным кластерам и субкластерам. Вероятно, все эти данные можно объяснить новообразованием рас на территории Краснодарского края. Нечеткое распределение в отдельные кластеры и субкластеры свидетельствует, что образование современных рас произошло недавно и они генетически не обособлены друг от друга.
Л И Т Е Р А Т У Р А
1. V e a r F., T o u r v i e i l l e d e L a b r o u h e D., M i l l e r J.F. Inheritance of the wide-range downy mildew resistance in the sunflower line RHA 419. Helia, 2003, 26(39): 19-24.
2. V i r a n y i F. Downy mildew research in sunflower: facts and consideration (based on reports of to the FAO sub-group, Plasmopara halstedii for 1999-2001). Proc. FAO technical meeting, 7-9.10.2002, ENSAM-INRA, Montpellier, France, 2002: 10-15.
3. G u l y a T.J. Distribution of Plasmopara halstedii races from sunflower around the world. Proc. 2nd Int. Downy Mildew Symposium «Advances in downy mildew research». Olomouc, Czech Republic, 2007, 3: 121-134.
4. Г а г к а е в а Т.Ю., Г а н н и б а л Ф.Б., Л е в и т и н М.М., Y l i - M a t t i l a T. Выделение и идентификация токсикогенных видов грибов из родов Fusarium и Alternariaс использованием полимеразной цепной реакции. Мат. II Всерос. съезда по защите растений. СПб, 2005, I: 150-151.
5. Б у л а т С.А., М и р о н е н к о Н.В. Идентификация грибов и анализ их генетической изменчивости методом полимеразной цепной реакции (ПЦР) с геноспецифичными и неспецифичными праймерами. Генетика, 1996, 32(2): 165-183.
6. B a l m a s V., S h e r m B., M i g h e l i Q. Characterization of isolates of Phoma tracheiphilia by RAPD-PCR, microsatellite-primers PCR and RDNA ITS1/ITS2 sequencing. J. Plant Pathol., 2003, 85(4): 312-315.
7. R o e c k e l - D r e v e t P., T o u r v i e i l l e J., G u l y a T.J., C h a r m e t G., N i-
c o l a s P., T o u r v i e i l l e d e L a b r o u h e D. Molecular variability of sunflower downy mildew, Plasmopara halstedii from different continents. Can. J. Microbiol., 2003, 49(8): 492-502.
8. I n t e l m a n n F., S p r i n g O. Analysis of total DNA by minisatellite and simple-sequence repeat primers for the use of population studies in Plasmopara halstedii. Can. J. Microbiol., 2002, 48: 555-559.
9. G i r e s s e X., T o u r v i e i l l e d e L a b r o u c h e D. , R i c h a r d - C e r v e r a S., D e l m o t t e F. Twelve polymorphic expressed sequence tags-derived markers for Plasmopara halstedii, the causal agent of sunflower downy mildew. Molec. Ecol. Notes, 2007, 7: 1363-1365.
10. Г у ч е т л ь С.З., Ч е л ю с т н и к о в а Т.А., И в е б о р М.В., А н т о н о в а Т.С., А р а с л а н о в а Н.М., Р а м а з а н о в а С.А. Применение RAPD-ПЦР маркеров для дифференциации физиологических рас Plasmoparahalstedii(Farl.) Berl. et de Toni, поражающих подсолнечник в Краснодарском крае. С.-х. биол., 2008, 5: 82-87.
11. A n t o n o v a T., I w e b o r M., A r a s l a n o v a N. Races of Plasmopara halstedii on sunflower in separate agrocenosises of Adigeya Republic, Krasnodar and Rostov region in Russia. Proc. 17th Int. sunflower conf. Cordoba, Spain, 2008, V1: 85-91.
12. T o u r v i e i l l e d e L a b r o u h e D., G u l y a T.J., R a s h i d Y.K, V i r a n y i F. New nomenclature of race of Plasmopara halstedii (sunflower downy mildew). Proc. 2th Int. sunflower conf. Toulouse, France, 2000, 2: I-61-I-66.
13. А н т о н о в а Т.С., А р а с л а н о в а Н.М., Г о л о в и н А.В., Ч е л ю с т н и к о-
в а Т.А., С а у к о в а С.В. К вопросу о расовой принадлежности возбудителя ложной мучнистой росы на подсолнечнике на Северном Кавказе. Науч.-техн. бюл. ВНИИМК, 2000, 123: 16-20.
14. Z o l a n M.E., P u k k i l a P.J. Inheritance of DNA methylation in Corpinus cinereous. Mol. Cell Biol., 1986, 6(1): 195-200.
15. Г л а з к о В.И., Д у б и н А.В., К а л е н д а р ь Р.Н., Г л а з к о Г.В., Ш е р е п и т ь-
к о В.И., С о з и н о в А.А. Генетические взаимоотношения между сортами сои, оцененные с использованием ISSR маркеров. Цитология и генетика, 1999, 33(5): 47-51.
16. D e l m o t t e F., C h e n W.J., R i c h a r d - C e r v e r a S., G r e i f C., P a p u r a D., G i r e s s e X., M o n d o r - G e n s o n G., C o r i o - C o s t e t M.-F. Microsatellite loci from the grape downy mildew (Plasmopara viticola). Mol. Ecol. Notes, 2006, 6: 379-381.
17. S a y s - L a s a g e V., R o e c k e l - D r e v e t P., V i g u i e A., T o u r v i e i l l e J., N i c o l a s P., T o u r v i e i l l e d e L a b r o u h e D. Molecular variability within Diaporthe/Phomopsis helianthi from France. Phytopathology, 2002, 92: 308-313.
18. A n t o n o v a T., G u c h e t l S., I w e b o r M., T c h e l u s t n i k o v a T. Differences in some RAPD-loci DNA of Plasmopara halstedii races affected sunflower in Krasnodar region of Russia. Proc.17th Int. sunflower conference. Cordoba, Spain, 2008, 1: 91-97.
INTRASPECIFIC POLYMORPHISM IN SUNFLOWER FALSE MILDEW REVEALED BY MOLECULAR MARKERS
S.Z. Guchetl’, T.S. Antonova, T.A. Chelyustnikova, S.A. Ramazanova, N.M. Araslanova
The intraspecific polymorphism in 42 isolates of six races of sunflower false mildew (Plasmopara halstedii (Farl.) Berl. et de Toni) in Krasnodar region, revealed by molecular markers with 36 primers and its combinations was studied with the use of PCR. The degree of polymorphism was estimated as weak, so long as only 10 primers and primer combinations show polymorphism.
Key words: downy mildew, sunflower, Plasmopara halstedii, races, RAPD, SSR, markers, polymorphism, differentiation.
ГНУ Всероссийский НИИ масличных культур |
Поступила в редакцию |