* Работа выполнена при частичной финансовой поддержке гранта РФФИ 08-04-90104-Мол_а.
|
Б 1 MDLTPSFNSLDSNGIPNSPDVDPALGETGGSEGLQKIDGESQLDWDSIIVSDVLDDEGRV 61 QVPTENEIYFNLGLNKGDEAANNRFSGSGTNCHAQGSLDTDNEDHHADQPCQDYIPDEKR 121 VVYNRMNPSMQPGCLFPNMKEFRIAMRQYAIKHEFELGIEVTSTTRYVGYCKGGDCPWRI 181 YAREEKKGLPTIVVAVLDDVHTCTSSGRRRTTTPTCGWVAFHAKPLLMKKPQMGAKELQQ 241 TLQTTHNVTIGYDTVWKGKEKALRELYGSWEESFQLLYSWKEAVIAVMPDSVIEIDVILE 301 DGKYYFSRFFCAFGPCISGFRDGCRPYLSVDSTALNGRWNGHLASATGVDGHNWMYPVCF 361 GFFQAETVDNWIWFMKQLKKVVGDMTLLAICSDAQKGLMHAVNEVFPYAERRECFRHLMG 421 NYVKHHAGSEHMYPAARAYRRDVFEHHVSKVRNVHKIAEYLDQHHKFLWYRSGFNKDIKC 481 DYITNNMAEVYNNWVKDHKDLPVCDLAEKIREMTMELFHRRRRIGHKLHGIILPSVLAIL 541 KARTRGLGHLSIVKCDNYMAEVRDSTNCMTKHVVNAELKQCSCEEWQHTGKPCQHGLALI 601 IAQDSRDVGMENFVDDYYSTERFKIAYSRRVEPIGDRSFWPSVDFASGVFAPIARRGLGR 661 QRKNRIKSCLEGGSARNKSTNENEKTKKRLKRQYTCPNCGELGHRQSSYKCPLNGTKKRK 721 RKPRINTTKNWIPKELRTSSQNVPVQPDVAEEVTEQELEDPQPETEQLGLALFQPLGAQI 781 TEQEADEPAEQAPPASPPPTRKWLVKKITPKKRLRISAQQKQY |
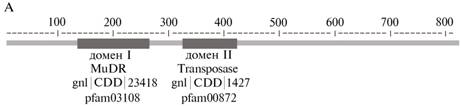
Рис. 1. Структурные домены в пределах аминокислотной последовательности (А) и транспозазы mudrА кукурузы gb|ААА21566 (Б). Области последовательностей, образующих домены, подчеркнуты. |
MudrA Z. mays, использованная в качестве исходной последова-тельности для поиска (query). Под CD-доменом понимаются определенные функциональные и/или структурные участки полипептидной цепи: MuDR-домен, специфичный для соответствующего семейства растительных транспозаз, и Transposase до-мен, общий для растительных и бактериальных транспозаз (рис. 1).
Домен gnl|CD|23418 найден в растительных белках, которые являются транспозазами для мобильных элементов семейства Mutator. Такие транспозоны содержат две рамки считывания. Молекулярная функ-ция этого домена неизвестна. gnl|CD|23418 pfam03108, MuDR, MuDR family transposase Query: 133 GCLFPNMKEFRIAMRQYAIKHEFELGIEVTSTTRYVGYCKGGDCPWRIYAREEKKGLPTI 192 Sbjct: 31 GQVFKSKEDCQILLAIYAIKNKFHFKVTKSDPKRYVLECRDEGCLWRVRATRVKES-DCF 89 Query: 193 VVAVLDDVHT-CTSSGRRRTTTPTCGWVA--FHAKPLLMKKPQMGAKELQQTLQTTHNVT 249 Sbjct: 90 EIRKYNSEHTCSVTERSNRHRQATPRVIGSVLKEK-YSGQFPGPRPKDLMELVLEDLGVK 148 Query: 250 IGYDTVWKGKEKAL 263 Sbjct: 149 ISYSKAWRAKEFAV 162 gnl|CD|1427, pfam00872, Transposase_mut, Transposase, Mutator family Query: 326 PYLSVDSTALNGRWNGHLAS-----ATGVDGHNWMYPVCFGFFQAETVDNWIWFMKQLKK 380 Sbjct: 163 PYLFLDATYVKVREEGRVVSKAVLIALGVTADGRREILGIEVGDGESAAFWLTFLDDLKA 222 Query: 381 VVGDMTLLAICSDAQKGLMHAVNEVFPYAERRECFRHLMGNYVKH 425 Sbjct: 223 RGLQGVLLVV-SDGHKGLVAAIRAVFPGASWQRCRVHFLRNLLAA 266 |
||||||||||||
Рис. 2. Парный элайнмент областей mudrA, содержащих структурные домены, и соответ-ствующих им консенсусных последовательностей. |
||||||||||||
Информация о существующих доменах содержится в виде консен-сусной последовательности (consensus sequence), то есть последователь-ности, смоделированной таким образом, что она составлена из наиболее консервативных аминокислот, определенных множественным элайнментом.
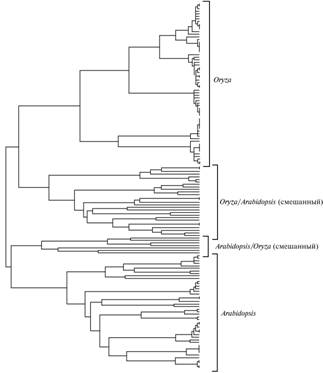
В области, включающей домен transposase, в каждом из геномов с помощью программы BLAST (базы данных NCBI и MIPS) было обнару-жено более 100 последовательностей. Элайнмент этих последовательностей (рис. 2) и его оценка дендрограммой UPGMA («unrooted tree») (программа TREECON) позволили выявить четыре главных кластера (рис. 3): кластер Oryza, соединенный со смешанным кластером Oryza/Arabidopsis, и кластер Arabidopsis, соединенный со смешанным кластером Arabidopsis/Oryza. По-строенная дендрограмма характеризует эволюционное расстояние между по-следовательностями, но не указывает, какие из кластеров последовательно-стей более древние. Однако можно предположить, что последовательности, образующие смешанные кластеры, соответствуют древним признакам, сфор-мировавшимся до дивергенции цветковых растений на классы двудольных и однодольных, а индивидуальные кластеры Oryza и Arabidopsis — более позд-ним признакам, которые появились после этой дивергенции (в том числе специфичные для каждого из сопоставляемых видов). Естественно, это пред-положение нуждается в дополнительных доказательствах.
При построении эволюционного древа была выбрана последова-тельность, которая заведомо несет более древние признаки (outgroup se-quence), что позволяет «укоренить» древо. При этом при построении древа учитывали не только статические взаимоотношения между последователь-ностями и кластерами, но также определяли направления эволюции и, следовательно, выясняли, какой из двух классов последовательностей несет более древние признаки, унаследованные от предшественника.
В качестве outgroup sequence можно было бы использовать транс-позазу семейства Мu-элементов растений, филогенетически более древних, чем цветковые, однако информация о последовательностях мобильных эле-ментов нецветковых растений в международных базах данных отсутствует. Также возможно выбрать в качестве outgroup sequence бактериальную транс-позазу, но при этом возникает ряд трудностей. Так, бактериальные транспо-зазы эволюционно удалены от растительных. Степень сходства индивиду-альных последовательностей бактериальных и растительных транспозаз, об-наруживаемая парным элайнментом, оценивается количеством идентичных аминокислот и консервативных замен, которое лишь частично обусловлено общими древними признаками. Неопределенная часть обнаруживаемых при парном сопоставлении последовательностей идентичностей и консерватив-ных замен определяется случайным совпадением новообразованных вторич-ных признаков. Подобные совпавшие признаки маскируют искомые древние признаки, специфичные для транспозаз вне зависимости от их источника, в связи с чем эволюционный анализ, основанный на случайно выбранной в качестве outgroup sequence бактериальной транспозазе, может оказаться не-достоверным. Проблема осложняется областью достоверной гомологии бакте-риальных и растительных транспозаз, ограниченной лишь доменом II (см. рис. 1), который и несет искомые, но не определяемые при парном сопо-ставлении древнейшие признаки. Наконец, обилие бактериальных транспозаз в международных банках данных затрудняет выбор адекватной последо-вательности, отражающей признаки реально существовавшего предшествен-ника растительных транспозаз.
Иными словами, в нашем распоряжении имеются сотни после-довательностей бактериальных траспозаз, среди которых требуется найти единственную, использование которой в качестве outgroup sequence дол-жно определить направление эволюции среди сотен известных расти-тельных транспозаз.
|
Рис. 3. Дендрограмма, описывающая взаимоотношения первичных структур транспозаз геномов Arabidopsis и Oryza. Использованная коллекция транспозаз ограничена последова-тельностями, элайнмент которых достаточно достоверен в со-поставляемой области, ограниченной доменом II. |
Исходя из по-следовательности рас-тительной транспозазы с заведомо древними признаками, с помо-щью парного элайн-мента можно устано-вить бактериальную outgroup sequence и наоборот. Поскольку направление эволюции растительных транспо-заз неизвестно, любая из них может быть ис-пользована для иденти-фикации бактериаль-ной транспозазы. В этом случае найден-ная транспозаза будет лишь казаться наибо-лее родственной из-за совпадения древних первичных и случай-ных вторичных приз-наков. Вследствие это-го использование для поиска различных слу-чайно выбранных рас-тительных транспозаз приводит к идентификации по меньшей мере нескольких бактериальных транспозаз, выбранных произвольно. Поэтому поиск адекватной outgroup sequence среди широкого спектра бактериальных последовательностей дол-жен опираться на использование той из растительных транспозаз, которая несет наиболее древние признаки. Между тем, именно это и составляет конечную цель исследования. Подобное противоречие можно преодолеть, использовав транспозазы из двух смешанных кластеров, a priory оце-ненных как древние, но и в таком случае выбор между этими двумя кластерами остается произвольным.
Для решения задачи идентификации единственной бактериальной последовательности, объективно пригодной в качестве outgroup sequence, использовали следующий прием (см. рис. 3). Программа NCBI позволяет установить консенсусную последовательность, характеризующую усреднен-ные общие признаки каждого из положений множественного элайнмента для большого числа бактериальных транспозаз. Консенсусная последова-тельность отражает консервативные признаки бактериальных транспозаз и, следовательно, предполагаемые консервативные признаки предшествен-ника растительных транспозаз. Поэтому использование в поиске не инди-видуальной (соответственно, в той или иной мере случайно выбранной) бактериальной транспозазы, а консенсусной последовательности должно привести к идентификации группы (кластера) растительных транспозаз, действительно несущих наиболее древние признаки.
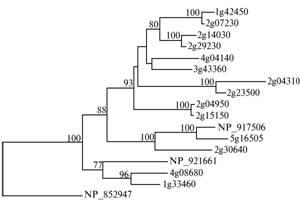
При поиске наиболее родственных транспозаз растений с помощью установленной консенсусной последовательности оказалось, что такие по-следовательности принадлежат кластеру Arabidopsis/Oryza (см. рис. 3), то есть из двух кластеров, оцененных как древние, более древним является именно этот смешанный кластер. Для каждой из содержащихся в нем по-следовательностей с помощью программы BLAST были определены наи-более схожие последовательности бактериальных транспозаз. Как показал про-веденный анализ, наиболее родственна всем последовательностям кластера транспозаза бактерии Mycoplasma gallisepticum (NP_852947). Она и исполь-зовалась нами в дальнейшем исследовании избранных транспозаз Arabi-dopsis и Oryza (рис. 4, 5).
|
Рис. 4 Эволюционное древо, описывающее взаимо-отношения первичных структур транспозаз геномов Arabidopsis и Oryza (соответственно NP_917506 и NP_921661). Использованная коллекция транспозаз A. thaliana ограничена последовательностями, элайнмент которых достаточно достоверен в сопоставляемой об-ласти, ограниченной доменом II. В качестве outgroup sequence используется бактериальная транспозаза Myco-plasma gallisepticum (NP_852947). |
Характерно, что оба смешанных кластера, пред-положительно оцененные как древние, действительно оказались таковыми среди выбранных последователь-ностей Arabidopsis. Отме-тим, что приведенные по-следовательности Arabidop-sis выделились как харак-терные представители сме-шанных кластеров и инди-видуального кластера Ara-bidopsis (см. рис. 2), обра-зующего несколько под-кластеров. Статистическая поддержка в пределах это-го индивидуального клас-тера невысока по двум причинам: первая — узкие границы элайнмента, до-ступные для анализа, вторая — последовательности индивидуального кла-стера Arabidopsis и бактериальной транспозазы эволюционно слишком далеки.
Bacteria consensus (query) |
Рис. 5. Схема эксперимента по обоснованию использования в качестве outgroup sequence последовательностей транспозаз Mycoplasma NP_852947 и Oryza NP_852947. |
Попутно отметим, что в целом последова-тельности транспозаз Ara-bidopsis вариабельны, и для достоверного установления их эволюционных взаимо-отношений анализируемая область элайнмента дол-жна быть достаточно про-тяженной, вследствии чего мы увеличили длину исследуемых последователь-ностей, включив домен I MuDR, характерный только для растительных транспозаз, а также область между доменами I и II. При этом необходимо выбирать в качестве outgroup sequence другую последовательность, находящуюся на меньшем эволюци-онном расстоянии и несущую древние признаки. Методом парного элайн-мента установили, что таким условиям удовлетворяет последовательность, принадлежащая самому древнему кластеру Orysa — NP_921661.
А AAA21566 RIAMRQYAIKHEFELGIEVTSTTRYVGYCKG-GDCPWRIYAEEKKGLPTIVVAVLDDVHTCTSSGRRR-TT At1g42450 TTKLAIHPIRRKFHFIYAKSCPNIVFAMCVS-HTCLWRVYATELEEIDRFEIKCATQLHTCSVDARGDFHK At2g14030 NVKLAVHALNSRFHFRRDRSYKKLMTLTCIS-ELCLWRVYIVKLEDSDNYQIRSATLEHTCTVEERSNYHR At4g04140 RIALSIYAINRIFRFKFTRYEKHYLVAECYD-KKCDWRVRAHQVGGDSEYEVRLAKLEHVCKVQTRSRFSK At2g04310 HEVVDRAAFANSFGYVIKKSDKERYVLKCAK-ESCSWRLRASNISNTDIFSIRRYNKMHSCTRLSKSSSRR At2g04950 VFTLRMSAVKHSFEFHTVKSDLTKYVLHCID-ENCSWRLRATRAGGSESYVIRKYVSHHSCDSSLRNVSHR At5g16505 RRTLKDLAIALHFDLRIVKSDRSRFIAKCSK-EGCPWRIHAAKCPGVQTFTVRTLNSEHTCEGVRDLH-HQ At4g08680 KEAILEVTLKHGHNVVQDRWEKEKISFKCGMGGKCKWRVYCSYDPDRQLFVVKTSCTWHSCSPNGKCQILK AAA21566 TPTCGWVAFHAKPLLM-KKPQMGAKELQQTLQTTHNVTIGYDTVWKGKEKALRELYGSWEESFQLLYSWKE At1g42450 QASIAVIGKLVRTKYLGVGRGPRPNELRKMLRDEFNLNVSYLKAWRAQEIAMDNAMRSAMGSYTLIQPYFK At2g14030 GATTRVIRSIIKSKYDGNTRGPRAVDLQRILLTDYSVRISYWKAWKSREIALESVQGSATNSFSLLTAYIH At4g04140 HATSKVVAALLRAKYAKAFCGPRARDLPDSLLREHNVRMTYWKCWKAKELAVETAQGTDESSFSLLPVYLH At2g04310 KGNPQLVAALLHDHFPGQLETPVPRIIMELVQTKLGMKVSYSTALRGKYHAIYDLKGSPEESYKDINCYLY At2g04950 QASARTLGRLISNHFEGGKLPLRPKQLIEIFRKDHGVGINYSKAWRVQEHAAELARGLPDDSFEVLPRWFH At5g16505 QASVGWVARSVEARIR-DNPQYKPKEILQDIRDEHGVAVSYMQAWRGKERSMAALHGTYEEGYRFLPAYCE At4g08680 S---PVIARLFLDKLR-LNEKFMPMDIQEYIKERWKMVSTIPQCQRGRLLALKMLKKEYEEQFAHIRGYVE AAA21566 AVIAVMPDSVIEIDVILEDGYFSRF---FCAFGPCISGFRDGCRPYLSVDSTALNGRWNGHLASATGVDGH At1g42450 LPLETNPNSLVALDTEKYNKGVERFRYLFFALPAAVHGYAYM-RKVMVIVGTHLRGRYGGCLIAASAQDAN At2g14030 VLQEANPSSIVDLKTEIDAKGNYRFKYLFLAFAASIQGFSCM-KRVIVIGGAHLKGKYGGCLLTASAQDAN At4g04140 VLQLANPGTVYHLETELDDIGDDRFKYVFLSLGASVKRLKYI-RRVVVVDGTHLFGKYLGCLLTASCQDAN At2g04310 MLKKVNDGTITYLKL----DKNDKFQYVFVALGASIEGFRVM-RKVLIVDVTHLKNGYGGVLVFASAQDPN At2g04950 RVQVTNPGSITFFK----KDSANKFKYAFLAFGASIRGYKLM-KKVISIDGAHLISKFKGTLLGASAQDGN At5g16505 QIKLVNPGSFASVSALGPENCFQRL---FIAYRACISGFFSSCRPLLELDRAHLKGKYLGAILCAAAVDAD At4g08680 EIHSQNPGSVAFIDTYRNEKGEDVFNRFYVCFNILRTQWAGSCRPIIGLDGTFLKVVVKGVLLTAVGHDPN NP_921661 KLQRSNPGNSVHI-ALDPEEEDHVFQRFYVCFDACRRGFLEGCRRIIGLDGCFLKGPLKGELLSAIGRDAN AAA21566 NWMYPVCFGFFQAETVDNWIWFMKQLKKVV--GDMTLLAICSDAQKGLMHAVNEVFPYAERRECFRHL At1g42450 FQVFPITFGIVNSENDDAWTWFMERLTEAI--PDDPDLVFVSDRHSSIYASIRKVYPMSSHAACVIHL At2g14030 FQVYPLAFGVVDSKNDDAWEWFFRVLSTAI--PDGEILTFVSDRHSSIYTGLRKVYPKARHGACIVHL At4g04140 FQIFPIAFAVVDSETDHSWTWFMNKLSEII--KDGPDLTFVSDRNQSIFKSVGLVFPQAHHGACLVHI At2g04310 RHHYIIAFAVLDGENDASWEWFFEKLKTVV--PDTSELVFMTEKCKPHKGHTERVYRRSSRKLSRVYV At2g04950 FNLYPIAFAIVDSENDASWDWFLKCLLNII--PDENDLVFVSDRAASIASGLSGNYPLAHNGLCTFHL At5g16505 DGLFPLAIAIVDNESDENWSWFLSELRKLL--GSMPKLTILSERQSAVVEAVETHFPTAFHGFCLRYV At4g08680 NQIYPIAWAVVQSENAENWLWFVQQIKKDLNLEDGSRFVILSDRSKGLLSAVKQELPNAEHRMCVKHI NP_921661 NQLYPIAWAVVEYENKDSWNWFLGHLQKDINIPVGAAWVFITDQQKGLLSIVSTLFPFAEHRMCARHI |
|
|
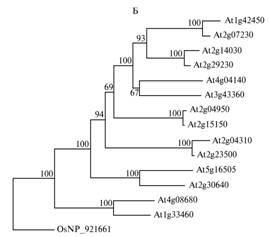
Рис. 6. Элайнмент области доменов I и II, а также междоменной вставки (281 позиция) (А, жирным шрифтом указаны аминокислоты, идентичные outgroup sequence) и эволюционное древо, описывающее взаимоотношения первичных структур транспозаз геномов Arabidopsis (Б, построено на основании элайнмен-та А; в качестве outgroup sequence использована транспозаза Orysa — NP_921661). |
Характерно, что соответствующее эволюционное древо не только демонстрирует статистически достоверные эволюционные взаимоотно-шения кластеров (рис. 6), но и обнаруживает иную топологию по срав-нению с предыдущим (см. рис. 4). Исходя из полученных данных, кластер 2g04310/2g23500 более древний, чем 2g04950/2g15150.
На основе использования в качестве outgroup sequence после-довательностей смешанного кластера Arabidopsis/Oryza может быть про-анализирован и более широкий спектр последовательностей Arabidopsis и Oryza, образующих соответствующие индивидуальные кластеры. При этом следует иметь в виду, что последние представлены сильно дивергирован-ными последовательностями, одновременный эволюционный анализ которых затруднителен.
Предложенный нами подход к выбору адекватной outgroup sequence при исследовании эволюционных взаимоотношений внутри семейства бел-ков, лишь отдаленно родственных другим семействам, может найти доста-точно широкое применение в молекулярно-эволюционных и биоинфор-мационных исследованиях, поскольку, как правило, в современных банках данных представлены многочисленные последовательности белков высоко-развитых эукариот и примитивных бактерий, тогда как промежуточные эво-люционные звенья не охарактеризованы. В качестве примера приведем эво-люционно родственные запасным белки растений купины, которые при-сутствуют в виде сильно дивергировавших последовательностей у прока-риот и эукариот (18).
Существование множества гомологичных транспозаз, а также сходство одного из структурных доменов с бактериальной транспозазой указывает на то, что эта система элементов в геноме Arabidopsis может быть очень древней.
Неожиданным оказалось то, что нами обнаружено большое число элементов, кодирующих транспозазу. Открытым остается вопрос, явля-ются ли все элементы, содержащие исследованные последовательности, автономными (способный к самостоятельной транспозиции элемент дол-жен содержать как активную транспозазу, так и участок ДНК, который может связываться с ферментом, — концевые инвертированные повторы либо консервативные мотивы). Возможно, что некоторые из рассмотрен-ных выше транспозаз дивергировали в такой степени, что утратили спо-собность к самостоятельной транспозиции (см. рис. 3). Высокое сходство последовательностей внутри консервативных доменов предполагает, что эти элементы все-таки содержат полноценную транспозазу. В случае утери необходимых для связывания фермента консервативных мотивов элемент становится дефектным, однако транспозаза может быть использована для перемещения других дефектных элементов. Предполагается, что MuDR-элемент кукурузы является предком остальных элементов этого семейства (6). Полученные результаты позволяют предположить, что рассмотренные последовательности дивергировали недавно и могут служить предше-ственниками новых дефектных элементов.
Происхождение подвижных элементов растений и содержащихся в них генов трудно обсуждать, поскольку до конца не выяснены ни их тонкая структура, ни сам механизм транспозиций (19). На основе проведенного анализа прежде всего можно поставить вопрос о сходстве механизмов транспозиций перемещающихся элементов прокариот и эукариот. Считается, что контролирующие элементы растений перемещаются во время редупликации хромосом, но, как известно, накопленные на сегодня данные свидетельствуют в пользу транспозиций контролирующих элементов с одновременной их эксцизией из участка первоначальной локализации (6, 11, 19). В частности, транспозиции, как правило, не приводят к увеличению суммарного числа копий элементов и при удвоении в одной парной хромосоме происходит их эксцизия из другой. В то же время для перемещающихся элементов бактерий характерна «репликативная» транспозиция, когда одна сестринская копия переходит в новое место, а другая сохраняет первоначальную локализацию (4).
Если вывод об «эксцизионной» транспозиции контролирующих и мобильных элементов растений подтвердится, то можно будет говорить о принципиальном отличии их от подвижных элементов прокариот, по крайней мере, по механизму перемещений, контролируемых и осуществляемых транспозазами, но по мутагенности и механизмам влияния на соседние гены сходство мобильных элементов растений и перемещающихся элементов бактерий несомненно. Это следует и из проведенных выше результатов сравнительного анализа первичных аминокислотных последовательностей транспозаз, а также мобильных элементов про- и эукариот. Аналогию между мобильными элементами, включая отдельные гены, неоднократно подчеркивали многие авторы (4, 6, 10, 20), но еще не ясно, насколько она глубока. Для ответа на этот вопрос следует ожидать дальнейшего накопления дополнительных данных.
Таким образом, сравнительный анализ первичных аминокислотных последовательностей транспозаз семейства MuDR у растений с полностью секвенированными геномами (Arabidopsis thaliana и Oryza sativa) выявил сход-ство последовательностей внутри консервативных доменов, из чего следует, что изученные мобильные генетические элементы класса II содержат полно-ценную транспозазу. При утере необходимых для связывания фермента кон-сервативных мотивов элемент становится дефектным, однако транспозаза может использоваться для перемещения других дефектных элементов. По-лученные результаты позволяют предположить, что рассмотренные последо-вательности дивергировали недавно и могут служить предшественниками но-вых дефектных элементов.
Л И Т Е Р А Т У Р А
- B o w e n N.J., J o r d a n I.K. Transposable elements and the evolution of eukaryotic complexity. Curr. Issues Mol. Biol., 2002, 4: 65-76.
- M i l l e r W.J., Mc D o n a l d J.F., P i n s k e r W. Molecular domestication of mobile elements. Genetica, 1997, 100: 261-270.
- Е в г е н ь е в М.В. Мобильные элементы и эволюция. Мол. биол., 2007, 41: 234-245.
- Х е с и н Р.Б. Непостоянство генома. М., 1985.
- V e l a z q u e z F., F r e n a n d e z S., de L o r e n z o V. The upstream-activating sequences of the sigma54 promoter Pu of Pseudomonas putida filter transcription read through from upstream genes. J. Biol. Chem., 2006, 281: 11940-11948.
- B e n n e t z e n J.L. Transposable element contributions to plant gene and genome evolution. Plant Mol. Biol., 2000, 42: 251-269.
- B e n n e t z e n J.L. Transposable elements, gene creation and genome rearrangement in flowering plants. Cur. Opin. Genet. Dev., 2005, 15: 621-627.
- V a n de P e e r Y., De W a c h t e r R. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment. Com. Appl. Biosci., 1994, 10: 569-570.
- H a r s h e y R.M., J a y a r a m M. The mu transposome through a topological lens. Crit. Rev. Biochem. Mol. Biol., 2006, 41: 387-405.
- Y u Z., W r i g h t S.I., B u r e a u T.E. Mutator-like elements in Arabidopsis thaliana. Structure, diversity and evolution. Genetics, 2000, 156: 2019-2031.
- X u Z., Y a n X., M a u r a i s S., F u H. e.a. Jittery, a mutator distant relative with a paradoxical mobile behavior: excision without reinsertion. Plant Cell, 2004, 16: 1105-1114.
- O h t s u b o F., S e k i n e Y. Bacterial insertion sequences. Curr. Top Microbiol. Im-munol., 1996, 204: 1-26.
- E f r o n B., H a l l o r a n E., H o l m e s S. Bootstrap confidence levels for phylogenetic trees. Proc. Natl. Acad. Sci. USA, 1996, 93: 13429-13434.
- B r o w n J.K. Bootstrap hypothesis tests for evolutionary trees and other dendrograms. Proc. Natl. Acad. Sci. USA, 1994. 91: 12293-12297.
- E f r o n B., G o n g G. A leisurely look at the Bootstrap, the Jack-knife, and cross-validation. The American Statistician, 1983, 37: 37-48.
- F e l s e n s t i e n J. Confidence limits on phylogenies: An approach using the bootstrap. Evolution, 1985, 39: 783-791.
- B e r l o c h e r S.H., S w o f f o r d D.L. Searching for phylogenetic trees under the frequency parsimony criterion: an approximation using generalized parsimony. Syst. Biol., 1997, 46: 211-215.
- D u n w e l l J.M. Cupins: a new superfamily of functionally diverse proteins that include germins and plant storage proteins. Biotechnol. Genet. Eng. Rev., 1998, 15: 1-32.
- J o n e s R.N. McClintock's controlling elements: the full story. Cytogenet. Genome Res., 2005, 109: 90-103.
- H e d g e s D.J, D e i n i n g e r P.L. Inviting instability: transposable elements, double-strand breaks, and the maintenance of genome integrity. Mutat. Res., 2007, 616: 46-59.
ГНУ ГНЦ РФ Всероссийский НИИ |
Поступила в редакцию |
FAMILY OF MuDR TRANSPOSASE IN GENOMES OF Arabidopsis thaliana AND Oryza sativa
Yu.V. Chesnokov, A.A. Shutova, A.D. Shutov
S u m m a r y
On the basis of comparison analysis of amino acid sequences of transposases from family of MuDR plants with entirely known genomes (Arabidopsis thaliana and Oryza sativa) the authors con-sidered the questions of the origin of mobile genetic elements in plants and the genes encoded by these elements. The existence of number homologous transposases denote that in A. thaliana genome this sys-tem may be very old. Based upon similarity of sequenses within conservative domens the conclusion was made that mobile genetic elements of class II have full transposase. In case of the loss of necessary for enzyme binding conservative motif this element becomes defective, however the transposase may be used for translocation of other defective elements. Obtained results permit to suppose that examined sequenses diverged recently and served as precursors of new defective elements.