doi: 10.15389/agrobiology.2022.6.1055rus
УДК 636.2.033:577.21:57.087
Исследования проведены при поддержке Министерства науки и высшего образования Российской Федерации. регистрационный номер темы Государственного задания № FGGN-2022-0011.
CИСТЕМА ОЦЕНКИ МЯСНОГО СКОТА ПО ПОКАЗАТЕЛЯМ ЭФФЕКТИВНОСТИ ИСПОЛЬЗОВАНИЯ КОРМА И ЭНЕРГИИ РОСТА НА ОСНОВЕ ПРИМЕНЕНИЯ ЦИФРОВЫХ И ГЕНОМНЫХ ТЕХНОЛОГИЙ (обзор)
А.А. БЕЛОУС✉, А.А. СЕРМЯГИН, Н.А. ЗИНОВЬЕВА
Мясное скотоводство характеризуется существенно более высокими затратами корма на получение единицы продукции по сравнению с другими отраслями животноводства. Для большинства видов сельскохозяйственных животных селекция на повышение эффективности использования корма до недавнего времени была затруднена из-за сложности индивидуальной оценки этого показателя. В основном улучшение признака происходило косвенно, посредством селекции на повышение интенсивности роста и уменьшение содержания жира в тушах. В 1960-1980 годах компания «Förster-Technik GmbH» (Германия) разработала автоматические кормовые станции индивидуального откорма для учета данных о затратах энергии на рост и развитие животных, что позволило вывести показатель конверсии корма (feed conversion rate, FCR), который остается одним из основных параметров эффективности использования корма (K.R. Koots с соавт., 1994). FCR как признак генетического отбора не имеет важного значения из-за умеренной наследуемости (А.А. Сермягин с соавт., 2020; Crews D.N. с соавт., 2005). В связи с этим и благодаря данным с фидлотов в 1963 году была разработана новая альтернативная концепция показателя FCR — прогнозируемое остаточное потребление корма (residual feed intake, RFI). RFI — это индивидуальная характеристика животного, которая определяется по результатам тестового откорма (продолжительность от 70 до 84 сут) с ежесуточным учетом потребленного корма и прироста живой массы (R.M. Koch с соавт., 1963). Преимущество RFI в качестве характеристики эффективности использования корма совместно с FCR заключается в том, что отбор по отрицательному значению RFI даст возможность сократить потребление корма без ущерба для роста. Прогнозируемое остаточное потребление корма не зависит от продуктивности, роста и размера тела, что делает этот признак важным для включения в селекционный отбор (G. Acetoze с соавт., 2015; J.A. Archer с соавт., 2000; G.E. Carstens с соавт., 2002). Установлено, что RFI коррелирует с FCR (коэффициенты генетической корреляции варьируют от 0,45 до 0,85), но RFI не зависит от среднесуточного прироста (ADG) и метаболической массы тела (MWT) (B.W. Kennedy с соавт., 1993; P.F. Artur с соавт., 2001). Утверждение, что особи с одной и той же массой тела требуют разное количество корма для достижения одной и той же продуктивности, составляет научную основу для оценки RFI у мясного скота. Благодаря тому, что показатель RFI наследственно обусловлен (коэффициенты наследуемости варьируют от 0,08 до 0,49), ведется направленный поиск локусов количественных признаков (quantitative trait loci, QTL) при помощи методологии GWAS (genome-wide association studies). С 2000-х годов были разработаны и внедрены способы оценки племенной ценности сельскохозяйственных животных с использованием информации по большому числу SNP (single nucleotide polymorphism), основанные на принципе линейного моделирования. Линейные модели в зависимости от подхода к структурированию данных подразделяются на rrBLUP (оценка эффекта каждого маркера), GBLUP (оценка племенной ценности на основании геномного родства) и один из наиболее распространенных методов одношаговой оценки — ssGBLUP (модель оценки геномной племенной ценности, учитывающая геномное родство наряду с родословной); нелинейные байесовские методы включают BayesA и BayesB. Научные исследования с использованием полногеномного анализа ассоциаций позволили разработать программы геномной селекции и идентифицировать ряд SNP, ассоциированных с показателями эффективности использования корма. Так, обнаружены семь позиционных генов-кандидатов, которые ранее ассоциировались с эффективностью использования корма и энергией роста у разных видов сельскохозяйственных животных, а недавно были выявлены у крупного рогатого скота породы ангус. Проведенный анализ зарубежных исследований позволяет нам рекомендовать применение описанных методов как в научно-исследовательской работе, так и в производственных целях с перспективой включения указанных параметров в критерии геномной оценки мясного скота разных пород, разводимых на территории России.
Ключевые слова: эффективность использования корма, конверсия корма, прогнозируемое остаточное потребление корма, геномные технологии, полногеномное ассоциативное исследование, GWAS, мясной скот, свиноводство.
Повышение эффективности использования корма — одна из основных целей селекции сельскохозяйственных животных, напрямую связанная с экономической эффективностью производства продукции животноводства. Особую актуальность проблема приобретает в мясном скотоводстве, характеризующимся существенно более высокими затратами корма на получение единицы продукции по сравнению с другими отраслями животноводства. Так, затраты корма на получение 1 кг прироста у мясного крупного рогатого скота (КРС) составляют в среднем 6,0 кг/кг, в то время как в свиноводстве и птицеводстве — соответственно 2,5 и 1,9 кг/кг. Несмотря на значительный прогресс в генетике и технологиях управления стадом (содержание, кормление, поддержание здоровья), в мясном скотоводстве затраты на корма составляют 60-65 % в себестоимости производства говядины, а на некоторых этапах технологического процесса могут превышать 80 % от общих затрат (1).
Хотя за последние 40 лет наблюдается существенное улучшение показателей, характеризующих эффективность использования корма, остаются возможности для дальнейшего прогресса в этом направлении (2). По оценкам, только за счет повышения эффективности использования корма на 10 % ежегодная экономия в мясном скотоводстве США может превысить 1 млрд долларов (3). Для большинства видов сельскохозяйственных животных селекция на повышение эффективности использования корма до недавнего времени была затруднена из-за сложности индивидуальной оценки этого показателя. В основном улучшение признака происходило косвенно, посредством селекции на повышение интенсивности роста и уменьшение содержания жира в тушах, однако эффективность такой селекции была относительно низкой. Проблема была решена благодаря разработке и внедрению автоматических кормовых станций, позволяющих осуществлять точный индивидуальный учет потребленного корма (4).
В настоящем обзоре выполнен анализ методических подходов, которые применяются для повышения эффективности использования корма в мясном скотоводстве.
Автоматические кормовые станции и показатели эффективности использования корма. Традиционная оценка эффективности использования корма основана на определении экономически-значимого показателя — конверсии корма (feed conversion rate, FCR), то есть отношения потребленного корма (сухого вещества) к приросту живой массы за определенный период времени. Животные с низким значением FCR потребляют меньше корма на 1 кг прироста живой массы, в то время как животные с более высоким FCR — больше. Однако при использовании показателя FCR в селекции необходимо принимать во внимание его сильную взаимосвязь с количеством потребленного корма и среднесуточными приростами у каждого животного. Для изучения образования и использования энергии в 1960-1980 годах компания «Förster-Technik GmbH» (Германия) разработала автоматические фид-лоты индивидуального откорма сельскохозяйственных животных. В настоящее время на рынке представлено несколько типов таких систем с разной степенью автоматизации, которые создавались поэтапно (5). На первом этапе (смешивание—распределение—выталкивание) стационарный кормосмеситель заполнялся из силосных бункеров благодаря встроенному мобильному оборудованию. Преимущество этого варианта в том, что группу животных можно кормить автоматически несколько раз в сутки, однако заполнение кормосмесителя по-прежнему требует значительного времени. На втором этапе были разработаны станции, в которых автоматически происходит наполнение смесителя—смешивание—распределение—(выталкивание). Такой тип станций характеризуется возможностью кормить всех животных в группах несколько раз в сутки, а фермер больше не привязан к фиксированному времени наполнения кормосмесителя и кормления животных. Третий этап предусматривает автоматизацию на этапах разгрузка и транспортировка—наполнение смесителя—смешивание—распределение—выталкивание. В системах этого типа полностью автоматическая подача пока что осуществляется только в сочетании с башенными или глубокими силосами, но такие системы следует классифицировать как сравнительно дорогие по конструкции и потребляемой мощности.
В настоящее время устоявшиеся и улучшенные технологии автоматического кормления КРС позволяют контролировать качество и потребление корма, скорость роста, кормовое поведение, эффективность использования корма, что особенно важно, поскольку развитие животных может значительно различаться. В связи с разной динамикой и временем развития организма у КРС мясного направления продуктивности необходимо проводить селекцию не только на повышение выхода мяса и улучшение его качества, но и на такие показатели, как конверсия корма и прогнозируемое остаточное потребление корма (рис.).
FCR взаимосвязана с общим потреблением энергии, со скоростью роста и живой массой (6). Генетический отбор по конверсии корма и среднесуточному приросту может быть проблематичен, поскольку обычно больше внимания уделяется признакам с большей изменчивостью (7). Генетическая корреляция в отношении FCR положительная, то есть отбор по конверсии корма должен приводить к более быстрому росту КРС мясного направления. Животные при этом будут иметь большую финальную метаболическую массу и окажутся более требовательны к условиям содержания. Отбор только по FCR, вероятно, окажется менее эффективным в долгосрочной перспективе. Следствием селекции на снижение конверсии корма может стать повышение массы при отъеме, при достижении годовалого возраста и увеличение затрат из-за возрастания потребности животных в нутриентах (2, 8, 9). Поскольку обеспечение кормами оказывает влияние на прибыльность предприятия, существенное значение приобретают способы оценки эффективности использования корма, многие из которых учитывают косвенные показатели (10, 11).
K.R. Koots с соавт. (12) сообщили об отрицательной взвешенной генетической корреляции между коэффициентом конверсии корма, скоростью роста и размером животных. Полученные значения коэффициентов корреляции указывают на то, что отбор на снижение FCR повышает эффективность использования корма и сопровождается увеличением скорости роста и ускорением наступления зрелости животного. Несмотря на то, что конверсия корма — это умеренно наследуемый признак (4), он не имеет важного значения как параметр, используемый для генетического улучшения показателей эффективности использования корма (13).
Альтернативным параметром оценки эффективности использования корма, который нашел широкое применение в отношении разных видов сельскохозяйственных животных, включая мясной скот, служит остаточное потребление корма (residual feed intake, RFI)— индивидуальная характеристика животного, которая определяется по результатам тестового откорма (продолжительность от 70 до 84 сут) с ежесуточным учетом потребленного корма и прироста живой массы. Концепция RFI была выведена в 1963 году R.M. Koch с соавт. (14, 15), и в последнее время этот параметр стал предпочтительным при измерении эффективности использования корма. Эта мера уникальна, поскольку, в отличие от FCR, разделяет потребление корма на два разных компонента: потребление корма для фактической продуктивности и прогнозируемое остаточное потребление корма. RFI — многофакторная и сложная характеристика мясного скота, вариабельность этого показателя обусловлена взаимодействием многих биологических процессов, на которые, в свою очередь, влияют физиологическое состояние и своевременное проведение ветеринарно-профилактических мероприятий.
Преимущество RFI в качестве характеристики эффективности использования корма в сочетании с FCR заключается в том, что отбор по отрицательному значению RFI даст возможность сократить потребление корма без ущерба для роста и физиологической зрелости животного (16, 17). КРС с отрицательными значениями RFI более эффективен, чем с положительными. Экономический анализ схем генетического улучшения, включающих тестирование особей по показателю RFI, показал существенные экономические выгоды по сравнению с методами, которые не включают тестирование по этому признаку. Кроме того, отбор особей по отрицательному RFI имеет дополнительное преимущество, которое заключается в снижении выбросов парниковых газов крупным рогатым скотом. Считается, что остаточное потребление корма представляет собой неотъемлемую часть основных метаболических процессов. Фактором, определяющим рентабельность животноводческих предприятий, остаются затраты на кормление. Поскольку при вычислениях величины RFI не используется показатель продуктивности животных, такой подход считается перспективным для планирования этой статьи расходов (11, 14, 16). Наряду с умеренной наследуемостью RFI не зависит от значений продуктивности, роста и размера тела, что делает его идеальным признаком для включения в селекционный отбор по эффективности использования корма (18-20).
Вклад генетической компоненты в значение RFI отмечали D.P. Berry с соавт. (11). Авторы указывают на то, что для достоверного расчета RFI требуется дополнительная информация о влиянии взаимодействия генотипа и окружающей среды на изменчивость эффективности использования корма. При математическом моделировании необходимо учитывать параметры генетической обусловленности количественных признаков (например, качества мяса) и ее проявление под воздействием среды, а также показатели, характеризующие состояния здоровья животных в анализируемых производственных условиях. В ряде работ показана генетическая обусловленность метаболической живой массы и кормового поведения (21), всасывания в кишечнике питательных веществ (22), митохондриальных функции (23) и процессов, связанных с регулированием аппетита (24). Так как на проявление откормочных качеств влияют питание, состояние здоровья и стадия вы-ращивания КРС, эффективность использования корма нельзя рассматривать в отрыве от применяемой технологической системы. RFI относится к умеренно наследуемым признакам (11), поэтому важно оценить потенциальные последствия при отборе по RFI. Кроме того, пока получено недостаточно информации о взаимосвязи между величиной RFI и показателями продуктивности животных, находящихся на пастбищах, что немаловажно при производстве говядины. Опубликованные данные о связи величины RFI у коров-матерей и их потомства в сочетании с умеренной повторяемостью и наследуемостью признака позволяют предположить, что селекция на отрицательный RFI как часть индекса множественной селекции возможна, кумулятивна и перспективна (1, 25).
В свиноводстве также были выполнены работы по изучению эффективности использования корма как полезного признака при выращивании животных и отборе в племенное стадо. H. Gilbert с соавт. (26) в обзорной статье обобщили результаты селекционного эксперимента по остаточному потреблению корма (RFI) у девяти поколений молодняка свиней крупной белой породы. После девяти поколений дивергентный отбор по прогнозируемому остаточному потреблению корма привел к очень значимым (p < 0,001) различиям по показателю RFI (-165 г/сут по сравнению с +300 г/сут) и среднесуточному потреблению корма (-270 г/сут). Отрицательные значения наблюдались по скорости роста (-12,8 г/сут, p < 0,05) и составу туши (толщина шпика +0,9 мм, p = 0,57; содержание постного мяса -2,64 %, p < 0,001) с заметным понижением коэффициента конверсии корма (-0,32 кг корма/кг прироста, p < 0,001). Пониженный предельный рН и цветовая характеристика мяса (p < 0,001) и незначительное влияние на органолептические качества мяса наблюдались у свиней с отрицательным RFI. Изменения качества мяса были связаны с нарушениями мышечного метаболизма. L. Fu с соавт. (27) провели полногеномный поиск ассоциаций (genome-wide association studies, GWAS) для показателей эффективности использования корма у свиней породы ландрас и обнаружили восемь общих областей QTL (quantitative trait loci), из которых три области, относящиеся к признакам ADFI и RFI, перекрывались. Анализ онтологии генов выявил шесть генов-кандидатов (PRELID2, GPER1, PDX1, TEX2, PLCL2, ICAM2), соответствующих признакам, связанным с эффективностью использования корма. Эти гены участвуют в процессах синтеза и распада жиров, транспорта липидов и метаболизме инсулина.
Взаимосвязь признаков эффективности использования корма. В работах, где изучались признаки эффективности использования корма, были установлены существенные индивидуальные различия в потреблении корма, которые оказались как ниже, так и выше прогнозируемых на основании живой массы и ее среднесуточного прироста. Утверждение о том, что особи с одной и той же массой тела требуют довольно разного количества корма для достижения одной и той же продуктивности, составляет научную основу для оценки RFI у мясного скота (27). Перспективы использования RFI в селекции наряду с его фенотипической независимостью от среднесуточного прироста обусловлены и тем, что признаки, которые используются для расчета этого показателя, наследственно обусловлены: коэффициенты наследуемости в разных популяциях скота варьируют от 0,08 до 0,49 (29-31).
Данные по генетической взаимосвязи признаков эффективности использования корма крупным рогатым скотом мясного направления с прогнозируемым остаточным потреблением корма, полученные зарубежными учеными, представлены в таблице 1. Остаточное потребление корма коррелирует с FCR, коэффициенты генетической корреляции варьируют от 0,45 до 0,85 (32). Это означает, что селекция на улучшение RFI приведет к непосредственному улучшению FCR (33, 36, 37). Существует положительная корреляция между RFI и потреблением корма, указывающая на то, что более эффективный (с низким RFI) КРС потребляет меньшее количество корма. Остаточное потребление корма с точки зрения фенотипа не зависит от среднесуточного прироста (ADG) и метаболической массы тела (MWT), используемых при его расчете. B.W. Kennedy с соавт. (7) утверждают, что такая связь не может проявляться и на генетическом уровне. P.F. Arthur с соавт. (6) показали, что генетическое влияние на массу при отъеме и массу при достижении годовалого возраста разделено на материнские, отцовские и индивидуальные эффекты животного. Генетическая взаимосвязь между FCR и ADG в большинстве исследований считается отрицательной и умеренной (6, 32). Особи, отобранные по снижению FCR, будут производить потомство с высокой конечной живой массой, что в итоге приведет к высоким затратам на содержание племенного стада.
В настоящее время генетическая селекция направлена на повышение эффективности использования корма и рентабельности в мясном скотоводстве. R.R. Westhuizen с соавт. (38) оценивали генетические вариации и ковариации между массой при отъеме, воспроизводственными качествами, продуктивными признаками и эффективностью использования корма. Показатели включали остаточное потребление корма (RFI), коэффициент конверсии корма (FCR), среднесуточный прирост живой массы (ADG), массу при отъеме (WW), высоту в холке (SHD), окружность мошонки (SCR) и рентабельность в период тестового откорма молодых бычков. Значимые генетические корреляции WW были установлены с SHD (rg = 0,50) и с ADG (rg = 0,28). Коэффициент наследуемости для FCR составил 0,34, для RFI — 0,31 с генетической корреляцией между признаками rg = 0,75. Предполагаемая генетическая корреляция рентабельности (rand-value) с FCR и RFI составила соответственно rg = -0,92 и rg = -0,59 (38).
Традиционные программы генетического улучшения животных включают отбор на общий прирост живой массы за период откорма в качестве основного признака и могут снизить рентабельность откорма мясного скота (39). Это происходит потому, что у бычков, отобранных только по приросту живой массы, одновременно увеличивается среднесуточное потребление корма. Следовательно, для достижения результата, желательного с точки зрения рентабельности производства, а также генетического совершенствования хозяйственно полезных признаков животных при проведении селек-ционных мероприятий необходимо учитывать комплексные показатели, характеризующие как эффективность использования корма, так откормочные качества.
Локусы количественных признаков. Интенсивное развитие молекулярно-генетических методов в первой четверти XXI века открывает новые возможности в ускорении прогресса в селекции животных, в том числе в повышении эффективности использования корма.
ДНК-технологии стали впервые использоваться в коммерческом мясном скотоводстве в 1990-х годах. Однако большинство хозяйственно полезных признаков животных имеют полигенный характер наследования, то есть определяются действием большого числа генов, получивших название локусов количественных признаков (QTL) (40). Большинство QTL оказывают лишь незначительное влияние на хозяйственно полезные свойства, и вероятность выявления их действия сильно зависит от размера выборки (41). Вместе с тем использование некоторых генов в селекции животных остается актуальным. Так, проводятся исследования по поиску и внедрению в программы селекционно-племенной работы ДНК маркеров кальпастатина (CAST) и кальпаина (CLPN), ассоциированных с нежностью мяса КРС.
В начале 2000-х годов были предложны подходы, обеспечивающие внедрение в селекцию животных методов геномной оценки (42). В последующем появились возможности для развития более совершенных процедур оценки племенной ценности животных с использованием информации по большому числу SNP (single nucleotide polymorphism), включая линейные модели rrBLUP (оценка эффекта каждого маркера), GBLUP (оценка племенной ценности по геному) и метод одношаговой оценки ssGBLUP (один из лучших на сегодняшний день; позволяет получить улучшенную геномную оценку племенной ценности с учетом данных и по геному, и по родословной), а также нелинейные байесовские методы BayesA и BayesB.
Геномная оценка впервые стала признаваться в качестве официальной для голштинской породы КРС в США с января 2009 года. С 2009 по 2013 год она была внедрена еще в 12 странах. В настоящее время геномная селекция становится традиционной практикой в молочном скотоводстве как за рубежом (43), так и в Российской Федерации (44). Использование геномной оценки в молочном скотоводстве позволило повысить точность прогноза племенной ценности молодых животных на 15-25 % и начать их более интенсивное использование, сократив тем самым интервал между поколениями и ускорив прогресс в селекции.
В мясном скотоводстве повышение селекционного ответа за счет внедрения геномной селекции оценивается в 29-58 % в зависимости от признака (45). Отсутствие фенотипической корреляции между RFI и среднесуточными приростами живой массы и наследственная обусловленность RFI сделали этот показатель предпочтительной мерой оценки при выявлении биологических механизмов эффективности использования корма для целей геномной селекции (7, 31, 46).
Для показателей потребления и эффективности использования корма было проведено точное картирование QTL с использованием 2194 маркеров на 24 аутосомах мясного скота (47). Животные в опыте происходили от быков пород ангус и шароле (20 популяций). Всего 4 QTL превысили порог достоверности полногеномных ассоциаций при p < 0,001, 3 — при p < 0,01, 17 — при p < 0,05 и 30 могли считаться суггестивными. Девятнадцать хромосом содержали QTL, значимые для показателя RFI. Результаты QTL для RFI, полученные в ряде исследований, в целом были аналогичными, пози-ции оказались сходными, но иногда различались по уровню значимости. Для FCR и DMI было обнаружено меньше QTL, чем для RFI, — соответственно 12 и 4. Некоторые хромосомы содержали QTL для FCR, но не для RFI, при этом все QTL для показателя DMI находились на хромосомах, где они были обнаружены и для RFI. Наиболее значимый QTL для RFI располагался на BTA3 (82 сМ; p = 7,60×10-5), для FCR — на BTA24 (59 сМ; p = 0,0002), а для DMI — на ВТА7 (54 сМ; p = 1,38×10-5). QTL для RFI, у которых результаты картирования были наиболее приближены к полученным в предыдущих исследованиях, находились на BTA1, BTA7, BTA18 и BTA19 (48). Обнаруженные локусы количественных признаков обеспечивают отправную точку для идентификации генов, влияющих на потребление и эффективность использования корма в маркерной селекции (49).
J.D. Nkrumah с соавт. (37) получили информацию о генотипе 20 быков-гибридов (ангус, шароле и альберта), имеющих не менее 400 потомков, по 100 микросателлитным маркерам и 355 SNP. Анализируемые признаки включали в себя показатель ADG на откормочной площадке, дневной DMI, отношение количества потребленного корма к приросту живой массы F:G (величина, обратная эффективности прироста G:F) и RFI. Предполагаемые QTL для ADG (p < 0,05) находились на BTA5 (130 сМ), BTA6 (42 сМ), BTA7 (84 сМ), BTA11 (20 сМ), BTA14 (74 сМ), BTA16 (22 сМ), BTA17 (9 сМ), BTA18 (46 сМ), BTA19 (53 сМ) и BTA28 (23 сМ). Для DMI предполагаемые QTL, для которых было превышено пороговое значение p < 0,05, были обнаружены на BTA1 (93 сМ), BTA3 (123 сМ), BTA15 (31 сМ), BTA17 (81 сМ), BTA18 (49 сМ), BTA20 (56 сМ) и BTA26 (69 сМ). E.L. Sherman с соавт. (47) выявили ассоциации SNP на BTA2, BTA5, BTA10, BTA20 и BTA29 с DMI, RFI и FCR. Это исследование показывает, что обнаруженные SNP могут влиять на иные биологические механизмы эффективности использования корма, чем контроль потребления корма и набора массы. Полученные SNP можно использовать в селекции (50).
Для размещения общедоступных данных по QTL крупного рогатого скота, кур и свиней была создана база данных Animal QTLdb (https://www.ani-malgenome.org/cgi-bin/QTLdb/SS/search). В 2007 году разработаны инструменты, позволяющие сопоставлять QTL с различными особенностями генома, такими как конечные последовательности бактериальной искусственной хромосомы, SNP и элементы массива олигонуклеотидов. В дополнение к Animal QTLdb для сопоставления QTL у разных видов используют виртуальную сравнительную карту (VCmap), которая была совместно разработана Университетом штата Айова (Iowa State University) и Медицинским колледжем Висконсина (Medical College of Wisconsin) (http://www.animalgeno-me.org/VCmap). Была усовершенствована методология извлечения и анализа данных по QTL, и за последние несколько лет достигнут значительный прогресс. В настоящее время база данных QTL расширена за счет включения еще двух видов (баран и радужная форель), что дало дополнительные возможности для сравнительного картирования. Представленные сведения постоянно уточняются новыми данными. О популярности Animal QTLdb свидетельствует число публикаций, в которых упоминается ее использование (на август 2022 года зафиксировано 1498 цитирований) (51-53). Получение более детальных сведений об изменениях количественных признаков и совершенствование терминологического аппарата генной онтологии повышают точность аннотирования QTL. Поскольку наборы генов стали доступны для анализа экспрессии с использованием микрочипов, а для изучения полногеномных ассоциаций (GWAS) были созданы массивы SNP вы-сокой плотности, QTL анализ больше не является единственным способом установления связи между генами и признаками, и вовлечение SNP в изучение полногеномных ассоциаций неизбежно. В соответствии с этой концепцией требуется метаанализ огромных объемов экспериментальных данных, которые продолжают быстро накапливаться.
Полногеномное ассоциативное исследование эффективности использования корма. Принимая во внимание экономическую важность разработки и внедрения программ геномной селекции, направленных на повышение эффективности использования корма и энергии роста, исследовательские группы в разных странах начали полногеномные ассоциативные исследования (GWAS) (54-57) с использованием ДНК-чипов средней (Bovine SNP50 BeadChip, «Illumina Inc.», США) (58, 60) и высокой (Bovine HD Bead Chip, «Illumina Inc.», США) плотности (59, 61). Результаты позволили уточнить методические аспекты разработки программ геномной селекции для мясного скота и идентифицировать ряд позиционных и функциональных SNP, ассоциированных с эффективностью использования корма и откормочными показателями.
Так, C.M. Seabury с соавт. (59) выполнили GWAS анализ для среднесуточного прироста живой массы (ADG), потребления сухого вещества (DMI), метаболической массы тела (MWT) и остаточного потребления корма (RFI), используя фактические и импутированные генотипы (Illumina 778K) для 3887 гол. трех американских популяций мясного скота (ангусы, герефорды, симангусы) и значения коэффициентов наследуемости, основанные на генотипах по SNP маркерам. По вычисленной доле аддитивно-генетической вариансы, которую можно объяснить эффектом действия маркеров (PVE≥1 %), и номинальному порогу p-значения (p ≤ 5e-05) в трех популяциях для всех изучаемых признаков были найдены QTL со средним или высоким эффектом действия. Идентичные или близко расположенные (±0,2 Mb) SNP, связанные с показателями ADG, DMI, MWT и RFI, в совокупности подтвердили потенциал плейотропных QTL или близко расположенных мутаций для нескольких признаков внутри и между анализируемыми популяциями. Основанные на маркерах коэффициенты наследуемости по всем признакам варьировали от 0,18 до 0,60 при геномном анализе с использованием чипа Illumina 778K HD и от 0,17 до 0,57 — при использовании чипа Illumina Bovine SNP50. Идентификация QTL, обнаруженных с помощью чипа Illumina Bovine SNP50, дала неоднозначные результаты. Вероятно, применения чипа низкой плотности оказалось недостаточно для обнаружения значимых QTL в исследованных популяциях и соответствующие программы селекции или отбора должны быть основаны на анализе полиморфизмов (импутированных или непосредственно детектированных) на чипах более высокой плотности.
Работы M.H. Santana с соавт. (62) были направлены на идентификацию SNP, связанных с показателями DMI и RFI у КРС породы неллор, с использованием панелей средней (Illumina® BovineSNP50 v2 BeadChip), высокой плотности (Illumina® BovineHD BeadChip) и их комбинации для поиска возможных генов-кандидатов с известной функцией. По результатам анализа авторы выявили для DMI три SNP, у которых был превышен порог значимости в тесте множественного сравнения Бонферрони, для RFI — два SNP.
С использованием чипа Illumina 778K у КРС породы ангус обнаружили семь QTL со значимым эффектом, распределенных по семи аутосо-мам. Большинство позиционных генов-кандидатов, находящихся в обнаруженном QTL или вблизи него (XIRP2, HSPB8, TOX/TRNAC-GCA, DDB1, DAK, ADPRHL1, CDC-16), ранее ассоциировались с эффективностью использования корма и энергией роста у других видов животных (бройлеры, домашняя птица, свиньи) (63, 64) и ожирением у людей (65, 66); эти гены также участвуют в возобновлении клеточного цикла у человека после контрольной точки S-фазы (67-70). Более того, один позиционный ген-кандидат (DAK) вовлечен в биосинтез рибофлавин-4´,5´-фосфата (65), который действует как акцептор электронов в окислительном метаболизме углеводов, аминокислот и жирных кислот и может отдавать электроны в электрон-транспортную цепь (69, 70). Рибофлавин необходим для производства энергии, которая требуется для роста и развития организма (65, 66, 69, 70).
У КРС породы герефорд полногеномный анализ RFI с использованием ДНК чипа Illumina 778K показал наличие четырех QTL с высоким эффектом, распределенных по четырем аутосомам (6_113 Mb, 19_54 Mb, 3_29 Mb, 1_72 Mb) (табл. 2). Оценка позиционных генов-кандидатов (STC2, RAB28, DLG1) выявила ассоциации с ожирением человека, адипогенной дифференцировкой, диабетом I типа и ревматоидным артритом, а также подавлением роста и развития костей и мышц у мышей. Ген NCAPG, связанный с развитием массы тела у КРС, ранее был предложен K. Setoguchi с соавт. (74) для молекулярных тест-систем в качестве кандидата.
F. Zhang с соавт. (75) показали 12 из 16 значимых SNP для RFI, при этом 3 гена оказались расположены внутри SNP (табл. 3). Полногеномные SNP для показателя DMI были расположены на 11 хромосомах, и в SNP rs207689046 был выявлен ген LCORL. Также было обнаружено, что полногеномные SNP на нескольких хромосомах связаны с признаками ADG и MWT. Из 12 SNP, которые объясняли более 0,30 дисперсии фенотипа для среднесуточного прироста, три SNP находились вблизи гена или были сцеплены с ним: rs110987922 расположен на расстоянии 121223 п.н. от гена LCORL, а rs134215421 — на 1166 п.н. и сцеплен с геном PLAG1. По показателю метаболической массы тела 6 из 10 генов были расположены внутри SNP, а один ген был идентифицирован в близлежащем регионе. На SNP rs39111019 (BTA6, 118907 п.н., сцеплен с геном LCORL) приходилось 5,80 % фенотипической дисперсии.
В целом результаты GWAS анализа показывают, что подавляющее большинство выявленных закономерностей носят популяционно-зависимый характер. Вследствие генетических (linkage disequilibrium, неравновесное сцепление генов) и фенотипических различий возникает необходимость проведения GWAS для каждой породы или популяции скота, для которой необходимо разработать программу геномной селекции.
Таким образом, представленный обзор публикаций по эффективности использования корма, включая его конверсию, свидетельствует об актуальности рассматриваемой тематики в странах — лидерах мясного животноводства. При оценке этого показателя необходимо учитывать комплекс факторов — технологических, биологических и экономических, поэтому применяемый (в том числе в практике животноводства) математический аппарат усложняется. Очевидно, что в России проблеме эффективности использования корма должно уделяться необходимое внимание. В перспективе описанные в настоящем обзоре фенотипические и генотипические параметры могут быть включены в набор критериев для геномной оценки мясного скота пород, разводимых в России.
1. Генетическая взаимосвязь (коэффициенты корреляции) откормочных показателей с RFI у бычков различных пород (r±SE) |
|||||||
Признак |
Порода |
||||||
ангус (6) |
шароле (19) |
герефорд (17) |
помесь1 (32) |
помесь2 (33) |
помесь3 (37) |
||
Число животных |
1180 |
792 |
540 |
2284 |
1481 |
740 |
464 |
ADG, г |
-0,04±0,08 |
-0,10±0,13 |
0,09±0,29 |
0,01 |
0,09±0,20 |
0,25±0,16 |
0,46±0,45 |
MWT, кг |
-0,06±0,08 |
|
0,22±0,29 |
-0,17 |
-0,20±0,16 |
0,16±0,13 |
0,27±0,33 |
FCR, кг/кг |
0,66±0,05 |
0,85±0,05 |
0,70±0,22 |
0,69 |
0,41±0,32 |
0,64±0,10 |
0,62±0,09 |
BW1, кг |
-0,45±0,22 |
|
0,34±0,34 |
|
|
|
|
BW0, кг |
-0,26±0,14 |
0,32±0,10 |
0,15±0,28 |
0,81 |
0,43±0,15 |
0,19±0,15 |
|
Feed intake, г |
0,66±0,05 |
0,79±0,04 |
0,64±0,16 |
|
|
0,78±0,06 |
0,73±0,18 |
Примечание. Помесь1 — смесь пород шароле, лимузин, симментал, герефорд и ангус, помесь2 — брахманский скот и санта-гертрудис, помесь3 — гибрид альберта, ангус, шароле; ADG — среднесуточный прирост, MWT — метаболическая масса тела, FCR — конверсия корма, BW1 — начальная живая масса, BW0 — конечная живая масса, Feed intake — потребление корма. |
|||||||
2. Характеристика основных SNP (single nucleotide polymorphism) каждой хромосомы в значимых ассоциациях с RFI (residual feed intake) в популяциях крупного рогатого скота мясного направления продуктивности |
|||||
Порода |
Позиция (Chr_Mb) |
-log10 |
Ген-кандидат |
Функции у разных видов |
Ссылка |
Ангус |
2_30 |
5,51 |
XIRP2 |
Эффективность кормления и рост (крупный рогатый скот) |
(63) |
17_58 |
4,77 |
HSPB8 |
Признаки ожирения (человек) |
(64) |
|
14_27 |
4,56 |
TOX/TRNAC-GCA |
Признаки ожирения (человек) |
(65) |
|
29_40 |
4,55 |
DDB1/DAK |
Клеточный цикл, эффективность питания и рост (человек, курица, утка, свинья) |
||
12_91 |
4,39 |
ADPRHL1/CDC-16 |
Эффективность кормления и рост (курица, свинья) |
||
Герефорд |
6_113 |
6,51 |
RAB28 |
Признаки ожирения (человек) |
(73) |
20_49 |
3,65 |
STC2 |
Роль в ожирении (человек), подавление роста и развитие костей, мышц (мышь) |
(75) |
|
6_47 |
1,70 |
NCAPG |
Связан с тушей и массой тела (крупный рогатый скот) |
(74) |
|
1_72 |
4,34 |
DLG1 |
Поглощение глюкозы (человек) |
(73) |
|
3. Основные SNP каждой хромосомы в значимых ассоциациях с RFI, DMI, ADG и MWT в многопородной популяции крупного рогатого скота мясного направления продуктивности |
|||||
Признак |
BTA |
Позиция, п.н. |
SNP |
p-значение |
Ген-кандидат |
RFI |
1 |
121 176 492 |
rs109479784 |
8,27E-06 |
SNORA70 |
2 |
28 511 594 |
rs379241952 |
9,69E-07 |
B3GALT1 |
|
3 |
6 835 555 |
rs110523019 |
1,74E-07 |
DDR2 |
|
4 |
89 834 757 |
rs42645457 |
6,12E-06 |
GPR37 |
|
5 |
9 075 556 |
rs446215391 |
6,77E-07 |
SYT1 |
|
12 |
54 262 083 |
rs382972340 |
8,21E-06 |
U6 |
|
13 |
35 856 785 |
rs382536070 |
6,60E-06 |
LYZL1 |
|
16 |
13 105 979 |
Chr16:13105979 |
8,38E-06 |
RGS2 |
|
23 |
48 775 591 |
rs382491772 |
8,90E-06 |
F13A1 |
|
DMI |
1 |
25 084 372 |
rs211318336 |
8,30E-06 |
U2 |
2 |
112 157 337 |
rs109570141 |
1,96E-06 |
U6atac |
|
4 |
3 153 240 |
rs472695088 |
2,80E-06 |
SNORA31 |
|
6 |
39 105 359 |
rs207689046 |
2,77E-25 |
LCORL |
|
10 |
31 282 009 |
rs109256612 |
7,06E-06 |
DPH6 |
|
12 |
54 262 083 |
rs382972340 |
1,03E-06 |
U6 |
|
13 |
19 004 111 |
rs384869645 |
5,56E-06 |
PARD3 |
|
14 |
24 973 953 |
rs110092040 |
1,12E-08 |
MOS |
|
16 |
78 179 941 |
rs380573663 |
3,66E-06 |
CRB1 |
|
20 |
4 791 751 |
rs43357086 |
4,33E-09 |
5S_rRNA |
|
22 |
30 879 104 |
rs211404023 |
3,71E-06 |
5S_rRNA |
|
ADG |
4 |
112 725 016 |
Chr4:112725016 |
3,79E-06 |
CUL1 |
5 |
106 247 266 |
rs137822220 |
8,25E-07 |
CCND2 |
|
6 |
39 113 335 |
rs110987922 |
3,28E-07 |
LCORL |
|
7 |
93 244 933 |
rs109901274 |
8,44E-08 |
ARRDC3 |
|
14 |
25 006 125 |
rs134215421 |
4,82E-13 |
PLAG1 |
|
20 |
4 916 731 |
rs42661323 |
3,65E-09 |
STC2 |
|
24 |
15 100 338 |
rs111029508 |
5,47E-06 |
snoU54 |
|
25 |
40 587 255 |
rs448890458 |
5,.31E-06 |
CARD11 |
|
28 |
45 058 986 |
rs469759962 |
9,68E-06 |
TMEM72 |
|
29 |
41 512 334 |
rs137389740 |
3,74E-06 |
SCGB1A1 |
|
MWT |
1 |
118 345 325 |
rs210255011 |
3,85E-06 |
ERICH6 |
5 |
106 266 665 |
rs110358394 |
3,20E-07 |
CCND2 |
|
6 |
39 111 019 |
Chr6:39111019 |
1,59E-04 |
LCORL |
|
7 |
93 244 933 |
rs109901274 |
9,61E-09 |
ARRDC3 |
|
11 |
68 821 419 |
rs446606774 |
7,92E-07 |
GALNT14 |
|
14 |
25 006 125 |
rs134215421 |
2,08E-28 |
PLAG1 |
|
20 |
4 563 925 |
rs41934045 |
6,12E-21 |
ERGIC1 |
|
21 |
21 679 784 |
rs209660822 |
8,25E-06 |
AP3S2 |
|
26 |
8 545 128 |
rs133223744 |
3,27E-06 |
A1CF |
|
Примечание. DMI — потребление сухого вещества, RFI — прогнозируемое остаточное потребление корма, ADG — среднесуточный прирост, MWT — метаболическая масса тела. Составлено на основании данных, приведенных в статье F. Zhang с соавт. (75). |
|||||
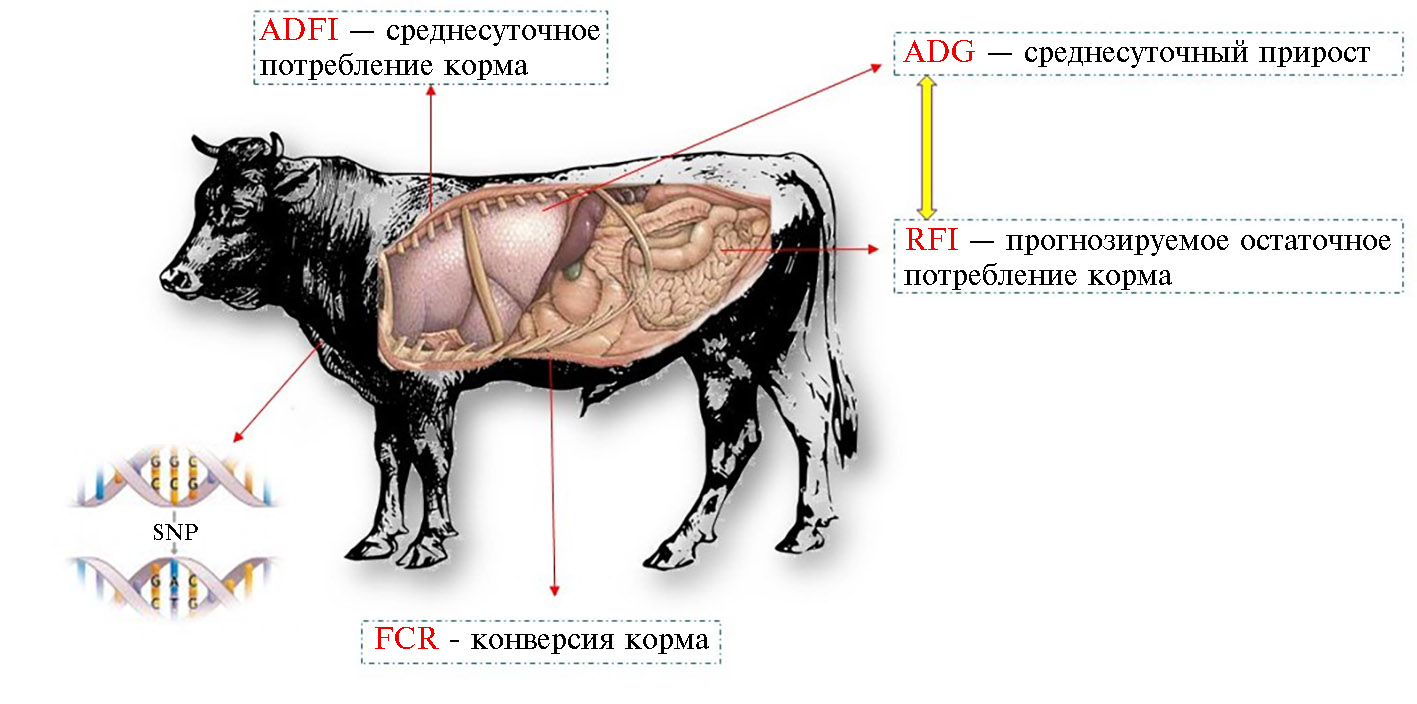
Показатели эффективности использования корма (ADFI, ADG, RFI, FCR) как селекционно значимые признаки при улучшении продуктивных качеств крупного рогатого скота мясного направления. На основе фенотипического описания по ADFI, ADG, RFI, FCR и поиска SNP (single nucleotide polymorphism) осуществляется поиск ассоциаций геномных вариантов с этими признаками для их вовлечения в селекционные программы.
ЛИТЕРАТУРА
- Lowe M., Gereffi G. A value chain analysis of the U.S. beef and dairy industries. Center on Globalization, Governance & Competitiveness, Duke University, Duke, NC, USA, 2009 (doi: 10.13140/RG.2.1.1502.9523).
- Archer J.A., Richardson E.C., Herd R.M., Arthur P.F. Potential for selection to improve efficiency of feed use in beef cattle: a review. Australian Journal of Agricultural Research, 1999, 50(2): 147-162 (doi: 10.1071/A98075).
- Taylor J., Kerley M., Schnabel R., Pomp D., Garrick D., Hansen S., Loy D., Tait J. R., Weaber R., Seabury C., Beever J., Faulkner D., Shike D., Fahrenkrug S., Spangler M., Sonstegard T., Freetly H., Pollak J., Johnson K., Neibergs H. National program for the genetic improvement of feed efficiency in beef cattle. Faculty Papers and Publications in Animal Science, 2016: 907.
- Сермягин А.А., Белоус А.А., Требунских Е.А., Зиновьева Н.А. Показатели кормового поведения как новые селекционные признаки в разведении свиней. Сельскохозяйственная биология, 2020, 55(6): 1126-1138 (doi: 10.15389/agrobiology.2020.6.1126rus).
- Oberschätzl-Kopp R., Peis R., Haidn B., Reiter K. Studies on dairy cow behaviour with automatic feeding in a herd milked by an AMS. LandTechnik, 71(2), 2016: 55-65 (doi: 10.15150/lt.2016.3122).
- Arthur P.F., Archer J.A., Johnston D.J., Herd R.M., Richardson E.C., Parnell P.F. Genetic and phenotypic variance and covariance components for feed intake, feed efficiency and other postweaning traits in Angus cattle. Journal of Animal Science, 2001, 79(11): 2805-2811 (doi: 10.2527/2001.79112805x).
- Kennedy B.W., van der Werf J.H., Meuwissen T.H. Genetic and statistical properties of residual feed intake. Journal of Animal Science, 1993, 71(12): 3239-3250 (doi: 10.2527/1993.71123239x).
- Deutscher G.H. Reducing calving difficulty by heifer and sire selection and management. Proc. The Range Beef Cow Symposium XIV. Gering, Nebraska, 1995: 183.
- Strohbehn D., Bryce S., Maxwell D., Anderson L., Wilson D. Influence of Angus sire birth weight EPD on performance of crossbred first-calf heifers: a progress report. In: Iowa State Beef Research Report. Iowa, 1993: 54-60.
- Nielsen M.K., MacNeil M.D., Dekkers J.C.M., Crews D.H., Rathje T.A., Enns R.M., Weaber R.L. Review: Life-cycle, total-industry genetic improvement of feed efficiency in beef cattle: Blueprint for the Beef Improvement Federation. Professional Animal Scientist, 2013, 29: 559-565 (doi: 10.15232/S1080-7446(15)30285-0).
- Berry D.P., Crowley J.J. Cell biology symposium: genetics of feed efficiency in dairy and beef cattle. Journal of Animal Science, 2013, 91(4): 1594-1613 (doi: 10.2527/jas.2012-5862).
- Koots K.R., Gibson J.P., Wilton J.W. Analyses of published genetic parameter estimates for beef production traits. 2. Phenotypic and genetic correlations. Animal Breeding Abstracts, 1994, 62(11): 825-853.
- Crews D.H.Jr. Genetics of efficient feed utilization and national cattle evaluation: a review. Genet. Mol. Res., 2005, 4(2): 152-165.
- Koch R.M., Swiger L.A., Chambers D., Gregory K.E. Efficiency of feed use in beef cattle. Journal of Animal Science, 1963, 22(2): 486-494 (doi: 10.2527/jas1963.222486x).
- Savietto D. Berry D.P. Friggens N.C. Towards an improved estimation of the biological components of residual feed intake in growing cattle. Journal of Animal Science, 2014, 92(2): 467-476 (doi: 10.2527/jas.2013-6894).
- Archer J.A., Arthur P.F., Herd R.M., Richardson E.C. Genetic variation in feed efficiency and its component traits. Proc. 6th World Congress on Genetics Applied to Livestock Production. Armidale, NSW, Australia, 25: 81-84.
- Herd R.M., Bishop S.C. Genetic variation in residual feed intake and its association with other production traits in British Hereford cattle. Livestock Production Science, 2000, 63(2): 111-119 (doi: 10.1016/S0301-6226(99)00122-0).
- Acetoze G., Weber K.L., Ramsey J.J., Rossow H.A. Relationship between liver mitochondrial respiration and proton leak in low and high RFI steers from two lineages of RFI Angus bulls. International Scholarly Research Notices, 2015: 194014 (doi: 10.1155/2015/194014).
- Archer J.A., Bergh L. Duration of performance tests for growth rate, feed intake and feed efficiency in four biological types of beef cattle. Livestock Production Science, 2000, 65(1-2): 47-55 (doi: 10.1016/S0301-6226(99)00181-5).
- Carstens G.E., Theis C.M., White M.B., Welsh T.H., Warrington B.G., Randel R.D., Forbes T.D.A., Lippke H., Greene L.W., Lunt D.K. Residual feed intake in beef steers: I. Correlation with performance traits and ultrasound measures of body composition. Proc. Western Section, American Society of Animal Science, 2002, 53: 552-555.
- Herd R.M., Arthur P.F. Physiological basis for variation in residual feed intake. Journal of Animal Science, 2009, 87(suppl_14): E64-E71 (doi: 10.2527/jas.2008-1345).
- Montanholi Y., Fontoura A., Swanson K., Coomber B., Yamashiro S., Miller S. Small intestine histomorphometry of beef cattle with divergent feed efficiency. Acta Veterinaria Scandinavica, 2013, 55(1): 9 (doi: 10.1186/1751-0147-55-9).
- Lancaster P.A., Carstens G.E., Michal J.J., Brennan K.M., Johnson K.A., Davis M.E. Relationships between residual feed intake and hepatic mitochondrial function in growing beef cattle. Journal of Animal Science, 2014, 92(7): 3134-3141 (doi: 10.2527/jas.2013-7409).
- Perkins S.D., Key C.N., Garrett C.F., Foradori C.D., Bratcher C.L., Kriese-Anderson L.A., Brandebourg T.D. Residual feed intake studies in Angus-sired cattle reveal a potential role for hypothalamic gene expression in regulating feed efficiency. Journal of Animal Science, 2014, 92(2): 549-560 (doi: 10.2527/jas.2013-7019).
- Basarab J.A., McCartney D., Okine E.K., Baron V.S. Relationships between progeny residual feed intake and dam productivity traits. Canadian Journal of Animal Science, 2007, 87(4): 489-502 (doi: 10.4141/CJAS07026).
- Gilbert H., Billon Y., Brossard L., Faure J., Gatellier P., Gondret F., Labussière E., Lebret B., Lefaucheur L., Le Floch N., Louveau I., Merlot E., Meunier-Salaün M.-C., Montagne L., Mormede P., Renaudeau D., Riquet J., Rogel-Gaillard C., van Milgen J., Vincent A., Noblet J. Review: divergent selection for residual feed intake in the growing pig. Animal, 2017, 11(9): 1427-1439 (doi: 10.1017/S175173111600286X).
- Fu L., Jiang Y., Wang C., Mei M., Zhou Z., Jiang Y., Song H., Ding X.A Genome-wide association study on feed efficiency related traits in landrace pigs. Front. Genet., 2020, 11: 692 (doi: 10.3389/fgene.2020.00692).
- Liu M.F., Goonewardene L.A., Bailey D.R.C., Basarab J.A., Kemp R.A., Arthur P.F., Okine E.K., Makarechian M. A study on the variation of feed efficiency in station tested beef bulls. Canadian Journal of Animal Science, 2000, 80(3): 435-441 (doi: 10.4141/A99-030).
- Crowley J.J., McGee M., Kenny D.A., Crews D.H., Evans R.D., Berry D.P. Phenotypic and genetic parameters for different measures of feed efficiency in different breeds of Irish performance tested beef bulls. Journal of Animal Science, 2010, 88(3): 885-894 (doi: 10.2527/jas.2009-1852).
- Lu D., Miller S., Sargolzaei M., Kelly M., Vander Voort G., Caldwell T., Wang Z., Plastow G., Moore S. Genome-wide association analyses for growth and feed efficiency traits in beef cattle. Journal of Animal Science, 2013, 91(8): 3612-3633 (doi: 10.2527/jas.2012-5716).
- Saatchi M., Beever J.E., Decker J.E., Faulkner D.B., Freetly H.C., Hansen S.L., Helen Y., Kristen A.J., Stephen D.K., Monty S.K., JaeWoo K., Daniel D.L., Elisa M., Holly L.N., Pollak E.J., Shnabel R.D., Seabury C.M., Shike D.W., Snelling W.M., Spangler M.L., Weaber R.L., Garrick D.J., Taylor J.F. QTLs associated with dry matter intake, metabolic mid-test weight, growth and feed efficiency have little overlap across 4 beef cattle studies. BMC Genomics, 2014, 15: 1004 (doi: 10.1186/1471-2164-15-1004).
- Schenkel F.S., Miller S.P., Wilton J.W. Genetic parameters and breed differences for feed efficiency, growth, and body composition traits of young beef bulls. Canadian Journal of Animal Science, 2004, 84(2): 177-185 (doi: 10.4141/A03-085).
- Robinsonab D.L., Oddy V.H. Genetic parameters for feed efficiency, fatness, muscle area and feeding behaviour of feedlot finished beef cattle. Livestock Production Science, 2004, 90(2-3): 255-270 (doi: 10.1016/j.livprodsci.2004.06.011).
- Hoque M.A., Arthur P.F., Hiramoto K., Oikawa T. Genetic parameters for carcass traits of field progeny and their relationships with feed efficiency traits of their sire population for Japanese Black (Wagyu) cattle. Livestock Science, 2006, 100(2-3): 251-260 (doi: 10.1016/j.livprodsci.2005.09.006).
- Hoque M.A., Arthur P.F., Hiramoto K., Oikawa T. Genetic relationship between different measures of feed efficiency and its component traits in Japanese Black (Wagyu) bulls. Livestock Science, 2006, 99(2-3): 111-118 (doi: 10.1016/j.livprodsci.2005.06.004).
- Baker S.D., Szasz J.I., Klein T.A., Kuber P.S., Hunt C.W., Glaze J.B., Falk D., Richard J., Miller C., Battaglia R.A., Hill R.A. Residual feed intake of purebred Angus steers: Effects on meat quality and palatability. Journal of Animal Science, 2006, 84(4): 938-945 (doi: 10.2527/2006.844938x).
- Nkrumah J.D., Sherman E.L., Li C., Marques E., Crews Jr. D.H., Bartusiak R., Brenda M.M., Wang Z., Basarab J.A., Moore S.S. Primary genome scan to identify putative quantitative trait loci for feedlot growth rate, feed intake and feed efficiency of beef cattle. Journal of Animal Science, 2007, 85(12): 3170-3181 (doi: 10.2527/jas.2007-0234).
- Van Der Westhuizen R.R., Westhuizen J., Schoeman S.J. Genetic variance components for residual feed intake and feed conversion ratio and their correlations with other production traits in beef bulls. South African Journal of Animal Science, 2004, 34(4): 257-264.
- Almeida R. Consumo e eficiência alimentar de bovinos em crescimento Tese (Doutorado). Piracicaba-SP, Piracicaba, 2005: 181 (doi: 10.11606/T.11.2005.tde-09112005-150314).
- Geldermann H. Investigations on inheritance of quantitative characters in animals by gene markers. I. Methods. Theoretical and Applied Genetics, 1975, 46: 319-330 (doi: 10.1007/BF00281673).
- Hayes B., Goddard M.E. The distribution of the effects of genes affecting quantitative traits in livestock. Genet. Sel. Evol., 2008, 33: 209-229 (doi: 10.1186/1297-9686-33-3-209).
- Meuwissen T.H.E., Hayes B.J., Goddard M.E. Prediction of total genetic value using genome-wide dense marker maps. Genetics, 2001, 157(4): 1819-1829 (doi: 10.1093/genetics/157.4.1819).
- Goddard M.E., Hayes B.J. Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nature Reviews Genetics, 2009, 10: 381-391 (doi: 10.1038/nrg2575).
- Sermyagin A.A., Gladyr’ E.A., Kharitonov S.N., Ermilov A.N., Strekozov N.I., Brem G., Zinovieva N.A. Genome-wide association study for milk production and reproduction traits in Russian holstein cattle population. Agricultural Biology, 2016, 51(2): 182-193 (doi: 10.15389/agrobiology.2016.2.182eng).
- Van Eenennaam A.L., van der Werf J.H.J., Goddard M.E. The value of using DNA markers for beef bull selection in the seedstock sector. Journal of Animal Science, 2011, 89(2): 307-320 (doi: 10.2527/jas.2010-3223).
- Do D.N., Strathe A.B., Ostersen T., Pant S.D., Kadarmideen H.N. Genome-wide association and pathway analysis of feed efficiency in pigs reveal candidate genes and pathways for residual feed intake. Front. Genet., 2014, 5: 307 (doi: 10.3389/fgene.2014.00307).
- Sherman E.L., Nkrumah J.D., Li C., Bartusiak R., Murdoch B., Moore S.S. Fine mapping quantitative trait loci for feed intake and feed efficiency in beef cattle. Journal of Animal Science, 2009, 87(1): 37-45 (doi: 10.2527/jas.2008-0876).
- Chander D., Sharma V.K., Dudi K., Baban B.N., Sharma S.Ph., Negesse T., Kundu S.S., Dutta M.M., Gupta R., Singh D. Residual feed intake as a tool for selecting more efficient animals: a review. Indian Journal of Animal Nutrition, 2017, 34(3): 238-255 (doi: 10.5958/2231-6744.2017.00041.X).
- Higgins M.G., Fitzsimons C., McClure M.C., McKenna C., Conroy S., Kenny D.A., McGee M., Waters S.M., Morris D.W. GWAS and eQTL analysis identifies a SNP associated with both residual feed intake and GFRA2 expression in beef cattle. Sci. Rep., 2018, 8: 14301 (doi: 10.1038/s41598-018-32374-6).
- Moore S.S., Mujibi F.D., Sherman E.L. Molecular basis for residual feed intake in beef. Journal of Animal Science,2009, 87(suppl_14) (doi: 10.2527/jas.2008-1418).
- Hu Z.-L., Park C.A., Reecy J.M. Bringing the animal QTLdb and CorrDB into the future: meeting new challenges and providing updated services. Nucleic Acids Research, 2022, 50(D1): D956-D961 (doi: 10.1093/nar/gkab1116).
- Hu Z.-L., Park C.A., Reecy J.M. Building a livestock genetic and genomic information knowledgebase through integrative developments of Animal QTLdb and CorrDB. Nucleic Acids Research, 2019, 47(D1): D701-D710 (doi: 10.1093/nar/gky1084).
- Hu Z.-L., Park C.A., Reecy J.M. Development of animal QTLdb and CorrDB: resynthesizing big data to improve meta-analysis of genetic and genomic information. The 11th World Congress on Genetics Applied to Livestock Production (WCGALP). New Zealand, 2018: 954.
- Serão N.V., González-Peña D., Beever J.E., Faulkner D.B., Southey B.R., Rodriguez-Zas S.L. Single nucleotide polymorphisms and haplotypes associated with feed efficiency in beef cattle. BMC Genet., 2013, 14: 94 (doi: 10.1186/1471-2156-14-94).
- Snelling W.M., Allan M.F., Keele J.W., Kuehn L.A., McDaneld T., Smith T.P.L., Sonstegard T.S., Thallman R.M., Bennett G.L. Genome-wide association study of growth in crossbred beef cattle. Journal of Animal Science, 2010, 88(3): 837-848 (doi: 10.2527/jas.2009-2257).
- Bolormaa S., Pryce J.E., Kemper K., Savin K., Hayes B.J., Barendse W., Zhang Y., Reich C.M., Mason B.A., Bunch R.J., Harrison B.E., Reverter A., Herd R.M., Tier B., Graser H.U., Goddard M.E. Accuracy of prediction of genomic breeding values for residual feed intake and carcass and meat quality traits in Bos taurus, Bos indicus, and composite beef cattle. Journal of Animal Science, 2013, 91(7): 3088-3104 (doi: 10.2527/jas.2012-5827).
- Barendse W., Reverter A., Bunch R.J., Harrison B.E., Barris W., Thomas M.B. A validated whole-genome association study of efficient food conversion in cattle. Genetics, 2007, 176(3): 1893-1905 (doi: 10.1534/genetics.107.072637).
- Sherman E.L., Nkrumah J.D., Moore S.S. Whole genome single nucleotide polymorphism associations with feed intake and feed efficiency in beef cattle. Journal of Animal Science, 2010, 88(1): 16-22 (doi: 10.2527/jas.2008-1759).
- Seabury C.M., Oldeschulte D.L., Saatchi M., Beever J.E., Decker J.E., Halley Y.A., Bhattarai E.K., Molaei M., Freetly H.C., Hansen S.L., Yampara-Iquise H., Johnson K.A., Kerley M.S., Kim J., Loy D.D., Marques E., Neibergs H.L., Schnabel R.D., Shike D.W., Spangler M.L., Weaber R.L., Garrick D.J., Taylor J.F. Genome-wide association study for feed efficiency and growth traits in U.S. beef cattle. BMC Genomics, 2017, 18: 386 (doi: 10.1186/s12864-017-3754-y).
- Matukumalli L.K., Lawley C.T., Schnabel R.D., Taylor J.F., Allan M.F., Heaton M.P., O'Connell J., Moore S.S., Smith T.P.L., Sonstegard T.S., Van Tassell C.P. Development and characterization of a high-density SNP genotyping assay for cattle. PLoS ONE, 2009, 4(4): e5350 (doi: 10.1371/journal.pone.0005350).
- Rincon G., Weber K.L., Van Eenennaam A.L., Golden B.L., Medrano J.F. Hot topic: performance of bovine high-density genotyping platforms in Holsteins and Jerseys. J. Dairy Sci., 2011, 94(12): 6116-6121 (doi: 10.3168/jds.2011-4764).
- Santana M.H.A., Utsunomiya Y.T., Neves H.H., Gomes R.C., Garcia J.F., Fukumasu H., Silva S.L., Junior G.A.O., Alexandre P.A., Leme P.R., Brassaloti R.A., Coutinho L.L., Lopes T.G., Meirelles F.V., Eler J.P., Ferraz J.B.S. Genome-wide association analysis of feed intake and residual feed intake in Nellore cattle. BMC Genet., 2014, 15: 21 (doi: 10.1186/1471-2156-15-21).
- Connor E.E., Kahl S., Elsasser T.H., Parker J.S., Li R.W., Van Tassell C.P., Baldwin VI R.L., Barao S.M. Enhanced mitochondrial complex gene function and reduced liver size may mediate improved feed efficiency of beef cattle during compensatory growth. Functional & Integrative Genomics, 2010, 10: 39-51 (doi: 10.1007/s10142-009-0138-7).
- Ribeiro R., Monteiro C., Catalán V., Hu P., Cunha V., Rodríguez A., Gomez-Ambrosi J., Fraga A., Principe P., Lobato C., Lobo F., Morais A., Silva V., Sanches-Magalhaes J., Oliveira J., Pina F., Lopes C., Medeiros R., Frühbeck G. Obesity and prostate cancer: gene expression signature of human periprostatic adipose tissue. BMC Med., 2012, 10: 108 (doi: 10.1186/1741-7015-10-108).
- Cabezas A., Costas M.J., Pinto R.M., Couto A., Cameselle J.C. Identification of human and rat FAD-AMP lyase (cyclic FMN forming) as ATP-dependent dihydroxyacetone kinases. Biochemical and Biophysical Research Communications, 2005, 338(4): 1682-1689 (doi: 10.1016/j.bbrc.2005.10.142).
- Tang J., Wen Z.G., Guo Z.B., Huang W., Guo Y.M., Xie M., Hou S.S. Dietary riboflavin supplementation improve the growth performance and antioxidant status of starter white Pekin ducks fed a corn-soybean meal diet. Livestock Science, 2014, 170: 131-136 (doi: 10.1016/j.livsci.2014.10.016).
- Kong B.-W., Song J.J., Lee J.Y., Hargis B.M., Wing T., Lassiter K. Gene expression in breast muscle associated with feed efficiency in a single male broiler line using a chicken 44K oligo microarray. I. Top differentially expressed genes. Poultry Science, 2011, 90(11): 2535-2547 (doi: 10.3382/ps.2011-01435).
- Fontanesi L., Galimberti G., Calò D.G., Fronza R., Martelli P.L., Scotti E., Colombo M., Schiavo G., Casadio R., Buttazzoni L., Russo V. Identification and association analysis of several hundred single nucleotide polymorphisms within candidate genes for back fat thickness in Italian Large White pigs using a selective genotyping approach. Journal of Animal Science, 2012, 90(8): 2450-2464 (doi: 10.2527/jas.2011-4797).
- Powers H.J., Corfe B.M., Nakano E. Riboflavin in development and cell fate. In: Water soluble vitamins. Subcellular Biochemistry, vol. 56 /O. Stanger (ed.). Springer, Dordrecht, 2012: 229-245 (doi: 10.1007/978-94-007-2199-9_12).
- Henriques B.J., Olsen R.K., Bross P., Gomes C.M. Emerging roles for riboflavin in functional rescue of mitochondrial β-oxidation flavoenzymes. Curr. Med. Chem., 2010, 17(32): 3842-3854 (doi: 10.2174/092986710793205462).
- Leung-Pineda V., Huh J., Piwnica-Worms H. DDB1 targets Chk1 to the Cul4 E3 Ligase complex in normal cycling cells and in cells experiencing replication stress. Cancer Research, 2009, 69(6): 2630-2637 (doi: 10.1158/0008-5472.CAN-08-3382).
- de las Heras-Saldana S., Clark S.A., Duijvesteijn N., Gondro C., van der Werf J.H.J., Chen Y. Combining information from genome-wide association and multi-tissue gene expression studies to elucidate factors underlying genetic variation for residual feed intake in Australian Angus cattle. BMC Genomics, 2019, 20(939) (doi: 10.1186/s12864-019-6270-4).
- Comuzzie A.G., Cole S.A., Laston S.L., Voruganti V.S., Haack K., Gibbs R.A., Butte N.F. Novel genetic loci identified for the pathophysiology of childhood obesity in the Hispanic population. PLoS ONE, 2012, 7(12): e51954 (doi: 10.1371/journal.pone.0051954).
- Setoguchi K., Furuta M., Hirano T., Nagao T., Watanabe T., Sugimoto Y., Takasuga A. Cross-breed comparisons identified a critical 591-kb region for bovine carcass weight QTL (CW-2) on chromosome 6 and the Ile-442-met substitution in NCAPG as a positional candidate. BMC Genet., 2009, 10: 43 (doi: 10.1186/1471-2156-10-43).
- Zhang F., Wang Y., Mukiibi R., Chen L., Vinsky M., Plastow G., Basarab J., Stothard P., Li C. Genetic architecture of quantitative traits in beef cattle revealed by genome wide association studies of imputed whole genome sequence variants: I: feed efficiency and component traits. BMC Genomics, 2020, 21: 36 (doi: 10.1186/s12864-019-6362-1).