doi: 10.15389/agrobiology.2021.6.1099rus
УДК 636.5:573.6.086.83:577.21
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований, грант № 18-29-07079
СОЗДАНИЕ СИСТЕМ РЕДАКТИРОВАНИЯ ГЕНОМА НА ОСНОВЕ CRISPR-CAS9 ДЛЯ НОКАУТА ГЕНОВ FGF20 И HR В ЭМБРИОНАЛЬНЫХ И ГЕНЕРАТИВНЫХ КЛЕТКАХ КУР И ПЕРЕПЕЛОВ
А.Н. ВЕТОХ1 ✉, П.В. СЕРГИЕВ2, 3, 4, М.П. РУБЦОВА2, 5, Н.А. ВОЛКОВА1, Е.К. ТОМГОРОВА1, Л.А. ВОЛКОВА1,
Н.А. ЗИНОВЬЕВА1
Технологии геномного редактирования с применением сайт-специфических эндонуклеаз (ZNF, TALEN, CRISPR/Cas9) находят все более широкое применение в животноводстве, в том числе в птицеводстве. С их использованием связывают надежды не только на ускорение процесса создания пород с улучшенными хозяйственно полезными признаками, высокой устойчивостью к инфекционным заболеваниям, но и с созданием особей, несущих фенотипы, привнесение которых в популяции животных и птицы методами традиционной селекции невозможно или затруднено. Одним из направлений применения технологии геномного редактирования, представляющим интерес для промышленного птицеводства в рамках улучшения товарных качеств птицеводческой продукции, может быть создание особей, лишенных оперения. Для этого мы выбрали гены FGF20 и HR, связанные с развитием и ростом волос у млекопитающих (F. Benavides с соавт., 2009) и перьев у птиц (K.L. Wells с соавт., 2012). Цель исследования заключалась в создании системы для нокаута генов FGF20 и HR у кур и FGF20 у перепелов с использованием методов редактирования генома. Инактивацию генов FGF20 и HR проводили в области III экзона каждого гена с учетом анализа их структуры. С использованием биоинформатического инструментария и ресурсов (https://zlab.bio/guide-design-resources, https://www.ncbi.nlm.nih.gov/) были подобраны оптимальные участки разрезания генов FGF20 и HR, а также гидовые РНК и праймеры для амплификации выбранных участков этих генов. Для создания генетических конструкций, обеспечивающих разрезание в областях, кодирующих FGF20 и HR, был выбран вектор pX458 (F.A. Ran с соавт., 2013). Для лигирования использовали гибридизованные олигонуклеотиды: 5´-CACCGAAAGATGGTACTCC-CAGAGA-3´ и 3´-CTTTCTACCATGAGGGTCTCTCAAA-5´ (для гена FGF20 кур), 5´-CACCGTCC-ATGTTTGTACACGTTGG-3´ и 3´-CAGGTACAAACATGTGCAACCCAAA-5´ (для гена FGF20 кур и перепелов); 5´-CACCGACGTGGCTGACGCGGCACT-3´ и 3´-CTGCACCGACTGCGCCA-5´ (для гена HR кур). Эффективность клонирования конструкций подтвердило секвенирование. Полученные плазмиды использовались для редактирования генома эмбриональных (фибробласты) и генеративных (примордиальные зародышевые клетки — ПЗК, сперматогонии) клеток кур и перепелов в экспериментах in vitro. Клетки-мишени трансфицировали посредством электропорации. Эффективность трансфекции оценивали на сортере клеток BD FacsAria III («BD Biosciences», США) по экспрессии маркерного гена eGFP. Доля трансфицированных in vitro эмбриональных фибробластов, ПЗК и сперматогониев кур с нокаутом гена FGF20 достигала соответственно 5,7; 0,9 и 1,2 %, с нокаутом гена HR — 7,4, 0,8 и 1,0 %. Процент эмбриональных фибробластов, ПЗК и сперматогониев перепелов с нокаутом гена FGF20 составил соответственно 6,3; 0,9 и 1,1 %. Геномная ДНК была выделена из трансформированных клеток кур и перепелов и использована для амплификации и секвенирования участков генов FGF20 и HR, в которые были внесены делеции. Показано наличие множественных мутаций в амплифицированных участках ДНК. Полученные данные свидетельствуют об успешности создания систем для нокаута генов FGF20 и HR у кур и FGF20 у перепелов с использованием генетических конструкций на основе вектора pX458.
Ключевые слова: редактирование генома, CRISPR/Cas9, куры, перепела, примордиальные зародышевые клетки, сперматогонии, FGF20,HR.
Развитие технологий геномного редактирования с применением сайт-специфических эндонуклеаз (ZNF, TALEN, CRISPR/Cas9) открыло новые возможности для введения направленных генетических изменений в эмбриональные линии животных и птицы (1, 2). Эти технологии находят все более широкое применение благодаря высокой эффективности и специфичности (3). В животноводстве, в том числе в птицеводстве, с геномным редактированием связывают не только надежды на ускорение получения пород с улучшенными хозяйственно полезными признаками (4, 5), повышенной устойчивостью к инфекционным заболеваниям (6, 7), но и создание особей, несущих фенотипы, привнесение которых в популяции животных и птицы методами традиционной селекции невозможно или затруднено (8, 9). Получение линий сельскохозяйственной птицы, передающих привнесенные генетические изменения по наследству, требует проведения манипуляций на уровне генеративных клеток. Однако особенности эмбрионального развития и репродуктивной физиологии птиц не позволяют использовать для генно-инженерных манипуляций методы, применяемые на млекопитающих, например микроинъекции и соматическое клонирование (10, 11). Вместе с тем значительная часть эмбрионального периода у птиц проходит вне организма самок, что облегчает доступ к эмбриону на ранних стадиях развития. На сегодняшний день разработан достаточно широкий спектр методов и подходов по введению генетических конструкций в эмбриональные клетки птиц (12). Опубликован ряд работ по успешному получению генетически модифицированных кур и перепелов, экспрессирующих репортерные гены LacZ (13) и GFP (14), бактериальный ген β-лактамазы (15), интерферон человека α2b (16), β-интерферон человека (17), гранулоцитарный колониестимулирующий фактор человека (18), моноклональные антитела (19), антагонист рецептора β-интерлейкина (20) и гормон роста человека (21). Однако технологии, применявшиеся для модификации генома животных и птицы до 2012 года, носили неспецифичный характер, за исключением модельных научных систем. C 2012 года технология CRISPR/Cas9 последовательно вытесняет прочие приемы промышленного трангенеза. Имеется ряд работ по успешному использованию CRISPR/Cas9 для модификации клеток млекопитающих с последующим получением особей с заданными свойствами (22, 23). У птиц до недавнего времени в качестве наиболее эффективного способа привнесения наследственных изменений в генеративные клетки кур рассматривалась трансформация бластодермальных клеток на стадии Х с использованием лентивирусных и ретровирусных векторов (24). В последние годы с развитием новых методов редактирования генома, в частности TALEN и CRISPR, возрастает интерес к использованию в качестве клеток-мишеней для введения генетических конструкций генеративных клеток — примордиальных зародышевых клеток (ПЗК) и сперматогониев. ПЗК — это эмбриональные клетки, характеризующиеся плюриопотентностью, то есть способностью дифференцироваться как в мужские, так и женские половые клетки. Сперматогонии относят к стволовым клеткам семенников самцов. Сперматогонии представляют собой немногочисленную популяцию сперматогенных клеток, располагающихся на базальной мембране семенных канальцев. Они обладают способностью к самообновлению и дифференцировке с образованием высокоспециализированных зрелых половых клеток самцов — спермиев.
Такие свойства ПЗК и сперматогониев открывают широкие возможности для реализации их потенциала в качестве мишеней для геномного редактирования у сельскохозяйственной птицы с целью создания особей с заданными свойствами. Возможность манипуляций с клетками этого типа in vitro позволяет точно интегрировать экспрессионные конструкции в определенный локус под заранее выбранную эндогенную промоторно-энхансерную систему. Более того, эти технологии позволяют сохранить экспрессию эндогенного гена. Использование ПЗК в качестве вектора предусматривает их выделение из эмбрионов-доноров, трансформацию в культуре in vitro и введение в эмбрионы-реципиенты. При работе со сперматогониями проводят трансформацию донорских сперматогониев in vitro, отбор трансформированных клеток и их трансплантацию в семенники стерильных самцов-реципиентов с последующим получением спермы для осеменения самок с целью получения генетически модифицированного потомства.
Показаны возможности технологий TALEN и CRISPR для генетической модификации клеток кур in vitro (25, 26). Опубликован ряд работ по успешному получению кур и перепелов с заданными свойствами с использованием различных систем геномного редактирования (27, 28). Одно из возможных направлений геномного редактирования, представляющих практический интерес для улучшения товарных качеств птицеводческой продукции, — создание особей, лишенных оперения. Установлены гены (FGF20 и HR), необходимые для развития и роста волос у млекопитающих (29) и перьев у птиц (30).
В настоящем сообщении представлены результаты исследований по созданию системы редактирования на основе CRISPR/Cas9 для нокаута генов FGF20 и HR, контролирующих развитие оперения у птиц. На эмбриональных фибробластах и генеративных клетках (ПЗК и сперматогонии) показана эффективность использования созданных конструкций для внесения делеций в области генов FGF20 и HR кур и перепелов в условиях in vitro.
Нашей целью была разработка системы редактирования генома для нокаута генов FGF20 и HR у кур и перепелов, пригодной для получения генетически модифицированной сельскохозяйственной птицы.
Методика. Эмбриональные фибробласты выделяли из 5-суточных эмбрионов кур (Gallus gallus domesticus, порода русская белая) или 4-суточных эмбрионов перепелов (Coturnix coturnix, порода японский перепел). Эмбрионы извлекали в асептических условиях. Для дезагрегации эмбрионы сначала механически измельчали ножницами, затем подвергали ферментативной обработке, инкубируя кусочки ткани в 0,15 % растворе трипсина («Gibco, Thermo Scientific», США) в течение 15 мин при 37 °С. Примордиальные зародышевые клетки выделяли из 6-суточных эмбрионов кур и 4-суточных эмбрионов перепелов. Диссоциацию проводили посредством последовательной механической и ферментативной обработки, как описано выше, но при концентрации трипсина 0,05 %. Сперматогенные клетки выделяли посредством последовательной механической и ферментативной обработки ткани семенника 1-недельных самцов. Для ферментативной обработки использовали 0,25 % раствор трипсина, инкубировали в течение 30 мин при 37 °С.
Долю жизнеспособных клеток в полученной клеточной суспензии после механической и ферментативной обработки эмбрионов и ткани семенника оценивали с помощью окрашивания 0,4 % трипановым синим в течение 10 мин при 37 °С. Подсчет окрашенных клеток проводили в счетчике клеток Countess («Thermo Fisher Scientific», США).
Суспензии эмбриональных клеток фибробластов и ПЗК, полученные после ферментативной обработки эмбрионов, переносили в чашки Петри и культивировали в ростовой среде DMEM HG («Gibco, Thermo Scientific», США) с высоким содержанием глюкозы (4,5 г/л) и добавлением 10 % сыворотки плода коровы (Fetal bovine serum, FBS, «HyClone», США), глутамина (2 мМ), 2-меркаптоэтанола (10-6 мМ) и антибиотика гентамицина (50 мкг/мл). Для первичной культуры клеток семенника в качестве ростовой использовали среду DMEM HG с содержанием глюкозы 4,5 г/л, дополненную 20 % FBS, альфа-глутамином (2 мМ), МЕМ (100×), антибиотиком-антимикотиком (100×) и ITS (100×). Культивирование сперматогониев осуществляли в среде DMEM HG, дополненной 5 % FBS, 2 мМ альфа-глутамином, МЕМ (10 мкл/мл), антибиотиком антимикотиком (100×), ITS (10 мкл/мл), меркаптоэтанолом (5×10-5 М), альбумином (5 мг/мл), DL-lactic acid (1 мкл/мл), EGF (20 нг/мл), bFGF (10 нг/мл), LIF (2 нг/мл).
В составе культуральных сред использовали ростовые добавки, аминокислоты и антибиотики производства «Gibco» («Thermo Scientific», США) и «Sigma» (США).
Клетки кур и перепелов культивировали при 37 °С и 5 % СО2. Для пассирования и проведения молекулярно-генетических исследований клетки снимали с субстрата 0,25 % раствором трипсина.
Микроскопирование полученных культур эмбриональных фибробластов, ПЗК и сперматогониев кур и перепелов проводили с помощью инвертированного микроскопа Nikon Eclipse TS100 («Nikon», Япония).
Последовательности гидовых РНК выбирали с помощью интернет ресурсов (https://zlab.bio/guide-design-resources, https://www.ncbi.nlm.nih.gov/), используя вариант сборки генома курицы GalGal5 и названия генов FGF20 (Gene ID: 428779) и HR (Gene ID: 107049623) и перепела Coturnix japonica 2.0 и название гена FGF20 (Gene ID: 107313688), были выбраны оптимальные участки разрезания гена FGF20 кур и HR перепелов. Для создания генетических конструкций для разрезания выбранных участков генома были гибридизованы пары следующих олигонуклеотидов: 5´-CACCGAAAGATG-GTACTCCCAGAGA-3´ и 3´-CTTTCTACCATGAGGGTCTCTCAAA-5´ (для гена FGF20 кур), 5´-CACCGTCCATGTTTGTACACGTTGG-3´ и 3´-CAGG-TACAAACATGTGCAACCCAAA-5´ (для гена FGF20 кур и перепелов); 5´-CA-CCGACGTGGCTGACGCGGCACT-3´ и 3´-CTGCACCGACTGCGCCA-5´ (для гена HR кур). Гибридизованные олигонуклеотиды были лигированы с плазмидой pX458 (Addgene #48138), линеаризованной эндонуклеазой рестрикции BbsI (ER1011, «Thermo Scientific», США) в соответствии с описанием (31). После лигирования и трансформации клеток Escherichia coli выросшие колонии пересевали в жидкую среду LB с ампициллином и использовали для выделения плазмид. Анализ результатов клонирования проводили с помощью секвенирования. Успешно клонированные конструкции использовали для трансфекции клеток.
Для выделения геномной ДНК при создании конструктов и оценке эффективность разрезания целевого участка генома использовали коммерческий набор QuickExtract™ DNA Extraction Solution («Lucigen Corporation», США) в соответствии с рекомендациями производителя. Амплификацию проводили в ПЦР смеси PCR MM на основе Taq ДНК полимеразы (K0171, «Thermo Scientific», США) в объеме 25 мкл при температуре гибридизации 60 °С и элонгации 1 мин при 72 °С. Для амплификации участка гена FGF20 использовали праймеры F20_CHK2F 5´-TGTTCCTTTGTGCAGGAGAA-ACT-3´ и F20_CHK2R 5´-TCCCTCTCTCCTCAGCTGTATC-3´, гена HR — HR_CHK2F 5´-CGCGGCCTTCCCGTG-3´ и HR_CHK2R 5´-GGCCGTTC-GGCCGTTT-3´.
Системы редактирования вводили в эмбриональные фибробласты, ПЗК и сперматогонии методом электропорации, используя Neon™ Transfec-tion System («Invitrogen», США). Трансфицированные клетки отбирали с помощью высокопроизводительного клеточного сортера BD FACSAria III («BD Biosciences», США).
Эффективность разрезания целевого участка генома оценивали посредством амплификации и секвенирования соответствующих фрагментов геномной ДНК с разработанными праймерами в указанных выше режимах (последовательности амплифицируемых фрагментов представлены на рисунках 1 и 2). Секвенирование (выполнено в компании ООО «Синтол», г. Москва) проводили по методу Сэнгера с использованием прямого праймера для амплификации.
Результаты. На первом этапе разработки системы геномного редактирования CRISPR/Cas9 для нокаута генов FGF20 и HR мы провели биоинформатический поиск гомолога гена HR у птиц и подобрали последовательности гидовых РНК для инактивации генов FGF20 и HR.
Ген FGF20 курицы G. gallus domesticus расположен на 4-й хромосоме (рис. 1, А). Для инактивации этого гена мы выбрали III экзон. На рисунке представлены подобранные с использованием интернет-сервера участки разрезания в гене FGF20 кур и перепелов, оптимальные для внесения в геном целевых мутаций с помощью системы CRISPR/Cas9, а также участки гибридизации двух гидовых РНК и праймеров для амплификации выбранных участков гена FGF20 (см. рис. 1, Б). Одна из гидовых РНК была универсальной для курицы и перепела, вторая — комплементарной только для курицы. Праймеры, подобранные для амплификации выбранных участков гена FGF20, соответствовали геному как курицы, так и перепела.
Ген HR расположен на 22-й хромосоме курицы (рис. 2, А). Для его инактивации был выбран III экзон, кодирующий аминокислоты активного центра фермента HR, представляющего собой лизин-деметилазу. Показан участок генома курицы G. gallus domesticus с подобранными участками гибридизации праймеров и гидовой РНК (рис. 2, Б).
Олигонуклеотиды для выбранных гидовых РНК синтезировали и использовали при создании конструкций для инактивации генов FGF20 и HR. Плазмиду pX458 обрабатывали эндонуклеазой рестрикции BbsI и по сайту разрезания лигировали с гибридизованными олигонуклеотидами F20C, F20U и HR (рис. 3).
После лигирования и трансформации компетентных клеток E. coli JM109 (32) получили клоны трансформированных клеток, из которых выделяли плазмиды. Успешность клонирования конструкций подтвердило секвенирование полученных плазмид, которые далее использовали для трансфекции клеток кур и перепелов.
Эффективность систем редактирования, разработанных для нокаута генов FGF20 и HR, сначала оценили на эмбриональных фибробластах ввиду простоты и доступности их получения. Эмбриональные фибробласты кур и перепелов с плазмидой, кодирующей компоненты системы геномного редактирования CRISPR/Cas9, отделяли от нетрансфицированных клеток при помощи клеточного сортера (рис. 4). Разделение клеток основано на том, что в плазмиде pX458 присутствуют области, соответствующие генам Cas9 иGFP, кодирующие области которых отделены друг от друга последовательностью, кодирующей P2А пептид. Таким образом, клетки, в которых происходит синтез Cas9, также содержат GFP и могут быть отделены от нетрансфицированных клеток по флуоресценции (см. рис. 4). Доля успешно трасфицированных эмбриональных клеток кур (размер выборки 10 000 клеток) при использовании систем для нокаута генов FGF20 и HR достигала соответственно 5,7 и 7,4 %. Согласно распределению клеток по интенсивности флуоресценции, эффективность трансфекции эмбриональных клеток перепелов посредством системы редактирования для нокаута гена FGF20 составила 6,3 %.
Трансфицированные эмбриональные фибробласты кур и перепелов, отсортированные на сортере, использовали для выделения геномной ДНК с целью оценки эффективности редактирования генов FGF20 и HR. Выделенная ДНК была использована для ПЦР-амплификации локусов, содержащих участки комплементарности гидовым РНК (см. рис. 1, рис. 2). Выделенные ПЦР-продукты проанализировали с помощью секвенирования по Сэнгеру. В проанализированной популяции трансфицированных эмбриональных фибробластов кур (рис. 5, рис. 6) и перепелов выявили множественные микроделеции.
Анализ полученных данных (см. рис. 5, рис. 6) позволил сделать вывод о высокой эффективности внесения микроделеций в гены FGF20 и HR с помощью созданных нами конструкций. Из двух конструкций, нацеленных на ген FGF20, более эффективной оказалась конструкция F20U, которую использовали для дальнейших экспериментов вместе с конструкцией HR.
При получении генетически модифицированной птицы наибольший интерес представляет возможность направленной модификации клеток гонад с целью их дальнейшего использования для получения потомства с измененным геномом. Для этих целей могут использоваться как зрелые половые клетки, так и их предшественники — ПЗК и сперматогонии.
При дезагрегации эмбрионов кур и перепелов с применением механической и ферментативной обработки мы получили суспензию разобщенных клеток (доля нежизнеспособных не превышала 5 %). В суспензии присутствовали разные типы эмбриональных клеток, разделение которых по адгезии (33) позволило получить популяцию эмбриональных клеток, максимально обогащенную ПЗК. Доля ПЗК от общего числа других типов клеток в культуре эмбриональных клеток кур и перепелов достигала соответственно 88 и 81 %. Немногочисленная популяция фибробластов, оставшаяся после разделения клеток, служила фидерным слоем, на который прикреплялись и культивировались ПЗК, образуя колонии (рис. 7).
После дезагрегации ткани семенников петушков и перепелов с при-менением раствора трипсина получили суспензию, состоящую преимущественно из клеток Сертоли и сперматогониев. При дальнейшем культивировании клетки Сертоли распластывались на поверхности чашек Петри. Сперматогонии прикреплялись к клеткам Сертоли, образуя колонии на 7-8-е сут культивирования (рис. 8).
Полученные культуры ПЗК и сперматогониев кур и перепелов использовали для трансфекции созданными конструкциями для нокаута генов FGF20 и HR. Доля трансфицированных in vitro ПЗК и сперматогониев кур с нокаутом гена FGF20 достигала соответственно 0,9 и 1,2 %, с нокаутом гена HR — 0,8 и 1,0 %. Доля ПЗК и сперматогониев перепелов с нокаутом гена FGF20 составила соответственно 0,9 и 1,1 %. Следует отметить относительно низкую эффективность трансфекции этих клеток-мишеней, однако благодаря отбору на сортере можно получить чистую популяции трансфицированных клеток и размножить их до необходимых объемов в условиях in vitro.
Результативность использования ПЗК в качестве клеток-мишеней при геномном редактировании для получения птицы с нокаутом разных генов изучена несколькими научными группами: показана эффективность получения кур с нокаутом генов миостатина (5), тяжелой цепи иммуноглобулина (34), DDX4 (35), овомуцина (8), NHE1 (6) с применением систем CRISPR/Cas9 (5, 6, 8, 34) и TALEN (35). В упомянутых публикациях для трансфекции ПЗК использовали два метода — электропорацию (6, 34, 35) и липофекцию (5, 8). При использовании метода электропорации в большинстве указанных случаев для отбора трансфицированных клеток осуществляли их культивирование в ростовых средах, содержащих селективный антибиотик. При отборе трансфецированных ПЗК посредством сортера без их предварительного культивирования в селективной среде эффективность трансфекции была невысокой (1 %), что согласуется с полученными нами данными.
Информация по использованию сперматогониев в качестве мишеней для введения систем редактирования генома в изученных нами доступных информационных источниках не была найдена.
Таким образом, нами получены системы геномного редактирования для нокаута генов FGF20 и HR у кур и гена FGF20 уперепелов. С использованием биоинформатического поиска подобраны оптимальные участки разрезания данных генов, а также гидовые РНК и праймеры для амплификации выбранных участков ДНК-мишеней. На основе вектора pX458 созданы генетические конструкции для внесения делеций в областях, кодирующих FGF20 и HR. Показана эффективность внесения микроделеций в указанные гены кур и перепелов с использованием полученных конструкций в системе in vitro: на культурах эмбриональных фибробластов частота трансфекции — соответственно 5,7 и 6,3 %, ПЗК — 0,9 и 0,9 %, сперматогониев — 1,2 и 1,1 %. Эти результаты свидетельствуют об успешном создании систем редактирования генома у сельскохозяйственной птицы с использованием генетических конструкций на основе вектора pX458.
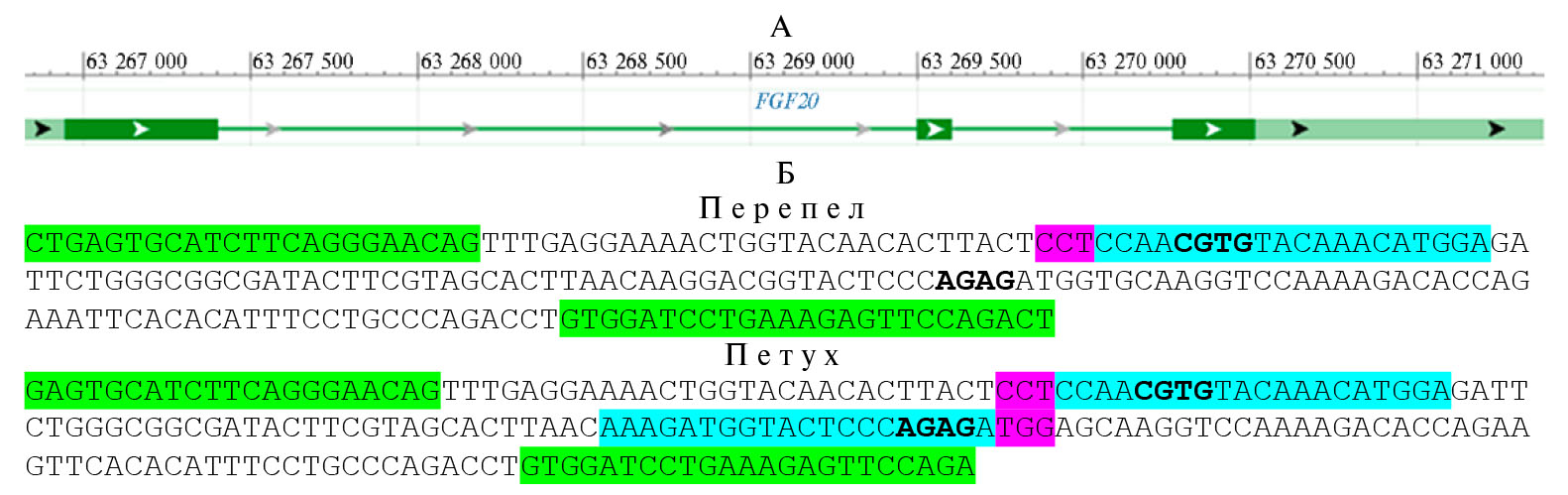
Рис. 1. Схема гена FGF20 (А) и участки гибридизации гидовых РНК к нему (Б) у Gallus gallus domesticus и Coturnix japonica. Показана геномная координата 4-й хромосомы G. gallus domesticus с расположением экзонных (толстые линии) и интронных (тонкие линии) участков гена. Светло зеленым отмечены некодирующие, темно-зеленым — кодирующие участки. Стрелки показывают направление транскрипции. Представлено выравнивание участков генома перепела C. japonica и курицы G. gallus domesticus с участками гибридизации разработанных праймеров (помечено зеленым) и гидовых РНК (помечено бирюзовым). PAM (protospacer adjacent motive) сайты показаны фиолетовым.
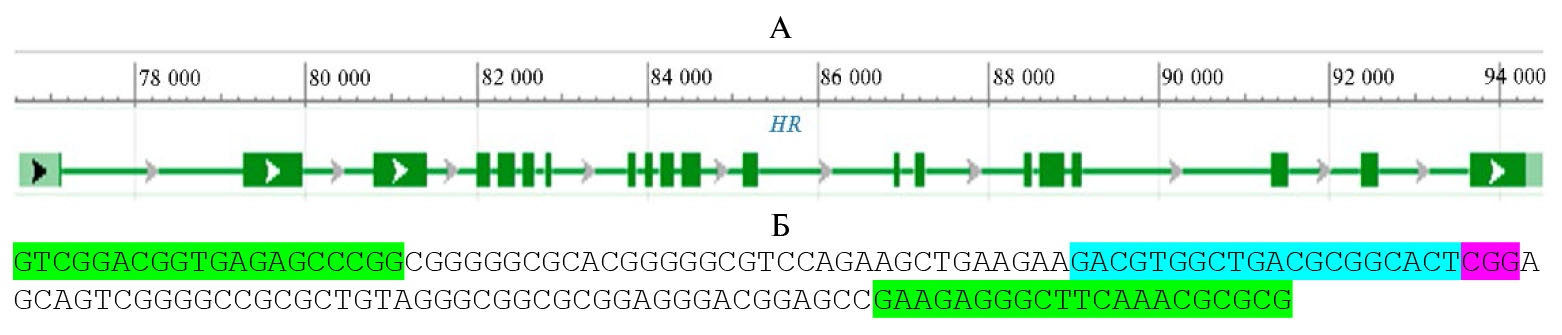
Рис. 2. Схема гена HR (А) и участка гибридизации гидовых РНК к нему (Б) у Gallus gallus domesticus. Показана геномная координата 22-й хромосомы G. gallus domesticusи экзонных (толстые линии) и интронных (тонкие линии) участков гена HR. Светло-зеленым отмечены некодирующие, темно-зеленым — кодирующие участки. Стрелки показывают направление транскрипции. Представлен участок генома G. gallus domesticus с участками гибридизации праймеров (помечено зеленым) и гидовых РНК (помечено бирюзовым). PAM (protospacer adjacent motive) сайты показаны фиолетовым.

Рис. 3. Олигонуклеотиды, определяющие специфичность гидовых РНК и использованные для создания плазмид для инактивации генов FGF20 и HR методом CRISPR/Cas9.
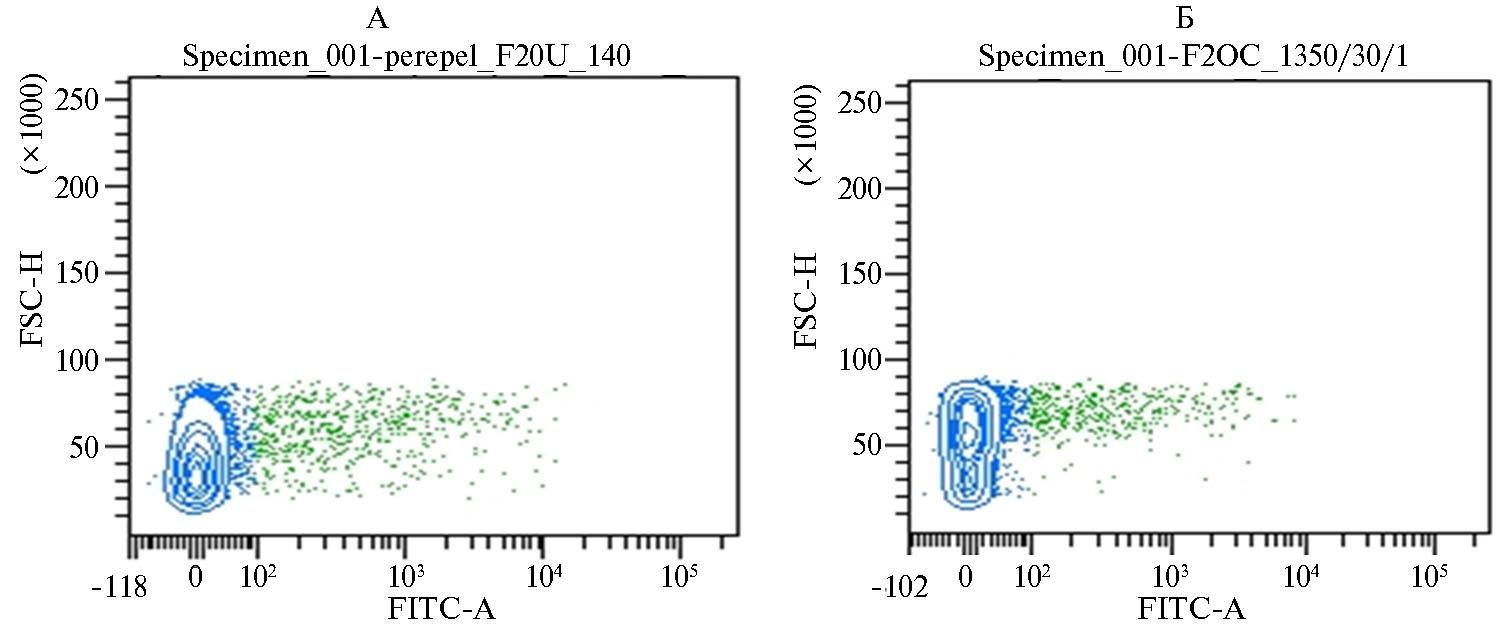
Рис. 4. Пример результата сортировки популяции клеток эмбриональных фибробластов перепелов (A) и кур (Б) после трансфекции конструкциями на основе плазмиды pX458. По оси X показана интенсивность флуоресценции в зеленом диапазоне, по оси Y — светорассеяние. Зеленым отмечены клетки, обладающие зеленой флуоресценцией.
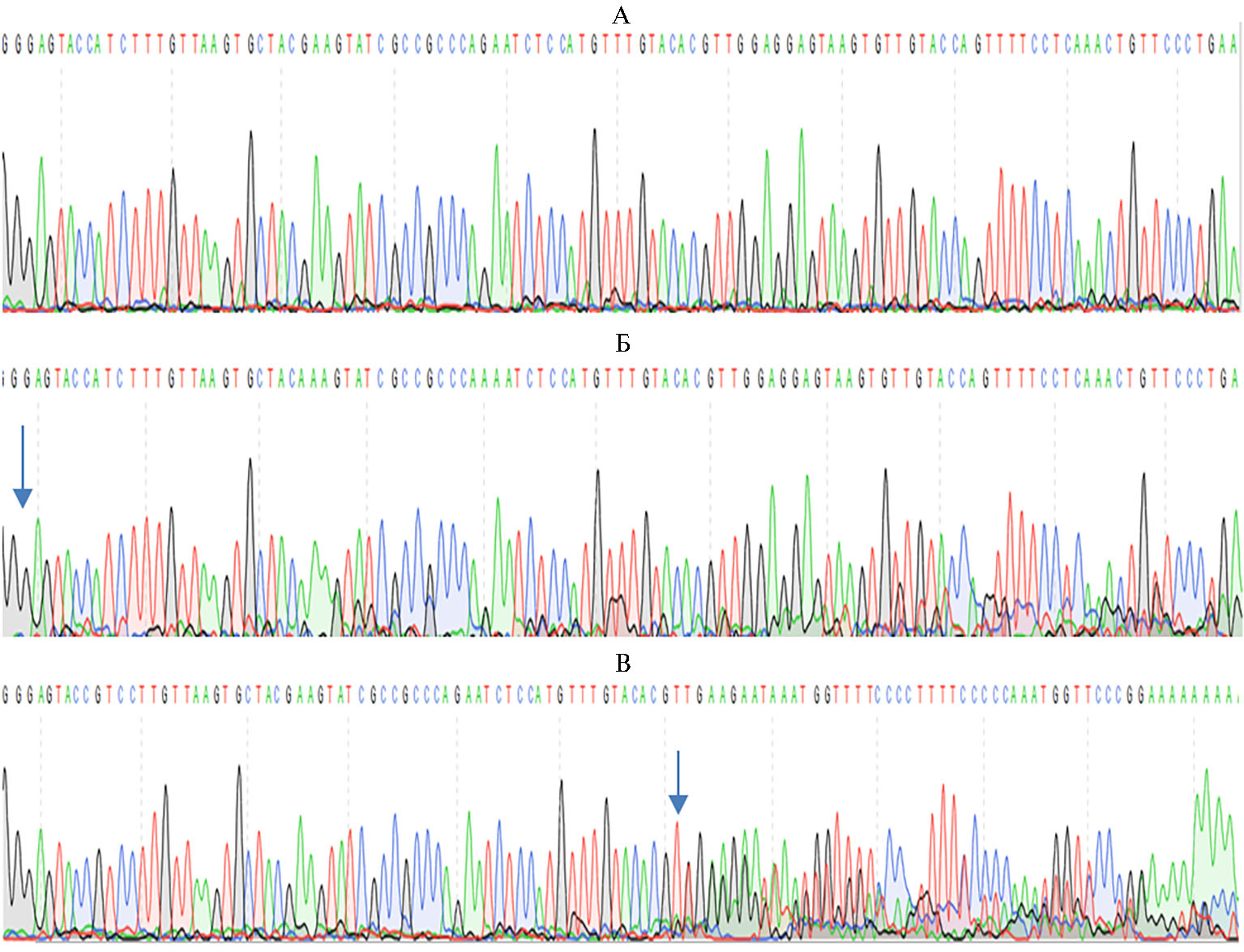
Рис. 5. Анализ эффективности разработанной системы CRISPR/Cas9-редактирования гена FGF20 методом секвенирования по Сэнгеру: А — ген FGF20 курицы, не подвергнутый редактированию; Б — ген FGF20 курицы после редактирования при помощи Cas9 и гидовой РНК F20C; В — ген FGF20 курицы после редактирования при помощи Cas9 и гидовой РНК F20U. Стрелками показаны участки разрезания генов. Видно наложение результатов секвенирования продуктов множественных микроделеций. При секвенировании гетерогенных продуктов микроделеций, в отличие от гомогенного ампликона дикого типа, происходит наложение пиков, соответствующих разным нуклеотидам (зеленый — A, черный — G, синий — C, красный — T).
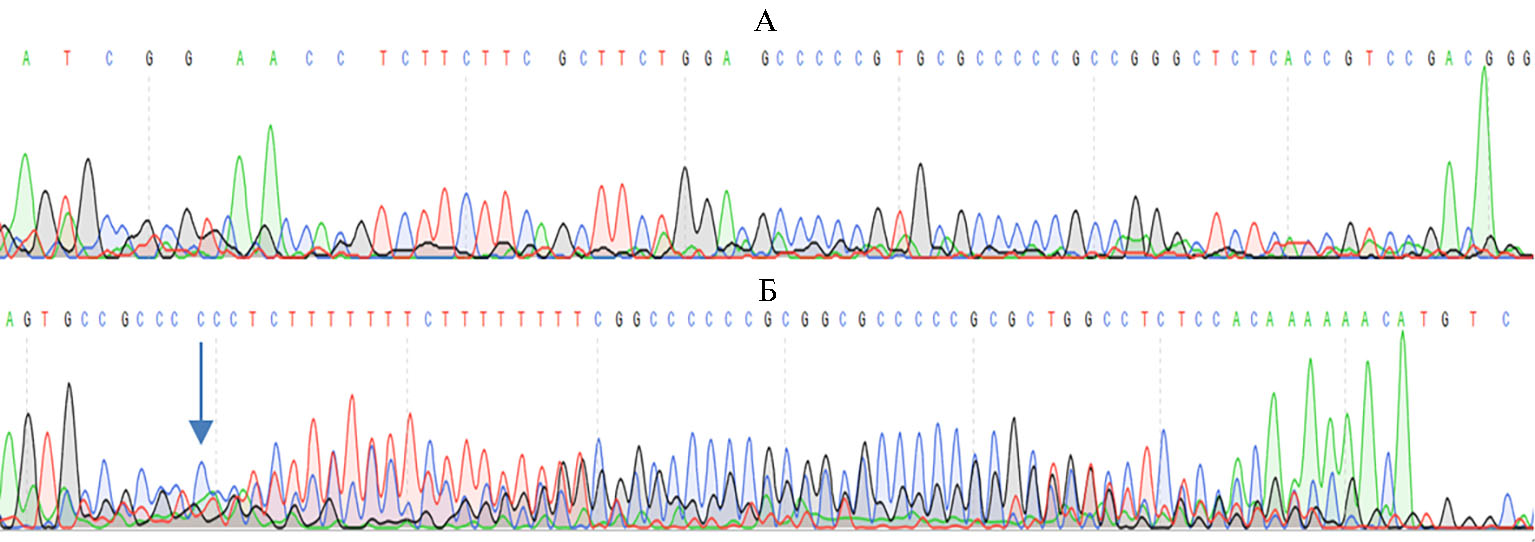
Рис. 6. Анализ эффективности разработанной системы CRISPR/Cas9-редактирования гена HR методом секвенирования по Сэнгеру: А — ген HR курицы, не подвергнутый редактированию; Б — ген HR курицы после редактирования при помощи Cas9 и гидовой РНК HR. Стрелками показаны участки разрезания генов. Видно наложение результатов секвенирования продуктов множественных микроделеций. При секвенировании гетерогенных продуктов микроделеций, в отличие от гомогенного ампликона дикого типа, происходит наложение пиков, соответствующих разным нуклеотидам (зеленый — A, черный — G, синий — C, красный — T).
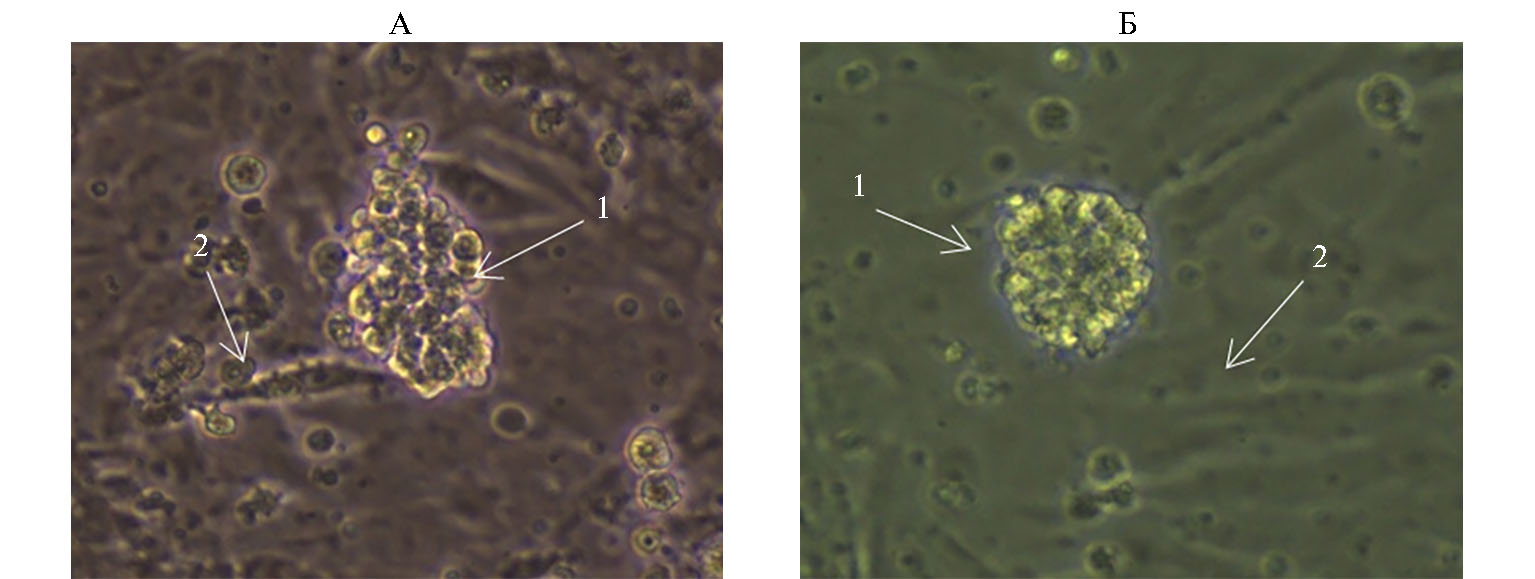
Рис. 7. Колонии примордиальных зародышевых клеток (ПЗК) кур (А) и перепелов (Б), использованные при трансфекции конструкциями для нокаута генов FGF20 и HR: 1 — ПЗК, 2 — фибробласты (фидерный слой). Нативный препарат, световая микроскопия (микроскоп Nikon Eclipse TS100, «Nikon Сo.», Япония; увеличение ×400).
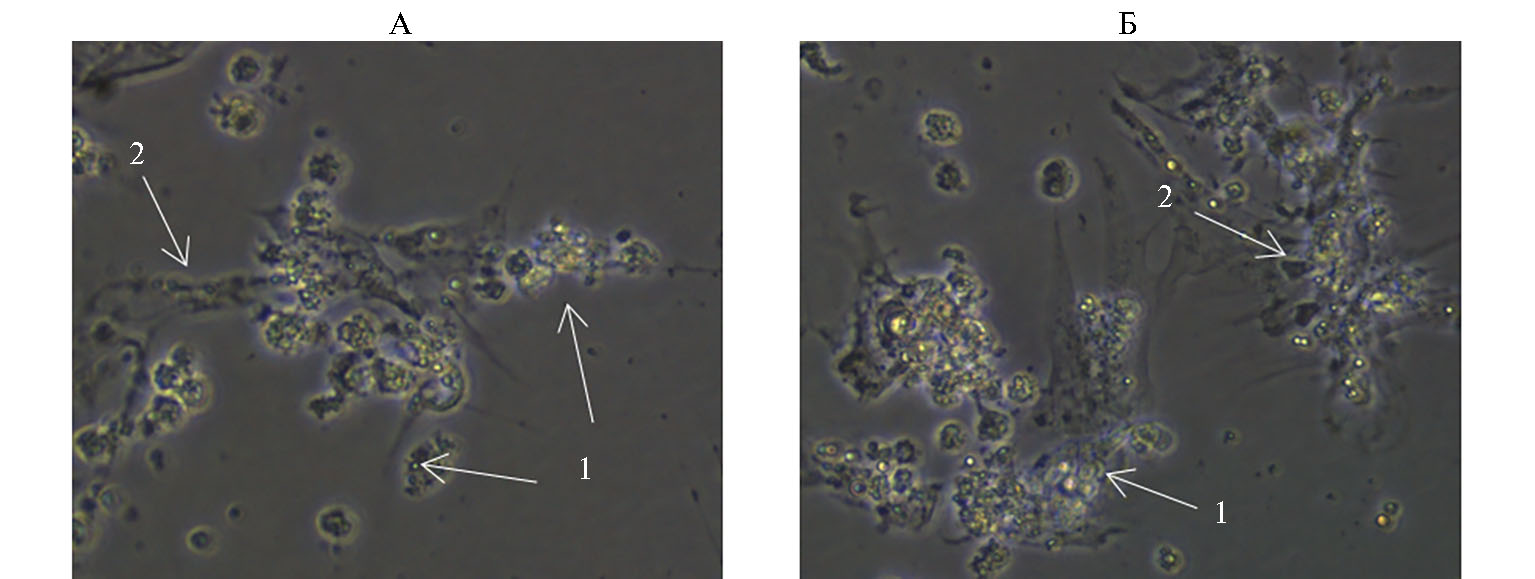
Рис. 8. Культуры сперматогенных клеток семенника петуха (А) и перепела (Б), использованные при трансфекции конструкциями для нокаута генов FGF20 и HR: 1 — сперматогонии, 2 — клетки Сертоли. Нативный препарат, световая микроскопия (микроскоп Nikon Eclipse TS100, «Nikon Сo.», Япония; увеличение ×400).
ЛИТЕРАТУРА
- Bahrami S., Amiri-Yekta A., Daneshipour A., Jazayeri S.H., Mozdziak P.E., Sanati M.H., Gourabi H. Designing a transgenic chicken: applying new approaches toward a promising bioreactor. Cell Journal, 2020, 22(2): 133-139 (doi: 10.22074/cellj.2020.6738).
- Cooper C. A., Doran T.J., Challagulla A., Tizard M.L.V., Jenkins K.A. Innovative approaches to genome editing in avian species. Journal of Animal Science and Biotechnology, 2018, 9: 15 (doi: 10.1186/s40104-018-0231-7).
- Petersen B. Basics of genome editing technology and its application in livestock species. Reproduction in Domestic Animals, 2017, 52(S3): 4-13 (doi: 10.1111/rda.13012).
- Bhattacharya T.K., Shukla R., Chatterjee R.N., Bhanja S.K. Comparative analysis of silencing expression of myostatin (MSTN) and its two receptors (ACVR2A and ACVR2B) genes affecting growth traits in knock down chicken. Scientific Reports, 2019, 9: 7789 (doi: 10.1038/s41598-019-44217-z).
- Kim G-D., Lee J.H., Song S., Kim S.W., Han J.S., Shin S.P., Park B.-C., Park T.S. Generation of myostatin-knockout chickens mediated by D10A-Cas9 nickase. FASEB, 2020, 34: 5688-5696 (doi: 10.1096/fj.201903035R).
- Hellmich R., Sid H., Lengyel K., Flisikowski K., Schlickenrieder A., Bartsch D., Thoma T., Bertzbach L.D., Kaufer B.B., Nair V., Preisinger R., Schusser B. Acquiring resistance against a retroviral infection via CRISPR/Cas9 targeted genome editing in a commercial chicken line. Frontiers in Genome Editing, 2020, 2: 3 (doi: 10.3389/fgeed.2020.00003).
- Koslová A., Trefil P., Mucksová J., Reinišová M., Plachý J., Kalina J., Kučerová D., Geryk J., Krchlíková V., Lejčková B., Hejnar J. Precise CRISPR/Cas9 editing of the NHE1 gene renders chickens resistant to the J subgroup of avian leukosis virus. PNAS, 2020, 117(4): 2108-2112 (doi: 10.1073/pnas.1913827117).
- Oishi I., Yoshii K., Miyahara D., Kagami H., Tagami T. Targeted mutagenesis in chicken using CRISPR/Cas9 system. Scientific Reports, 2016, 6: 23980 (doi: 10.1038/srep23980).
- Oishi I., Yoshii K., Miyahara D., Tagami T. Efficient production of human interferon beta in the white of eggs from ovalbumin gene-targeted hens. Scientific Reports, 2018, 8: 10203 (doi: 10.1038/s41598-018-28438-2).
- Stern C.D. The marginal zone and its contribution to the hypoblast and primitive streak of the chick embryo. Development, 1990, 109: 667-682 (doi: 10.1242/dev.109.3.667).
- Mozdziak P.E., Petitte J.N. Status of transgenic chicken models for developmental biology. Developmental Dynamics, 2004, 229: 414-421 (doi: 10.1002/dvdy.10461).
- Коршунова Л.Г., Карапетян Р.В., Зиадинова О.Ф., Фисинин В.И. Трансгенная птица — создание и области применения (обзор). Сельскохозяйственнаябиология, 2019, 54(6): 1080-1094 (doi: 10.15389/agrobiology.2019.6.1080rus).
- Mozdziak P.E., Borwornpinyo S., Mccoy D.W., Petitte J.N. Development of transgenic chickens expressing bacterial beta-galactosidase. Developmental Dynamics, 2003, 226: 439-445 (doi: 10.1002/dvdy.10234).
- Byun S.J., Kim S.W., Kim K.W., Kim J.S., Hwang I.S., Chung H.K., Kan I.S., Jeon I.S., Chang W.K., Park S.B., Yoo J.G. Oviduct-specific enhanced green fluorescent protein expression in transgenic chickens. Bioscience, Biotechnology, and Biochemistry, 2011, 75(4): 646-649 (doi: 10.1271/bbb.100721).
- Harvey A.J., Speksnijder G., Baugh L.R., Morris J.A., Ivarie R. Expression of exogenous protein in the egg white of transgenic chickens. Nature Biotechnology, 2002, 20: 396 (doi: 10.1038/nbt0402-396).
- Rapp J.C., Harvey A.J., Speksnijder G.L., Hu W., Ivarie R. Biologically active human interferon a-2b produced in the egg white of transgenic hens. Transgenic Research, 2003, 12(5): 569-575 (doi: 10.1023/A:1025854217349).
- Lillico S.G., Sherman A., McGrew M.J., Robertson C.D., Smith J., Haslam C., Barnard P., Radcliffe P.A., Mitrophanous K.A., Elliot E.A., Sang H.M. Oviduct-specific expression of two therapeutic proteins in transgenic hens. PNAS, 2007, 104(6): 1771-1776 (doi: 10.1073/pnas.0610401104).
- Kwon M.S., Koo B.C., Choi B.R., Park Y.Y., Lee Y.M., Suh H.S., Park Y.S., Lee H.T., Kim J.H., Roh J.Y., Kim N.H., Kim T. Generation of transgenic chickens that produce bioactive human granulocyte-colony stimulating factor. Molecular Reproduction and Development, 2008, 75(7): 1120-1126 (doi: 10.1002/mrd.20860).
- Kamihira M., Ono K., Esaka K., Nishijima K., Kigaku R., Komatsu H., Yamashita T., Kyogoku K., Iijima S. High-level expression of single-chain Fv-Fc fusion protein in serum and egg white of genetically manipulated chickens by using a retroviral vector. Journal of Virology, 2005, 79(17): 10864-10874 (doi: 10.1128/JVI.79.17.10864-10874.2005)
- Kwon S.C., Choi J.W., Jang H.J., Shin S.S., Lee S.K., Park T.S., Choi I.Y., Lee G.S., Song G., Han J.Y. Production of biofunctional recombinant human interleukin 1 receptor antagonist (rhIL1RN) from transgenic quail egg white. Biology of Reproduction, 2010, 82: 1057-1064 (doi: 10.1095/biolreprod.109.081687).
- Kodama D., Nishimiya D., Nishijima K., Okino Y., Inayoshi Y., Kojima Y., Ono K., Motono M., Miyake K,. Kawabe Y., Kyogoku K., Yamashita T., Kamihira M., Iijima S. Chicken oviduct-specific expression of transgene by a hybrid ovalbumin enhancer and the Tet expression system. Journal of Bioscience and Bioengineering, 2012, 113(2): 146-153 (doi: 10.1016/j.jbiosc.2011.10.006).
- Li W.R., Liu C.X., Zhang X.M., Chen L., Peng X.R., He S.G., Lin J.P., Han B, Wang L.Q., Huang J.C., Liu M.J. CRISPR/Cas9-mediated loss of FGF5 function increases wool staple length in sheep. FEBS Journal, 2017 284(17): 2764-2773 (doi: 10.1111/febs.14144).
- Wang X., Niu Y., Zhou J., Zhu H., Ma B., Yu H., Yan H., Hua J., Huang X., Qu L., Chen Y. CRISPR/Cas9-mediated MSTN disruption and heritable mutagenesis in goats causes increased body mass. Animal Genetics, 2018, 49(1): 43-51 (doi: 10.1111/age.12626).
- Scott B.B., Velho T.A., Sim S., Lois C. Applications of avian transgenesis. ILAR Journal, 2010, 51(4): 353-361 (doi: 10.1093/ilar.51.4.353)).
- Véron N., Qu Z., Kipen P.A.S., Hirst C.E., Marcelle C. CRISPR mediated somatic cell genome engineering in the chicken. Developmental Biology, 2015, 407(1): 68-74 (doi: 10.1016/j.ydbio.2015.08.007).
- Park T.S., Lee H.J., Kim K.H., Kim J.-S., Han J.Y. Targeted gene knockout in chickens mediated by TALENs. PNAS, 2014, 111(35): 12716-12721 (doi: 10.1073/pnas.1410555111).
- Cooper C.A., Challagulla A., Jenkins K.A., Wise T.G., O’Neil T.E., Morris K.R., Tizard M.L, Doran T.J. Generation of gene edited birds in one generation using sperm transfection assisted gene editing (STAGE). Transgenic Research, 2017, 26: 331-347 (doi: 10.1007/s11248-016-0003-0).
- Lee J., Kim D. H., Lee K. Muscle hyperplasia in Japanese quail by single amino acid deletion in MSTN propeptide. International Journal of Molecular Sciences, 2020, 21: 1504 (doi: 10.3390/ijms21041504).
- Benavides F., Oberyszyn T.M., Van Buskirk A.M., Reeved V.E., Kusewit D.F. The hairless mouse in skin research. Journal of Dermatological Science, 2009, 53(1): 10-18 (doi: 10.1016/j.jdermsci.2008.08.012).
- Wells K.L., Hadad Y., Ben-Avraham D., Hillel J., Cahaner A., Headon D.J. Genome-wide SNP scan of pooled DNA reveals nonsense mutation in FGF20 in the scaleless line of featherless chickens. BMC Genomics, 2012, 13: 257 (doi: 10.1186/1471-2164-13-257).
- Ran F., Hsu P., Wright J., Agarwala V., Scott D.A., Zhang F. Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 2013, 8: 2281-2308 (doi: 10.1038/nprot.2013.143).
- Inoue H., Nojima H., Okayama H. High efficiency transformation of Escherichia coli with plasmids. Gene, 1990, 96(1): 23-28 (doi: 10.1016/0378-1119(90)90336-P).
- Волкова Н.А., Багиров В.А., Томгорова Е.К., Ветох А.Н., Волкова Л.А., Зиновьева Н.А. Выделение, культивирование и характеристика примордиальных зародышевых клеток перепелов. Сельскохозяйственнаябиология, 2017, 52(2): 261-267 (doi: 10.15389/agrobiology.2017.2.261rus).
- Dimitrov L., Pedersen D., Ching K.H., Yi H., Collarini E. J., Izquierdo S., van de Lavoir M.-C., Leighton P.A. Germline gene editing in chickens by efficient CRISPR-mediated homologous recombination in primordial germ cells. PLoS ONE, 2016, 11: e0154303 (doi: 10.1371/journal.pone.0154303).
- Dimitrov L., Pedersen D., Ching K.H., Yi H., Collarini E. J., Izquierdo S., van de Lavoir M.-C., Leighton P.A. Germline gene editing in chickens by efficient CRISPR-mediated homologous recombination in primordial germ cells. PLoS ONE, 2016, 11: e0154303 (doi: 10.1371/journal.pone.0154303).
- Taylor L., Carlson D.F., Nandi S., Sherman A., Fahrenkrug S.C., McGrew M.J. Efficient TALEN-mediated gene targeting of chicken primordial germ cells. Development, 2017, 144: 928-934 (doi: 10.1242/dev.145367).