doi: 10.15389/agrobiology.2021.6.1123rus
УДК 636.2:577.21:575.174:575.2
Работа выполнена при поддержке гранта 19-76-20061 РНФ (http://rscf.ru).
ГЕНЕТИЧЕСКАЯ СТРУКТУРА АБОРИГЕННОГО ТАГИЛЬСКОГО СКОТА ПО STR- И SNP-МАРКЕРАМ
Ю.А. СТОЛПОВСКИЙ1 ✉, С.В. БЕКЕТОВ1, Е.В. СОЛОДНЕВА1, B.М. АБСАЛИКОВ2, А.С. АБДЕЛЬМАНОВА3,
Е.А. ГЛАДЫРЬ3, Н.А. ЗИНОВЬЕВА3
Разведение специализированных пород или нескольких внутрипородных линий снижает породное и генетическое разнообразие поголовья, и, как следствие, создается реальная угроза исчезновения аборигенного скота. Для изучения популяционно-генетической структуры местных пород, обладающих уникальными адаптивными признаками и устойчивостью к ряду заболеваний, чаще всего используют микросателлитный анализ либо более информативный метод полногеномного SNP (single nucleotide polymorphism) генотипирования. История создания тагильской породы насчитывает более 200 лет. В настоящее время в России и в мире осталось единственное генофондное стадо тагильского скота (около 600 гол.), однако молекулярно-генетические характеристики этого ограниченного по численности генофонда остаются недостаточно исследованными. В настоящем сообщении впервые представлены результаты идентификации STR- и SNP-генотипов уникальной местной тагильской породы. Цель работы — микросателлитный анализ (STR) и полногеномный анализ полиморфизма единичных нуклеотидов (SNPs) для оценки генетического разнообразия и установления популяционной структуры современной популяции аборигенного тагильского скота. Генотипы животных тагильской породы (TAGIL, n = 98; СПК им. Шорохова, Пермский край, 2021 год) исследовали методом мультиплексного анализа по 11 микросателлитам (TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA122, INRA023, TGLA126, BM1818, ETH225, BM1824). Для межпородной дифференциации по STR-маркерам методом главных компонент (PCA) привлекали генотипы пород, которые потенциально могли принимать участие в формировании современной популяции тагильского скота (TAGIL), — голштинской (HLST), холмогорской голштинизированной (татарстанский тип) (TAT), холмогорской чистопородной (печорский тип) (PECH), черно-пестрой (старый тип) (Ch_P_OLD), тагильской (TAG) (образцы из банка данных ОНИС БиоТехЖ, 2020 год, https://www.vij.ru/2-obshchaya/226-infrastruktura-test). Для генотипирования TAGIL по SNP-маркерам на основании результатов анализа STR-генотипов отобрали наиболее неродственных животных (n = 48) для охвата максимального спектра генетического разнообразия. Полногеномное генотипирование по SNP-маркерам выполняли с использованием ДНК-чипа высокой плотности GGP Bovine HD 150K BeadChip (~ 150 тыс. SNP, «Illumina, Inc.», США) (108432 SNP до и 62809 SNP после LD-фильтрации). Для анализа результатов генотипирования SNP-маркеров (популяционно-генетическое и филогенетическое исследования) сформировали базу данных полногеномных SNP-генотипов тагильского скота (TAGIL). В качестве группы сравнения в набор данных (data set) ввели животных голштинской породы (HLST) (n = 45). Мы четко дифференцировали породы отобранных животных (тагильскую и голштинскую) с помощью метода PCA. Кластерный анализ на основании генетических дистанций FST объединил животных тагильской и голштинской пород в две большие группы в соответствии с породной принадлежностью. Полногеномное SNP-генотипирование выявило геномные области, в которых аллельные варианты специфичны для тагильской породы. Анализ hapFLK показал наличие пяти районов (p < 0,01) (4-я, 5-я, 8-я, 11-я и 15-я хромосомы, длина от 1,20 Mb на BTA8 до 9,61 Mb на BTA5, число SNPs внутри регионов — от 24 до 92), находящихся под давлением отбора в исследованных группах тагильского скота. По данным STR-маркирования установлено участие холмогорского скота, а также черно-пестрой и голштинской пород в формировании тагильской породы. Отмечена наибольшая интрогрессия голштинского скота, который в последние десятилетия, вероятнее всего, использовался в качестве улучшающей породы для тагильского крупного рогатого скота. Более чем у 50 % животных тагильской породы обнаружен островок ROH (BTA14, позиции 24437778-25098364, длина 0,661 Mb), ранее идентифицированный у ярославской и холмогорской пород в качестве региона, находящегося под давлением отбора. Не исключено, что этот ROH-регион может быть элементом адаптивной генетической системы у аборигенных пород. У 40 % животных тагильской породы дополнительно присутствуют пять островков ROH. Полученные нами данные будут использованы для выявления генов и их вариантов, определяющих адаптивные и хозяйственно значимые признаки тагильской породы, изучения истории формирования ее генетической структуры, разработки регламентов мониторинга для сохранения породной специфичности и биоразнообразия тагильского скота.
Ключевые слова: молочный скот, тагильская порода, микросателлиты, SNP-генотипирование, биоразнообразие.
Интенсификация мирового молочного скотоводства приводит к преобладанию в структуре пород крупного рогатого скота (КРС) голштинских коров. Cогласно статистике, голштинский скот (чистопородный или помесный) в совокупности составляет 61 % от 3,47 млн молочных коров Великобритании (1), в США доля КРС с голштинской породой в родословной еще больше — 90 % от общего числа дойных коров (2). В России поголовье голштинского скота пока еще не столь велико (22,95 %), но преобладает черно-пестрая порода (49,98 %) (3), в том числе частично голштинизированная. Разведение специализированных пород или нескольких внутрипородных линий снижает породное и генетическое разнообразие поголовья, и, как следствие, создается реальная угроза исчезновения аборигенного скота. В этой связи изучение популяционно-генетической структуры местных пород, обладающих уникальными адаптивными признаками и устойчивостью к ряду заболеваний, привлекает все большее внимание (4). Чаще всего для таких исследований используют микросателлитный анализ либо более информативный метод полногеномного SNP (single nucleotide polymorphism) генотипирования, который стал активно применяться в популяционной генетике и при разведении КРС после расшифровки его генома (5).
С помощью STR (short tandem repeat) маркеров изучали генетическую структуру популяций местных пород индийских зебу — онголе, деони, гир, канкрей (6), мексиканского скота криолло (7), а также красной степной (8), суксунской, истобенской, ярославской, холмогорской, серой украинской и холмогорской (печорский тип) пород КРС (9). Полногеномный SNP скрининг использовали для установления генетического разнообразия и межпородной дифференциации аборигенного южноафриканского скота (африканер, дракенсбергер, нгуни) (10), пяти местных пород КРС в Бангладеш (читагонгская красная, пабна и зебу муншигандж, северный бенгальский серый, деши) (11), шести пород скота из провинции Сычуань в Китае (башаньский, сюаньханьский, пингву, саньцзянский, ганзи, ляншаньский) (12), ирландской породы керри (13) и российских стародавних пород — бестужевской, холмогорской, костромской, красной горбатовской и ярославской (14, 15).
Тагильский скот — одна из старейших отечественных пород крупного рогатого скота молочного направления в Российской Федерации. История создания этой породы насчитывает более 200 лет. Формирование основной группы тагильского скота происходило в Уральском регионе в районе Нижнего Тагила. Точных сведений о возникновении тагильского скота нет, но обсуждалось участие в его формировании английского короткорогого и холмогорского скота, завезенного из Архангельской губернии в 1842 году (16). Высказывалось мнение, что тагильская порода — это продукт скрещивания местного уральского скота с завозимыми животными холмогорской, ярославской и голландской пород (17) и использования быков черно-пестрой породы (18). Плановая селекционно-племенная работа по улучшению тагильского скота началась после выхода декрета Совета Народных Комиссаров от 19 июля 1918 года «О племенном животноводстве». Тогда был применен жесткий отбор чистопородных тагильских быков-производителей с последующим разведением «в себе», что позволило существенно повысить продуктивность тагильских коров, и в 1930 году была утверждена новая тагильская порода КРС (18, 19). Неприхотливый к условиям кормления и содержания тагильский скот при разведении на Урале мало уступал по удою черно-пестрому, превосходя его по жирномолочности. К тому же тагильские коровы в силу специфического строения таза выделяются легкостью отела.
Впоследствии массовая голштинизация КРС привела к вытеснению не только тагильской, но и черно-пестрой породы. В результате в настоящее время осталось единственное в России и мире генофондное стадо тагильского скота (около 600 гол.), сформированное на базе СПК им. Шорохова в Октябрьском районе Пермского края. Этот ограниченный по численности генофонд представляет несомненный интерес как источник ценных биологических и хозяйственных признаков, однако его молекулярно-генетические характеристики остаются недостаточно исследованными.
В настоящем сообщении впервые представлены результаты идентификации STR- и SNP-генотипов уникальной местной тагильской породы КРС, что позволит выявлять гены и их варианты, которые определяют биологические, адаптивные и продуктивные качества животных, имеющие экономическое значение.
Цель выполненной работы — провести микросателлитный анализ (STR) и полногеномный анализ полиморфизма единичных нуклеотидов (SNPs) для оценки генетического разнообразия и установления популяционной структуры современной популяции аборигенного тагильского скота.
Методика. ДНК выделяли из образцов крови (n = 98) животных тагильской породы (TAGIL). Образцы отбирали в СПК им. Шорохова (Пермский край, 2021 год), для выделения ДНК использовали набор QIAmp ® DNA Mini Kit («QIAGEN», Германия) согласно прилагаемому протоколу. Определяли чистоту и концентрацию полученных препаратов ДНК (OD260/280, ультрафиолетовый микроспектрофотометр Implen Nano-Photometer®, «Implen GmbH», Германия), концентрацию двуцепочечной ДНК измеряли с помощью флуориметра Qubit™ (1.0) («Life Тechnologies», США).
Генотипы животных исследовали методом мультиплексного анализа по 11 микросателлитам (TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA122, INRA023, TGLA126, BM1818, ETH225, BM1824), используя STR-панель, разработанную в ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста на основе рекомендаций International Society for Animal Genetics (ISAG).
Межпородную дифференциацию по STR-маркерам проводили методом главных компонент (PCA) с привлечением генотипов пород КРС, которые потенциально могли принимать участие в формировании изучаемой современной популяции тагильского скота (TAGIL), — голштинской (HLST), холмогорской голштинизированной (татарстанский тип) (TAT), холмогорской чистопородной (печорский тип) (PECH), черно-пестрой (старый тип) (Ch_P_OLD), тагильской (TAG) (образцы из банка данных ОНИС БиоТехЖ, 2020 год, https://www.vij.ru/2-obshchaya/226-infrastruktura-test).
Для генотипирования по SNP-маркерам отобрали образцы (n = 48) на основании результатов анализа STR-генотипов. В программе Structure 2.3.4 (https://web.stanford.edu/group/pritchardlab/structure.html) по коэффициенту подобия Q предварительно оценивали чистопородность и в программе ML-Relate (https://www.montana.edu/kalinowski/software/ml-relate/index.html) — степень родства индивидуумов исследуемой популяции тагильского скота.
Полногеномное генотипирование по SNP-маркерам выполняли с использованием ДНК-чипа высокой плотности GGP Bovine HD 150K BeadChip (~ 150 тыс. SNP, «Illumina, Inc.», США). Контроль качества и фильтрацию данных генотипирования для каждого образца и SNP выполняли с использованием программного пакета PLINK 1.9 (20) (https://www.co-gen-omics.org/plink/). Были применены следующие фильтры (в скобках даны со-ответствующие команды в программе PLINK): сall-rate по всем исследуемым SNP для индивидуального образца не ниже 90 % (--mind); call-rate для каждого из исследованных SNP по всем генотипированным образцам не ниже 90 % (--geno); частота встречаемости минорных аллелей (MAF) более 0,01 или 0,05 (--maf 0.01); отклонение SNP генотипов от распределения Харди-Вайнберга в совокупности протестированных образцов с достоверностью p-value < 10-6 (--hwe). Также оценивали неравновесие по сцеплению (LD, linkage disequilibrium) с помощью коэффициента корреляции Пирсона (r2 < 0,2) с шагом 50 kb (--indep-pairwaise).
Для анализа результатов генотипирования SNP-маркеров (популяционно-генетическое и филогенетическое исследования) сформировали базу данных полногеномных SNP-генотипов тагильского скота (TAGIL). В качестве группы сравнения в набор данных (data set) ввели животных голштинской породы (HLST) (n = 45).
На основании полученных SNP-генотипов для каждого из набора данных в R пакете diveRsity (21) расcчитывали наблюдаемую гетерозиготность (Ho), несмещенную ожидаемую гетерозиготность (uHe), аллельное разнообразие (Аr) и коэффициент инбридинга FIS (c 95 % доверительным интервалом).
Анализ главных компонент (PCA) выполняли в программе PLINK 1.9 с последующим построением графика в R пакете ggplot2 (22). Для филогенетических исследований в R пакете diveRsity (21) рассчитывали попарные значения FST (23). Матрицу попарных значений FST визуализировали в виде групповой генетической сети Neighbor-net в программе SplitsTree 4.14.5 (24) (https://splitstree4.software.informer.com/). Популяционную структуру и генетическую однородность изучаемых пород КРС устанавливали в программе Admixture 1.3 (25) с графическим представлением с помощью R пакета pophelper (26). Наиболее вероятное число предковых кластеров (К) определяли с помощью расчета значений ошибки кросс-валидации (CV error) для K от 1 до 5 в программе Admixture 1.3.
Для поиска локусов, находящихся под давлением отбора, использовали отбор 0,1 % SNPs, имеющих наибольшие значения FST при парном сравнении пород, а также анализ hapFLK и выявление островков ROH, перекрывающихся у некоторых индивидуумов (15). Для островков минимальный размер ROH принимали равным 0,5 Mb при 50 % животных, несущих перекрывающиеся ROH, и длине перекрывания ROH не менее 0,1 Mb.
Биоинформатическую обработку данных и построение графиков проводили с помощью программной среды R Project for Statistical Com-puting (27).
При hapFLK анализе был установлен порог достоверности p < 0,01. Для значений FIS приведены доверительные интервалы (CI, 95 %). При определении средних значений показателей (M) рассчитывали их стандартные ошибки (±SEM).
Результаты. Результаты анализа главных компонент (PCA) (рис. 1) были получены по данным проведенного нами микросателлитного профилирования животных тагильской породы в сравнении с данными выполненного ранее STR-генотипирования популяций голштинской, холмогорской (голштинизированной и чистопородной), черно-пестрой и тагильской пород КРС. Можно видеть, что значительная часть коров тагильской породы отличается от животных других пород, но массивы обеих популяций тагильского скота перекрываются с голштинской породой. На этом основании мы в дальнейшем рассматривали голштинскую породу в качестве группы сравнения.
Для охвата максимального спектра генетического разнообразия тагильской породы для SNP-генотипирования отбирали наиболее неродственных животных. На основе расчета коэффициента подобия (Q) и оценки степени родства (родственниками считали особей с Q ≥ 0,35) отобрали 51 животное (48 основных и 3 запасных). Отобранных особей разделили на пять групп: 1-я — Q ≥ 90 %, родственников не > 2 у каждого животного (хотя есть близкие с Q = 0,50; 37 коров); 2-я — 90 % > Q ≥ 80 %, родственников не > 2 у каждого животного (хотя есть близкие с Q = 0,50; 7 коров); 3-я — 80 % > Q ≥ 70 %, у каждого животного > 1 родственника (нет ближе, чем с Q = 0,49; 5 коров); 4-я — 70 % > Q ≥ 60 %, нет родственников (2 коровы); 5-я (прочие) — нежелательны для генотипирования.
Полногеномное генотипирование тагильского скота по SNP-маркерам проводили с использованием ДНК-чипа GGP Bovine HD 150K BeadChip, результаты получили для 48 животных (результативность генотипирования составила более 90 %). Эффективность генотипирования (call rate) исследуемых коров тагильской породы варьировала от 0,9900 до 0,9982. Всего после контроля качества для анализа отобрали 108432 SNP.
Таблица 1 обобщает данные проведенного нами популяционно-генетического исследования тагильской породы по SNP-маркерам в сравнении с голштинской. Можно видеть достоверно более высокое генетическое разнообразие (по показателям аллельного разнообразия Ar, наблюдаемой гетерозиготности Ho и несмещенной ожидаемой гетерозиготности uHe) тагильского скота по сравнению с голштинским. Это может быть следствием как менее жесткого отбора по хозяйственно полезным признакам, так и участия нескольких пород в создании тагильского скота. Следует также отметить избыток гетерозигот в обеих популяциях (см. табл. 1).
Мы четко дифференцировали породы отобранных животных (тагильскую и голштинскую) с помощью метода PCA (рис. 2). При этом в отличие от тагильской голштинская порода оказалась более консолидированной (см. рис. 2), первая компонента (PC1) отвечала за 7,28 % генотипической изменчивости и отделяла тагильский скот от голштинского.
Кластерный анализ на основании генетических дистанций FST (рис. 3) объединил животных тагильской и голштинской пород в две большие группы в соответствии с породной принадлежностью. Животные, относящиеся к одной и той же породе, группировались на соседних ветвях соответствующих кластеров.
При проведении структурного анализа расчет ошибки кросс-вали-дации (CV) показал минимальное значение этого показателя для числа кластеров K = 4. При K = 2 (рис. 4) каждая из двух сравниваемых пород (TАGIL и HLST) проявляет специфическую кластерную структуру, при этом в формировании тагильской породы прослеживается наличие специфических голштинских предковых геномных компонентов. Анализ генетической структуры при K = 3 и K = 4 указывает на участие в формировании тагильской породы еще трех различных предковых пород дополнительно к голштинской. Учитывая, что специфические голштинские геномные компоненты проявляются у большей части животных тагильской породы, а компоненты трех других предковых пород тагильского скота имеют лишь незначительные следы адмиксии в голштинской породе, полученный набор данных можно считать пригодным для поиска локусов, находящихся под давлением отбора.
При выборе 0,1 % SNPs, имеющих наибольшие значения FST при парном сравнении пород, идентифицировали 109 SNPs, локализованных на 24 хромосомах (за исключением BTA8, BTA23, BTA27, BTA28 и BTA29).
Анализ hapFLK показал наличие пяти районов (p < 0,01), находящихся под давлением отбора в исследованных группах тагильского скота (табл. 2) в сопоставлении с голштинским. Выявленные области локализовались на 4-й, 5-й, 8-й, 11-й и 15-й хромосомах, при этом для двух регионов был установлен более высокий уровень достоверности идентификации (p < 0,001). Длина регионов варьировала от 1,20 Mb (BTA8) до 9,61 Mb (BTA5), число SNPs, локализованных внутри указанных регионов, изменялось от 24 до 92.
В результате исследования геномов тагильского и голштинского скота на наличие участков гомозиготности (runs of homozygosity — RОН) мы выявили 37 островков ROH, которые встречались более чем у 50 % животных, причем 36 таких регионов было обнаружено в геноме голштинского скота, и лишь один — у тагильского (BTA14, позиции 24437778-25098364, длина 0,661 Mb). Интересно, что этот регион практически полностью перекрывался с идентичным регионом, идентифицированным в геноме голштинского скота (позиции 24437778-25175950, длина 0,750 Mb). Следует отметить, что регион на BTA14 в области 24,4-25,1 сМ ранее был идентифицирован как находящийся под давлением отбора у ярославской и холмогорской пород скота (15). Проведенная структурная аннотация выявила локализацию в этой области восьми генов — XKR4,TMEM68, TGS1,LYN,RPS20,MOS,PLAG1 и CHCHD7. Ранее в исследованиях на разных породах скота (голштинская, симментальская, вагю, хану и др.) было показано, что перечисленные гены ассоциированы с ростом, экстерьерной высокорослостью, живой массой и потреблением корма (28-33).
Исходя из того, что при совершенствовании тагильского скота его скрещивали не только с голштинской, но и с другими черно-пестрыми породами крупного рогатого скота (холмогорская и черно-пестрая), мы снизили до 40 % порог доли животных тагильской породы, в геноме которых встречаются общие ROH. Это позволило дополнительно идентифицировать у тагильской породы пять островков ROH (табл. 3).
Таким образом, в результате проведенных исследований дана характеристика популяционно-генетических особенностей современного тагильского скота и создана база полногеномных SNP-генотипов, отвечающая установленным критериям качества (число генотипированных животных с результативностью генотипирования более 90 %). По данным STR-марки-рования установлено участие холмогорского скота, а также черно-пестрой и голштинской пород в формировании тагильской породы. Отмечена наибольшая интрогрессия голштинского скота, который в последние десятилетия, вероятнее всего, использовался в качестве улучшающей породы для тагильского крупного рогатого скота. В анализируемой популяции животные тагильской породы дифференцируются от голштинского скота и представляют собой генотипически менее консолидированную и менее структурированную группу. В отличие от голштинского скота тагильский характеризовался достоверно (р < 0,05) более высоким генетическим разнообразием и избытком гетерозигот. Полногеномное SNP-генотипирование выявило геномные области, в которых аллельные варианты специфичны для тагильской породы. При сравнении тагильской и голштинской пород на 24 хромосомах идентифицированы 109 SNPs с наибольшими значениями FST при парном сравнении, выявлены пять регионов, находящихся под давлением отбора (p < 0,01) у тагильского и голштинского скота, которые локализованы на 4-й, 5-й, 8-й, 11-й и 15-й хромосомах. Более чем у 50 % жи-вотных тагильской породы обнаружен островок ROH (BTA14, позиции 24437778-25098364, длина 0,661 Mb), ранее идентифицированный у ярославской и холмогорской пород в качестве региона, находящегося под давлением отбора. Не исключено, что этот ROH-регион может быть элементом адаптивной генетической системы у аборигенных пород. У 40 % животных тагильской породы дополнительно присутствуют пять островков ROH. Полученные нами данные будут использованы для выявления генов и их вариантов, определяющих адаптивные и хозяйственно значимые признаки тагильской породы, изучения истории формирования ее генетической структуры, разработки регламентов мониторинга для сохранения породной специфичности и биоразнообразия тагильского скота.
1. Сравнительная характеристика генетического разнообразия популяции тагильского скота |
|||||
Порода |
n |
Ar |
Ho |
uHe |
FIS [CI 95 %] |
Тагильская |
48 |
1,999±0 |
0,408±0,001 |
0,398±0,000 |
-0,023 [-0,024; -0,022] |
Голштинская |
45 |
1,989±0 |
0,366±0,001 |
0,360±0,001 |
-0,014 [-0,015; -0,013] |
2. Характеристика участков хромосом (BTA), находящихся под давлением отбора у тагильского скота (TAGIL, n = 48, анализ методом hapFLK, СПК им. Шорохова, Пермский край, 2021 год) |
||||||
BTA |
Позиция |
Длина, Mb |
Наиболее значимый SNPs |
Число SNPs |
||
начальная |
конечная |
позиция |
p |
|||
4-я |
6 842 949 |
10 648 384 |
3,81 |
8 495 236 |
7,08E-04 |
63 |
5-я |
16 952 114 |
26 561 662 |
9,61 |
24 064 770 |
8,42E-04 |
92 |
8-я |
50 695 489 |
51 899 568 |
1,20 |
51 101 318 |
5,14E-03 |
24 |
11-я |
86 926 025 |
89 081 028 |
2,16 |
88 186 796 |
6,16E-03 |
31 |
15-я |
48 905 274 |
54 898 376 |
5,99 |
54 218 907 |
5,08E-03 |
59 |
3. Характеристика островков ROH, идентифицированных в геноме тагильского скота |
||||||
Порода |
BTA |
Число SNP |
Позиция |
Длина, Mb |
Доля животных, % |
|
начальная |
конечная |
|||||
TAGIL |
2-я |
15 |
65 513 882 |
65 946 493 |
0,433 |
41,7 |
TAGIL |
14-я |
15 |
33 026 716 |
33 348 218 |
0,322 |
41,7 |
TAGIL |
16-я |
4 |
44 372 045 |
44 552 678 |
0,181 |
41,7 |
TAGIL |
20-я |
17 |
71 433 871 |
71 720 853 |
0,287 |
41,7 |
TAGIL |
23-я |
18 |
479 600 |
936 645 |
0,457 |
41,7 |
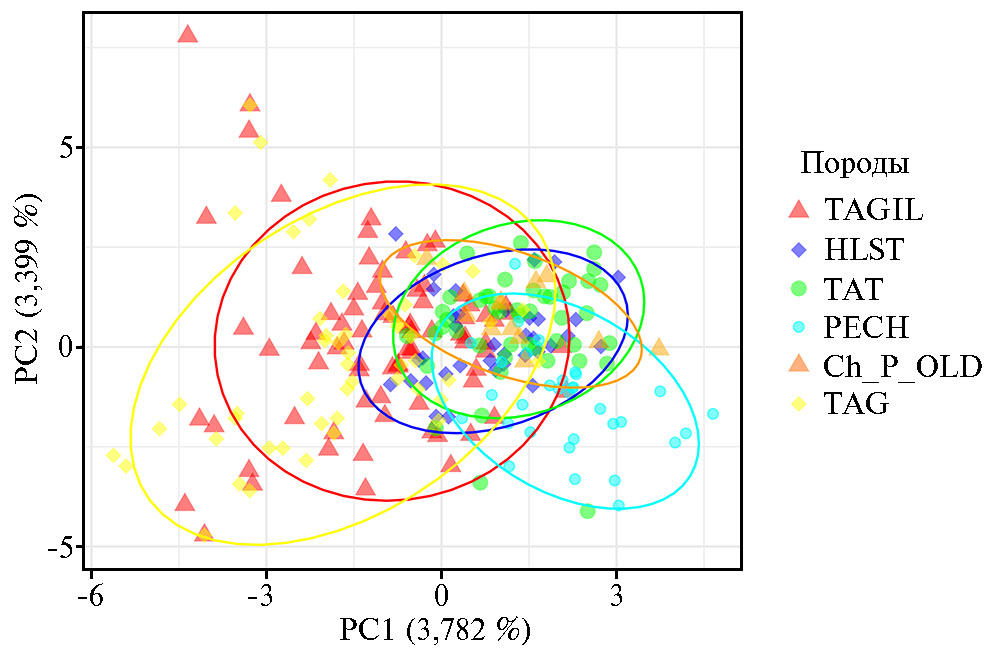
Рис. 1. Распределение особей тагильской породы на основании генотипов по STR-маркерам при анализе методом главных компонент: современная популяция тагильского скота (TAGIL, n = 98) (СПК им. Шорохова, Пермский край, 2021 год), голштинская по-рода (HLST), холмогорская гол-штинизированная порода (татарстанский тип) (TAT), холмогорский чистопородный скот (печорский тип) (PECH), черно-пестрая порода (старый тип) (Ch_P_OLD), тагильский скот (TAG) (образцы из банка данных ОНИС БиоТехЖ, 2020 год).
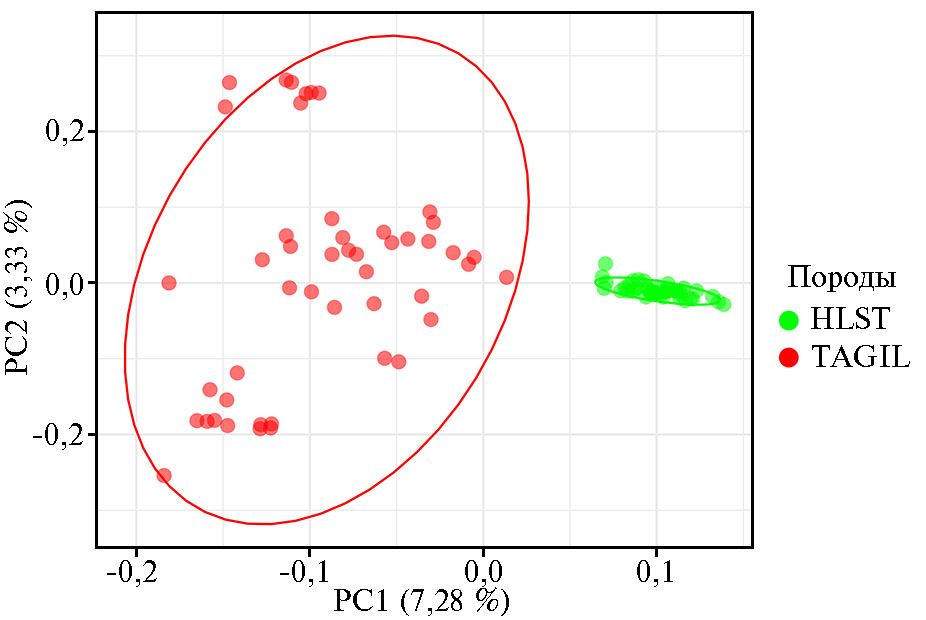
Рис. 2. Распределение особей тагильской (TAGIL, n = 48) (СПК им. Шорохова, Пермский край, 2021 год) и голштинской пород (HLST, банк данных ОНИС БиоТехЖ, 2020 год) на основании генотипов по 108432 SNPs.
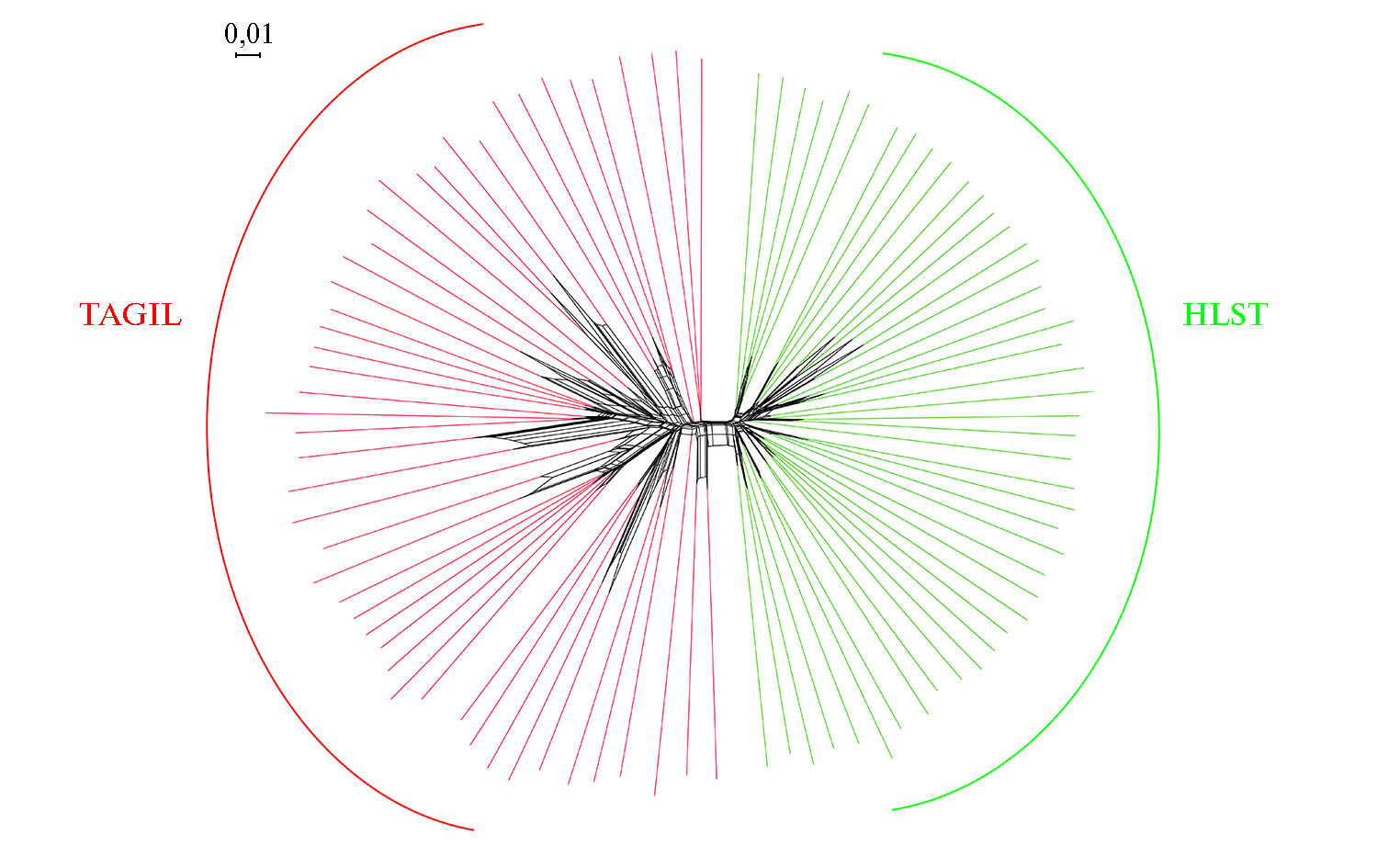
Рис. 3. Дендрограмма Neighbor-net, построенная на основании генотипов по 108432 SNPs для отобранных животных тагильской (TAGIL, n = 48) (СПК им. Шорохова, Пермский край, 2021 год) и голштинской пород (HLST, n = 45, банк данных ОНИС БиоТехЖ, 2020 год) (визуализация в программе SplitsTree 4.14.5).
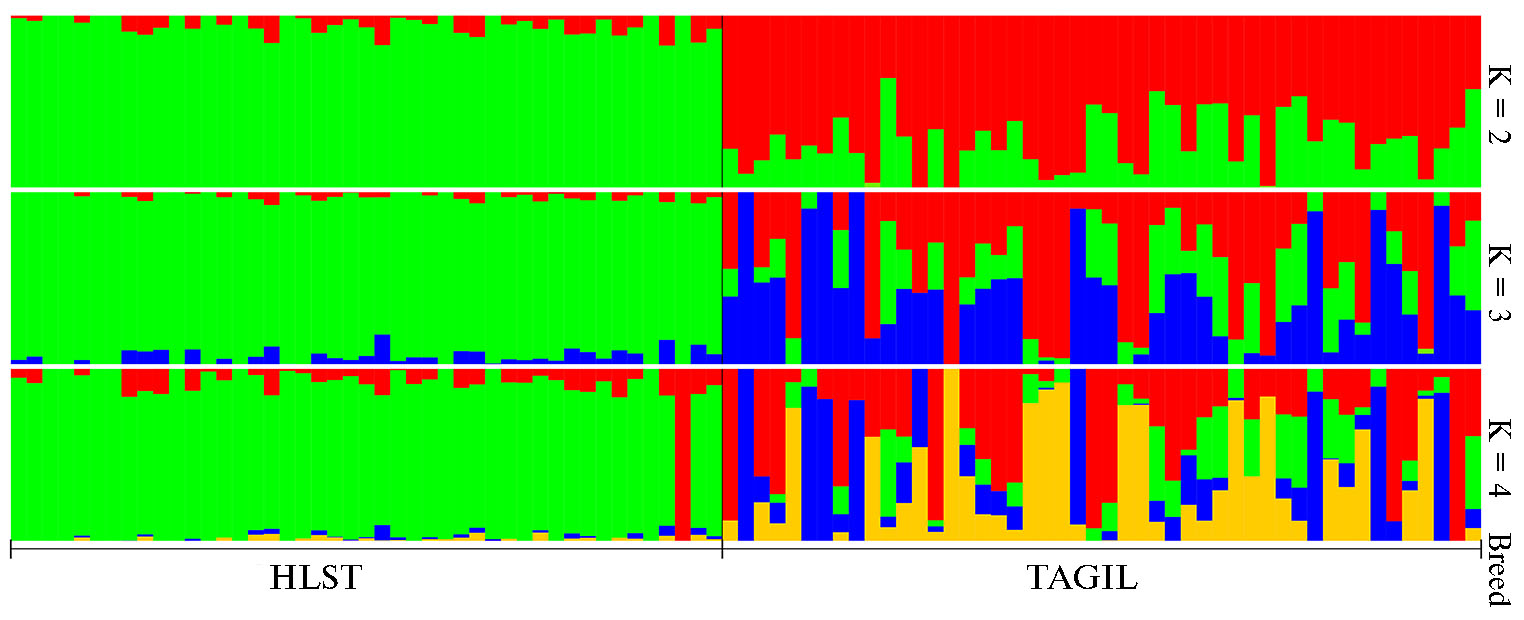
Рис. 4. Генетическая структура популяции тагильской породы крупного рогатого скота (TAGIL, n = 48, СПК им. Шорохова, Пермский край, 2021 год) по сравнению с голштинской (HLST, n = 45, банк данных ОНИС БиоТехЖ, 2020 год) по результатам анализа Admixture с использованием 62809 SNPs (программа Admixture 1.3, K — число кластеров). Синий и желтый цвета — геномные компоненты предковых пород, выявленные в тагильской породе.
ЛИТЕРАТУРА
- Most common of cattle in GB (NUTS 1 areas) in 01 April 2005. London, 2005, Version 1.
- Holstein Association USA. Режим доступа: http://holsteinusa.com. Без даты.
- Ежегодник по племенной работе в молочном скотоводстве в хозяйствах Российской Федерации (2019 г.). М., 2020.
- Mwai О., Hanotte О., Kwon Y.-J., Cho S. African indigenous cattle: unique genetic resources in a rapidly changing world. Asian Australas. J. Anim. Sci., 2015, 28(7): 911-921 (doi: 10.5713/ajas.15.0002R)
- Elsik C.G., Bovine Genome Sequencing and Analysis Consortium. The genome sequence of taurine cattle: a window to ruminant biology and evolution. Science, 2009, 324(5926): 522-528 (doi: 10.1126/science.1169588).
- Kale D.S., Rank D.N., Joshi C.G., Yadav B.R., Koringa P.G., Thakkar K.M., Tolenkhomba T.C., Solanki J.V. Genetic diversity among Indian Gir, Deoni and Kankrej cattle breeds based on microsatellite markers. Indian Journal of Biotechnology, 2010, 9: 126-130.
- Ulloa-Arvizu R., Gayosso-Vazquez A., Ramos-Kuri M., Estrada F.J., Montano M., Alonso R.A. Genetic analysis of Mexican Criollo cattle populations. Journal of Animal Breeding and Genetics, 2008, 125: 351-359 (doi: 10.1111/j.1439-0388.2008.00735.x).
- Kramarenko A.S., Gladyr E.A., Kramarenko S.S., Pidpala T.V., Strikha L.A., Zinovieva N.A. Genetic diversity and bottleneck analysis of the Red Steppe cattle based on microsatellite markers. Ukrainian Journal of Ecology, 2018, 8(2): 12-17 (doi: 10.15421/2018_303).
- Kiseleva T.Yu., Kantanen J., Vorobyov N.I., Podoba B.E., Terletsky V.P. Linkage disequilibrium analysis for microsatellite loci in six cattle breeds. Russian Journal of Genetics, 2014, 50(4): 406-414 (doi: 10.1134/S1022795414040048).
- Zwane А.А., Schnabel R.D., Hoff J., Choudhury A., Makgahlela M.L., Maiwashe A., Van Marle-Koster E., Taylor J.T. Genome-Wide SNP discovery in indigenous cattle breeds of South Africa. Front. Genet., 2019, 10(273): 1-16 (doi: 10.3389/fgene.2019.00273).
- Bhuiyan M.S.A., Lee S.-H., Hossain S.M.J., Deb G.K., Afroz M.F., Lee, S.-H., Bhuiyan A.K.F.H. Unraveling the genetic diversity and population structure of Bangladeshi indigenous cattle populations using 50K SNP markers. Animals, 2021, 11(2381): 1-15 (doi: 10.3390/ani1108238).
- Wang W., Gan J., Fang D., Tang H., Wang H., Yi J., Fu M. Genome-wide SNP discovery and evaluation of genetic diversity among six Chinese indigenous cattle breeds in Sichuan. PLoS ONE, 2018, 13(8): e0201534 (doi: 10.1371/journal.pone.0201534).
- Browett S., McHugo G., Richardson I.W., Magee D.A., Park S.D.E., Fahey A.G., Kearney J.F., Correia C.N., Randhawa I.A.S., MacHugh D.E. Genomic characterisation of the indigenous Irish Kerry Cattle breed. Front. Genet., 2018, 9(51): 1-17 (doi: 10.3389/fgene.2018.00051).
- Зиновьева Н.А., Доцев А.В., Сермягин А.А., Виммерс К., Рейер Х., Солкнер Й., Денискова Т.Е., Брем Г. Изучение генетического разнообразия и популяционной структуры российских пород крупного рогатого скота с использованием полногемного анализа SNP. Сельскохозяйственная биология, 2016, 51(6): 788-800 (doi: 10.15389/agrobiology.2016.6.788rus).
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Deniskova T.E., Abdelmanova A.S., Kharzinova V.R., Sölkner J., Reyer H., Wimmers K., Brem G. Selection signatures in two oldest Russian native cattle breeds revealed using high-density single nucleotide polymorphism analysis. PLoS ONE, 2020, 15(11): e0242200 (doi: 10.1371/journal.pone.0242200).
- Романов А.И. О Тагильском скоте в связи с его историей и учреждением земского рассадника на основах метизации с голландским скотом. В сб.: Труды совещания вет. врачей и представителей земств. Пермь, 1913: 11-129.
- Пути и формы создания и сохранения генофонда ценных локальных пород. Л., 1979.
- Кремер Л.А. Тагильский скот. М.-Л, 1931.
- Моисеева И.Г., Уханов С.В., Столповский Ю.А. и др. Генофонды сельскохозяйственных животных: генетические ресурсы животноводства России /Под ред. И.А. Захарова. М., 2006.
- Plink: Whole genome association analysis toolset. Режим доступа: http://zzz.bwh.harvard.edu/plink/. Дата обращения: 25.01.2017.
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodöhl P.A. diveRsity: an R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4(8): 782-788 (doi: 10.1111/2041-210X.12067).
- Wickham H. ggplot2: elegant graphics for data analysis. N.-Y., 2009.
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 (doi: 10.2307/2408641).
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 (doi: 10.1093/molbev/msj030).
- Alexander D.H., Novembre J., Lange K. Fast model-based estimation of ancestry in unrelated individuals. Genome Research, 2009, 19(9): 1655-1664 (doi: 10.1101/gr.094052.109).
- Francis R.M. pophelper: An R package and web app to analyse and visualise population structure. Molecular Ecology Resources, 2017, 17(1): 27-32 (doi: 10.1111/1755-0998.12509).
- R Core Team. R: a language and environment for statistical computing. R foundation for statistical computing, Vienna, 2018. Режим домтупа: https://www.R-project.org/. Без даты.
- Lindholm-Perry A.K., Kuehn L.A., Smith T.P., Ferrell C.L., Jenkins T.G., Freetly H.C., Snelling W.M. A region on BTA14 that includes the positional candidate genes LYPLA1, XKR4 AND TMEM68 is associated with feed intake and growth phenotypes in cattle. Anim Genet., 2012, 43: 216-219 (doi: 10.1111/j.1365-2052.2011.02232.x).
- Pryce J.E., Arias J., Bowman P.J., Davis S.R., Macdonald K.A., Waghorn G.C., Wales W.J., Williams Y.J., Spelman R.J., Hayes B.J.Accuracy of genomic predictions of residual feed intake and 250-day body weight in growing heifers using 625,000 single nucleotide polymorphism markers. J. Dairy Sci., 2012, 95(4): 2108-2119 (doi: 10.3168/jds.2011-4628).
- Taye M., Yoon J., Dessie T., Cho S., Oh S.J., Lee H.K., Kim H. Deciphering signature of selection affecting beef quality traits in Angus cattle. Genes Genomics, 2018, 40(1): 63-75 (doi: 10.1007/s13258-017-0610-z).
- Cheruiyot E.K., Bett R.C., Amimo J.O., Zhang Y., Mrode R., Mujibi F.D.N. Signatures of selection in admixed dairy cattle in Tanzania. Front. Genet., 2018, 9(607): 1-15 (doi: 10.3389/fgene.2018.00607).
- De Las Heras-Saldana S., Clark S.A., Duijvesteijn N., Gondro C., van der Werf J.H.J., Chen Y. Combining information from genome-wide association and multi-tissue gene expression studies to elucidate factors underlying genetic variation for residual feed intake in Australian Angus cattle. BMC Genomics, 2019, 20(1): 939 (doi: 10.1186/s12864-019-6270-4).
- Grigoletto L., Ferraz J.B.S., Oliveira H.R., Eler J.P., Bussiman F.O., Abreu Silva B.C., Baldi F., Brito L.F. Genetic architecture of carcass and meat quality traits in Montana tropical composite beef cattle. Front. Genet., 2020, 11(123): 1-13 (doi: 10.3389/fgene.2020.00123).