doi: 10.15389/agrobiology.2021.6.1111rus
УДК 636.2:577.2
Исследования выполнены при поддержке Российского научного фонда, проект № 21-66-00007. При проведении исследований было использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных».
ПОЛНОГЕНОМНОЕ ИССЛЕДОВАНИЕ АССОЦИАЦИЙ SNP С ВЫСОТОЙ В ХОЛКЕ В ПОПУЛЯЦИЯХ ЛОКАЛЬНЫХ И ТРАНСГРАНИЧНЫХ ПОРОД КРУПНОГО РОГАТОГО СКОТА В РОССИИ
А.С. АБДЕЛЬМАНОВА ✉, М.С. ФОРНАРА, Н.В. БАРДУКОВ, А.А. СЕРМЯГИН, А.В. ДОЦЕВ, Н.А. ЗИНОВЬЕВА
Рост животного — это классический количественный признак, который влияет на предрасположенность к определенным заболеваниям и связан с продуктивностью сельскохозяйственных животных. Картировано множество локусов количественных признаков (quantitative trait loci, QTLs), влияющих на составляющие роста крупного рогатого скота (КРС), что подтверждает его полигенный детерминизм. В настоящей работе в российских локальных породах установлено преобладание аллелей, связанных с высоким ростом в холке, в трех из четырех выявленных однонуклеотидных полиморфизмов (single nucleotide polymorphisms, SNP). Целью работы была идентификация локусов и характеристика аллельных вариантов, находящихся под давлением отбора и связанных с размерами тела, в популяциях российских локальных и разводимых на территории Российской Федерации трансграничных пород крупного рогатого скота различных направлений продуктивности, по-разному подверженных давлению искусственного отбора и неодинаково распространенных в мире. Исследовали биоматериал (цельную кровь, сперму и ушные выщипы, хранящиеся в УНУ «Банк генетического материала домашних и диких видов животных и птицы» ФГБНУ ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста) от 670 гол. крупного рогатого скота 13 пород: абердин-ангусской (n = 39), айрширской (n = 144), черно-пестрой (n = 50), голштинской (n = 184), истобенской (n = 22), джерсейской (n = 32), калмыцкой (n = 27), холмогорской (n = 26), киргизской (n = 24), монгольской (n = 26), тагильской (n = 26), якутской (n= 29) и ярославской (n = 41). Генотипирование проводилось на SNP-чипах разной плотности, GGP Bovine 150K и BovineHD BeadChip («Illumina, Inc.», США), в ходе обработки данных были определены общие для двух чипов SNP, которые использовались для дальнейшего анализа. C помощью программы PLINK 1.9 с использованием фильтров (--geno 0.1), (--mind 0.2), (--maf 0.05) было проведено полногеномное исследование ассоциаций данных генотипирования с признаками физического развития животных. Данные о высоте в холке для исследуемых пород получили из базы данных ФАО. Породы разделили на группы по следующим критериям: в соответствии с ростом (высокорослые, низкорослые), направлением продуктивности (молочные, мясные), степенью давления селекции (примитивные, заводские) и распространенностью в мире (российские, трансграничные). В общей сложности выявили четыре SNP, из которых три были локализованы на 4-й хромосоме (ARS-BFGL-NGS-116590, Hapmap53144-ss46525999, BovineHD0400021479), один — на 14-й хромосоме (BovineHD1400007259). Альтернативные аллели в обнаруженных SNP статистически значимо различаются по частоте встречаемости в разных группах пород животных, а также имеют статистически значимые положительные либо отрицательные корреляции с высотой в холке. Многообразие и разнородность пород, представленных в выборке, позволяют рассматривать выявленные следы селекции как характерные не для одной породы, региона или направления продуктивности, а для группы пород вида Bos taurus taurus, распространение которых от центра одомашнивания шло по Дунайскому пути. Таким образом, выявленные SNP могут использоваться в качестве генетических маркеров в программах селекции на увеличение роста животных и повышение их продуктивности.
Ключевые слова: Bostaurus, крупный рогатый скот, локальные породы, трансграничные породы, QTL, SNP-маркеры, ДНК-чипы, GWAS, PLAG1, высота в холке.
Племенная работа по совершенствованию крупного рогатого скота (КРС) имеет важное значение для повышения эффективности скотоводства, поскольку гармонично сложенные, развитые животные имеют высокую продуктивность и пользуются спросом на рынке племенной продукции (1).
Полногеномный анализ ассоциаций (whole-genome associated study, GWAS) позволил идентифицировать множество геномных вариаций, связанных с количественными признаками сельскохозяйственных животных (2). В последние несколько десятилетий проведены исследования по картированию локусов количественных признаков (quantitative trait loci, QTLs), ответственных за молочную продуктивность (3), устойчивость к болезням (4, 5), воспроизводительные качества (6), рост (7-9), мясные качества (10-13) и качества туши КРС (14-16).
Рост животного — это классический количественный признак, привлекающий внимание генетиков на протяжении долгого времени (17-19). Понимание молекулярных основ вариабельности роста между индивидуумами поможет не только получить информацию об общей архитектуре количественных признаков, но также даст представление о механизмах, контролирующих рост организма. Известно, что рост влияет на предрасположенность к определенным заболеваниям и связан с продуктивностью сельскохозяйственных животных. Несмотря на чувствительность к факторам окружающей среды, он обычно передается по наследству, в том числе у человека (~ 85 %) (20).
Доместикация КРС оказала сильное влияние на этот показатель. Рост у вида Bos taurus уменьшился примерно в 1,5 раза от Неолита до Средневековья и снова увеличился только в Раннем Новом времени (21). Вымерший тур (Bos primigenius) был намного крупнее своих домашних потомков (высота в холке ~ 2 м против ~ 1,1-1,5 м у современного КРС). Коэффициент наследуемости показателя колеблется в пределах ~ 25-85 % в зависимости от популяции (22, 23). Если механизмы генетической изменчивости способствовали восстановлению роста с Раннего Нового времени, их отпечатки селекции должны быть обнаружены на основе геномных данных современных пород КРС.
В настоящее время картировано множество QTLs, влияющих на составляющие роста КРС, что подтверждает его полигенный детерминизм. Например, сообщалось о картировании 38 и 52 QTLs, объясняющих более 60 % вариаций соответственно роста и массы взрослых особей в американской популяции коров абердин-ангусской породы (24). L. Karim с соавт. (25) исследовали животных голштино-фризской и джерсейской пород для идентификации QTLs, влияющих на признаки, важные для молочного скотоводства. Были проведены измерения для более чем 500 признаков, шесть из которых относились к размерам тела: масса при рождении, масса в 6, 8, 12, 18 и более 24 мес (живая масса), а также высота в холке в 18 мес.
I. Randhawa с соавт. (26) сравнили данные однонуклеотидного полиморфизма (single nucleotide polymorphism, SNP) по всему геному у ряда европейских и африканских пород B. taurus с высокой (145-155 см) и низкой (105-133 см) холкой и обнаружили, что наиболее значимый селекционный сигнал, связанный с ростом, картирован на 14-й хромосоме (BTA14) в регионе 24,79-28,25 Mbp (27). Этот отпечаток селекции был недавно подтвержден данными последовательностей полного генома четырех пород B. taurus и ограничен меньшей областью, охватывающей позиции 24,80-25,08 Mbp (28), где расположен ген плеоморфной аденомы 1 (PLAG1).
Ген PLAG1, состоящий из пяти экзонов и четырех интронов и имеющий пять транскриптов, был впервые обнаружен посредством позиционного картирования, когда исследователи изучали плеоморфные аденомы слюнных желез у человека (29).PLAG1 инициирует транскрипцию инсулиноподобного фактора роста 2 (IGF2), митогенного гормона, важного для роста и развития плода, и участвует в генетической изменчивости роста как у человека, так и у КРС (25, 30-32). Опубликовано значительное число работ, в которых исследовалась связь гена PLAG1 с легкостью отела (33), размерами тела (25), живой массой в раннем возрасте (14), характеристиками туши (34), массой при рождении (35) и другими признаками КРС (36). Российские локальные породы представляют большой интерес в качестве резерва биоразнообразия (37, 38), однако остаются неизученными в отношении гена PLAG1 несмотря на проведенные ранее полногеномные исследования (39, 40).
В настоящей работе впервые проведена оценка частоты встречаемости аллелей SNP, связанных с высотой в холке, в популяциях крупного рогатого скота, разводимых на территории России. В российских локальных породах установлено преобладание таких аллелей в трех из четырех выявленных однонуклеотидных полиморфизмов.
Целью работы была идентификация локусов и характеристика аллельных вариантов, находящихся под давлением отбора и связанных с размерами тела, в популяциях российских локальных и разводимых на территории Российской Федерации трансграничных пород крупного рогатого скота различных направлений продуктивности, по-разному подверженных давлению искусственного отбора и неодинаково распространенных в мире.
Методика. Исследовали биоматериал (цельную кровь, сперму и ушные выщипы, хранящиеся в УНУ «Банк генетического материала домашних и диких видов животных и птицы» ФГБНУ ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста) от 670 гол. крупного рогатого скота 13 пород: абердин-ангусской (ANG, n = 39), айрширской (AYR,n = 144), черно-пестрой (BLW, n = 50), голштинской (HLS, n = 184), истобенской (IST, n = 22), джерсейской (JRS, n = 32), калмыцкой (KLM, n = 27), холмогорской (KHL, n = 26), киргизской (KRG, n = 24), монгольской (MNG, n = 26), тагильской (TAG, n = 26), якутской (YKT, n= 29) и ярославской (YRS, n = 41).
ДНК выделяли с помощью коммерческих наборов ДНК-Экстран-1 и ДНК-Экстран-2 (ООО «Синтол», Россия) в соответствии с рекомендациями производителя. Перед генотипированием на SNP-чипах полученные препараты проходили контроль качества: измеряли концентрацию (не менее 15 нг/мкл) двуцепочечной ДНК (флуориметр Qubit 4.0, «Invitrogen, Life Technologies», США), а также соотношение OD260/OD280 (допустимые значения 1,8-2,0), характеризующее чистоту препаратов (спектрофотометр NanоDropÔ 8000 («Thermo Fisher Scientific, Inc.», США).
Образцы 11 пород генотипировали самостоятельно, генотипы двух пород — абердин-ангусской и джерсейской — были загружены из базы данных WIDDE (http://widde.toulouse.inra.fr/widde/widde/main.do?module=cattle).
Поскольку генотипирование образцов проводили на SNP-чипах разной плотности — GGP Bovine 150K и BovineHD BeadChip («Illumina, Inc.», США), в процессе обработки данных были определены общие для двух чипов SNP, которые использовались для дальнейшего анализа. Качество генотипирования контролировали в программе PLINK 1.9 (41) с использованием следующих фильтров: успешно генотипированы не менее 90 % локусов (--geno 0.1) не менее чем у 80 % животных (--mind 0.2), частота минорных аллелей не менее 5 % (--maf 0.05). После объединения наборов данных и контроля качества генотипирования для анализа отобрали 115237 SNP. C помощью PLINK 1.9 осуществляли полногеномное исследование ассоциаций (GWAS) однонуклеотидных полиморфизмов с высотой в холке. Поскольку SNP-чипы, на которых проводили генотипирование, были смоделированы с учетом позиций однонуклеотидных полиморфизмов, представленных в сборке генома Bos taurus UMD 3.1.1 (https://www.ncbi.nlm.nih.gov/as-sembly/GCF_000003055.6), для поиска генов, расположенных вблизи значимо ассоциированных SNP, также использовалась вышеуказанная сборка.
Данные о высоте в холке для исследуемых пород были получены из базы ФАО (DAD-IS, https://www.fao.org/dad-is/data/ru/). Поскольку величины показателей варьировали в разных странах, для каждой породы были рассчитаны средние значения без учета страны разведения и пола животного. Затем вычисляли среднее значение высоты в холке для всего массива исследованных пород. Расчеты и визуализацию результатов проводили в программной среде R с использованием собственных скриптов.
На основании данных о размерах тела животных все исследуемые породы были разделены на две группы. Породы, для которых средние значения высоты в холке не превышали средних значений, рассчитанных для всего массива, были отнесены к низкорослым (группа Low). Породы, для которых средние значения показателя превышали средние значения выборки, относили к высокорослым (группа High). Кроме того, исследованные породы подразделяли на группы по следующим критериям: в соответствии с направлением продуктивности (молочные, мясные), степенью давления селекции (примитивные, заводские) и распространенностью в мире (российские, трансграничные).
Достоверность разности частот встречаемости альтернативных аллелей для SNP, показавших значимые ассоциации с признаком высоты в холке, в разных группах животных определяли с использованием критерия c2 в программной среде R. При оценке достоверности разницы частот встречаемости альтернативных аллелей между трансграничными и российскими породами из анализа были исключены монгольский и киргизский скот, поскольку эти группы не разводят на территории России в товарных или племенных стадах.
Результаты. Характеристика исследованных пород КРС приведена в таблице 1.
Полногеномное исследование ассоциаций однонуклеотидных полиморфизмов с показателем высоты в холке (рис. 1) было проведено для 13 пород крупного рогатого скота. Обнаружены статистически значимые (p-value < 1e-80) ассоциации для некоторых однонуклеотидных полиморфизмов (рис. 2).
Выявлено в общей сложности четыре SNP, из которых три были локализованы на 4-й хромосоме, один — на 14-й хромосоме.
Мы провели поиск генов, расположенных на участке ±200 Kb от обнаруженных SNP (табл. 2). В проведенных ранее исследованиях было показано, что регион 24,8-25,2 Mb на 14-й хромосоме содержит гены, для которых установлена связь с размерами тела (42, 43), потреблением корма (44, 45) и фертильностью (46). На 4-й хромосоме в регионе 77,3-77,8 Mb выявлены гены, связанные с обменом веществ (47), составом (48, 49) и синтезом (50) молока, среднесуточным приростом живой массы (51). S.M. Ghoreishifar с соавт. (52) изучали следы селекции в группе из пяти пород КРС, происходящих из северной, средней и южной частей Швеции. Участки генома с наиболее значимыми сигналами отбора обнаружены на BTA5 (105,75-106,52 Mbp), BTA1 (2,25-2,52 Mbp) и BTA14 (24,42-25,11 и 25,35-25,73 Mbp). Аннотация генов в этих регионах выявила как уже известные, так и новые гены-кандидаты, ассоциированные с высотной гипоксией (DCAF8,PPP1R12A, SLC16A3, UCP2, UCP3, TIGAR), акклиматизацией к холоду (AQP3, AQP7, HSPB8), размером тела и ростом (PLAG1, KCNA6, NDUFA9, AKAP3, C5H5112orf1,RAD51AP1,FGF6, TIGAR, CCND2, CSMD3), устойчивостью к болезням и бактериальным инфекциям (CHI3L2, GBP6, PPFIBP1, REP15,CYP4F2, TIGD2, PYURF, SLC10A2, FCHSD2, ARHGEF17, RELT, PRDM2, KDM5B), с репродуктивными качествами (PPP1R12A, ZFP36L2, CSPP1), удоем и составом молока (NPC1L1, NUDCD3, ACSS1, FCHSD2).
Наши данные согласуются с опубликованными ранее, при этом регионы, описанные в предыдущих работах и расположенные на других хромосомах (53-55), не были выявлены в нашей выборке. Интересно, что в проведенных другими авторами исследованиях, в том числе с использованием контрастных пород, не обнаружено ассоциаций SNP, расположенных в регионе 77,3-77,8 Mb на BTA4, с высотой в холке (56-58).
Для выявленных SNP были рассчитаны частоты встречаемости альтернативных аллелей в каждой из пород (табл. 3).
Наиболее существенные различия между частотами альтернативных аллелей наблюдались в группе низкорослых пород: в локусе ARS-BFGL-NGS-116590 частоты аллелей G/T варьировали соответственно от 0 до 27,08 % и от 72,92 до 100 %; в локусе Hapmap53144-ss46525999 частоты аллелей T/C — от 0 до 29,17 % и от 70,83 до 100 %; в локусе BovineHD1400007259 частоты аллелей G/T — от 0 до 14,58 % и от 85,42 до 100 %. У высокорослых пород наибольшая разница в частотах аллелей T/G (соответственно 0 и 100 %) наблюдалась в локусе BovineHD1400007259 у животных черно-пестрой и абердин-ангусской пород.
Оценить статистическую значимость различий между частотами альтернативных аллелей для каждой исследуемой породы в отдельности не удалось в связи с отсутствием в некоторых породах одного из аллелей, поэтому такая оценка была проведена для групп, сформированных по разным признакам (табл. 4).
В большинстве случаев частоты альтернативных аллелей в исследованных группах статистически значимо различались. Исключение составила группа мясного скота по всем трем SNP, обнаруженным на BTA4. Кроме того, частоты в SNP BovineHD1400007259 не отличались от теоретически ожидаемых в группе примитивного скота. Такая же ситуация наблюдается в группе низкорослого скота. Это может быть связано с тем, что три из пяти формирующих ее пород, относятся к примитивному скоту (киргизский и монгольский скот, якутская порода). Интересно отметить, что у абердин-ангусской породы, несмотря на то, что она была отнесена к группе крупных пород, в распределении частот аллелей на 4-й хромосоме наблюдались те же тенденции, что и в группе мелких пород.
С высокими значениями высоты в холке связан аллель G в локусе BovineHD0400021479, аллель G в локусе ARS-BFGL-NGS-116590, аллель G в локусе BovineHD1400007259 и аллель Т в локусе Hapmap53144-ss46525999. Для SNP BovineHD1400007259, расположенного внутри гена PLAG1, частоты аллеля G, связанного с высоким ростом, согласовывались с полученными в предыдущих исследованиях для голштино-фризской и джерсейской пород (25). Ранее J. Hou с соавт. (58) изучали связь SNP rs109815800 (AC_000171.1: g.25015640, G > T, обозначенный на чипе как BovineHD1400007259) в гене PLAG1 с ростом крупного рогатого скота. Результаты анализа показали, что животные с генотипом T/T были ниже ростом, чем особи с генотипами T/G или G/G (p < 0,001).
Выявлена статистически значимая корреляция между частотами альтернативных аллелей и показателем высоты в холке для всех обнаруженных однонуклеотидных полиморфизмов (табл. 5). Почти во всех группах пород установлены статистически значимые корреляции между частотами аллелей, связанных с высоким ростом в холке, и высотой в холке. Интересно, что для группы низкорослых пород, против ожидания, не выявлено достоверной отрицательной корреляции в локусе BovineHD1400007259.
Таким образом, идентифицированы однонуклеотидные полиморфизмы ARS-BFGL-NGS-116590, Hapmap53144-ss46525999, BovineHD0400021479 и BovineHD1400007259, локализованные, согласно сборке генома UMD 3.1.1, на 4-й и 14-й хромосомах и ассоциированные с высотой в холке у крупного рогатого скота, разводимого на территории Российской Федерации. Частоты выявленных SNP в разных группах пород значимо отличались от случайно возможных. Альтернативные аллели в обнаруженных SNP имели статистически значимые положительные либо отрицательные корреляции с показателем высоты в холке. Следовательно, можно сделать вывод о неодинаковом селекционном давлении на эти локусы для разных групп пород. Многообразие и разнородность пород, представленных в выборке, позволяет рассматривать выявленные следы селекции как характерные не для одной породы, региона или направления продуктивности, а для группы пород вида Bos taurus taurus, распространение которых от центра одомашнивания шло по Дунайскому пути.
1. Характеристика исследованных российских локальных и трансграничных пород крупного рогатого скота (Bos taurus taurus) |
|||||
Порода |
Аббревиатура |
Группа |
Географическая классификация по ФАО |
Направление продуктивности |
Селекционное давление |
Абердин-ангусская |
ANG |
High |
Трансграничные |
Мясное |
Заводские |
Айрширская |
AYR |
High |
Трансграничные |
Молочное |
Заводские |
Черно-пестрая |
BLW |
High |
Трансграничные |
Молочное |
Заводские |
Холмогорская |
KHL |
High |
Российские |
Молочное |
Заводские |
Голштинская |
HLS |
High |
Трансграничные |
Молочное |
Заводские |
Ярославская |
YRS |
High |
Российские |
Молочное |
Заводские |
Тагильская |
TAG |
High |
Российские |
Молочное |
Заводские |
Истобенская |
IST |
High |
Российские |
Молочное |
Заводские |
Калмыцкая |
KLM |
Low |
Трансграничные |
Мясное |
Заводские |
Джерсейская |
JRS |
Low |
Трансграничные |
Молочное |
Заводские |
Киргизский скот |
KRG |
Low |
- |
Мясное |
Примитивные |
Монгольский скот |
MNG |
Low |
- |
Мясное |
Примитивные |
Якутская |
YKT |
Low |
Российские |
Мясное |
Примитивные |
Примечание. Low — породы, для которых средние значения высоты в холке не превышали средних значений, рассчитанных для всего массива; High — породы, для которых средние значения показателя превосходили средние значения выборки, относили к высокорослым. Прочерки означают, что породы исключены из группы, поскольку не разводятся на территории России. |
|||||
2. Гены, обнаруженные в регионе локализации однонуклеотидных полиморфизмов, значимо ассоциированных с высотой в холке у российских локальных и трансграничных пород крупного рогатого скота (Bos taurus taurus) |
|||||
Название SNP |
Хромосома |
Позиция |
Регион, Mb |
Гены |
Признаки |
Hapmap53144-ss46525999 |
4-я |
77555681 |
77,3-77,8 |
PURB, MIR4657, H2AFV, PPIA, ZMIZ2, OGDH, TMED4, DDX56, NPC1L1, NUDCD3, CAMK2B, YKT6 |
Продолжительность беременности, синтез компонентов молока, белковый и жировой обмен, среднесуточный прирост массы |
BovineHD0400021479 |
4-я |
77613816 |
|||
ARS-BFGL-NGS-116590 |
4-я |
77635835 |
|||
BovineHD1400007259 |
14-я |
25015640 |
24,8-25,2 |
LYN, RPS20, MOS, PLAG1, CHCHD7, SDR16C5, SDR16C6 |
Рост, конституция, потребление корма, фертильность |
3. Частоты аллелей в локусах, значимо связанных с признаком высоты в холке, у российских локальных и |
|||||||||
Порода |
Группа |
4-я хромосома |
14-я хромосома |
||||||
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
||||||
T |
Ga |
Ta |
C |
A |
Ga |
T |
Ga |
||
HLS |
High |
0,82 |
99,18 |
99,18 |
0,82 |
0,82 |
99,18 |
0,54 |
99,46 |
BLW |
High |
1,00 |
99,00 |
99,00 |
1,00 |
1,00 |
99,00 |
0,00 |
100,00 |
KHL |
High |
23,08 |
76,92 |
76,92 |
23,08 |
23,08 |
76,92 |
13,46 |
86,54 |
ANG |
High |
62,82 |
37,18 |
36,84 |
63,16 |
62,82 |
37,18 |
0,00 |
100,00 |
TAG |
High |
26,92 |
73,08 |
73,08 |
26,92 |
26,92 |
73,08 |
9,62 |
90,38 |
AYR |
High |
28,47 |
71,53 |
71,53 |
28,47 |
28,47 |
71,53 |
21,88 |
78,13 |
IST |
High |
45,00 |
55,00 |
54,55 |
45,45 |
36,36 |
63,64 |
9,52 |
90,48 |
YRS |
High |
45,12 |
54,88 |
55,00 |
45,00 |
45,12 |
54,88 |
17,50 |
82,50 |
KLM |
Low |
95,83 |
4,17 |
3,85 |
96,15 |
96,15 |
3,85 |
94,23 |
5,77 |
JRS |
Low |
95,31 |
4,69 |
4,69 |
95,31 |
95,31 |
4,69 |
100,00 |
0,00 |
KRG |
Low |
72,92 |
27,08 |
29,17 |
70,83 |
52,27 |
47,73 |
85,42 |
14,58 |
YKT |
Low |
98,28 |
1,72 |
0,00 |
100,00 |
87,50 |
12,50 |
98,28 |
1,72 |
MNG |
Low |
100,00 |
0,00 |
6,25 |
93,75 |
89,58 |
10,42 |
100,00 |
0,00 |
Примечание. HLS — голштинская, BLW — черно-пестрая, KHL — холмогорская, ANG — абердин-ангусская, |
|||||||||
4. Статистическая значимость различий между частотами альтернативных аллелей в локусах, |
||||||||||||
Группа пород |
4-я хромосома |
14-я хромосома |
||||||||||
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
|||||||||
T |
Ga |
p |
Ta |
C |
p |
A |
Ga |
p |
T |
Ga |
p |
|
Высокорослые |
0,204 |
0,796 |
*** |
0,795 |
0,205 |
*** |
0,201 |
0,799 |
*** |
0,090 |
0,910 |
*** |
Низкорослые |
0,349 |
0,651 |
*** |
0,082 |
0,918 |
** |
0,309 |
0,647 |
*** |
0,959 |
0,041 |
|
Мясные |
0,839 |
0,161 |
0,168 |
0,832 |
0,77 |
0,230 |
0,687 |
0,313 |
*** |
|||
Молочные |
0,218 |
0,782 |
*** |
0,781 |
0,219 |
*** |
0,215 |
0,785 |
*** |
0,152 |
0,848 |
*** |
Заводские |
0,276 |
0,724 |
*** |
0,721 |
0,279 |
*** |
0,275 |
0,725 |
*** |
0,177 |
0,823 |
*** |
Примитивные |
0,909 |
0,091 |
*** |
0,112 |
0,888 |
*** |
0,777 |
0,223 |
*** |
0,948 |
0,052 |
|
Трансграничные |
0,256 |
0,744 |
*** |
0,740 |
0,260 |
*** |
0,259 |
0,741 |
*** |
0,187 |
0,813 |
*** |
Российские |
0,486 |
0,514 |
* |
0,514 |
0,486 |
* |
0,448 |
0,552 |
0,306 |
0,694 |
*** |
|
Примечание. a — аллели, связанные с высоким ростом у крупного рогатого скота. |
||||||||||||
5. Оценка корреляционных взаимодействий между показателем высоты в холке и частотой альтернативных аллелей |
||||||||
Группа пород |
4-я хромосома |
14-я хромосома |
||||||
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
|||||
T/Ga |
p |
Ta/C |
p |
A/Ga |
p |
T/Ga |
p |
|
Высокорослые |
0,722 |
*** |
0,719 |
*** |
0,708 |
*** |
0,678 |
*** |
Низкорослые |
-0,172 |
-0,281 |
-0,498 |
** |
-0,063 |
|||
Мясные |
0,530 |
*** |
0,456 |
* |
0,090 |
0,767 |
*** |
|
Молочные |
0,908 |
*** |
0,908 |
*** |
0,907 |
*** |
0,751 |
*** |
Заводские |
0,829 |
*** |
0,827 |
*** |
0,821 |
*** |
0,729 |
*** |
Примитивные |
0,962 |
*** |
0,859 |
*** |
0,96 |
*** |
0,975 |
*** |
Трансграничные |
0,876 |
*** |
0,875 |
*** |
0,876 |
*** |
0,840 |
*** |
Российские |
0,981 |
*** |
0,982 |
*** |
0,989 |
*** |
0,971 |
*** |
Вся выборка |
0,838 |
*** |
0,826 |
*** |
0,752 |
** |
0,854 |
*** |
Примечание. a — аллели, связанные с высоким ростом у крупного рогатого скота. Коэффициенты корреляции показаны |
||||||||
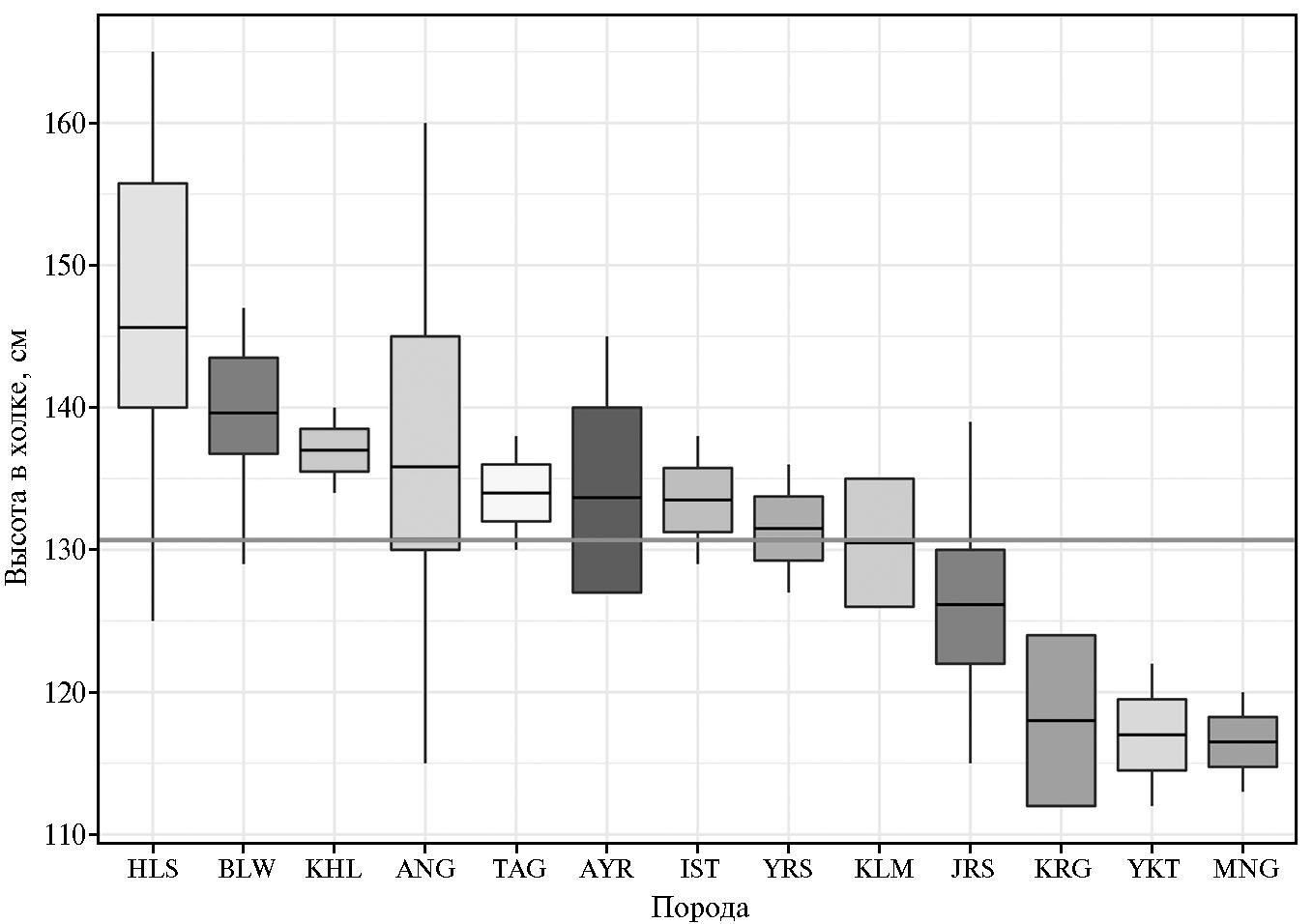
Рис. 1. Средние значения высоты в холке для российских локальных и трансграничных пород крупного рогатого скота (Bos taurus taurus): HLS — голштинская, BLW — черно-пестрая, KHL — холмогорская, ANG — абердин-ангусская, TAG — тагильская, AYR — айрширская, IST — истобенская, YRS — ярославская, KLM — калмыцкая, JRS — джерсейская, KRG — киргизский скот, YKT — якутская, MNG — монгольский скот. На диаграммах размаха для каждой породы приведены значения нижнего квартиля, арифметической средней и верхнего квартиля. Горизонтальная линия — среднее значение, рассчитанное для всего массива исследованных пород.
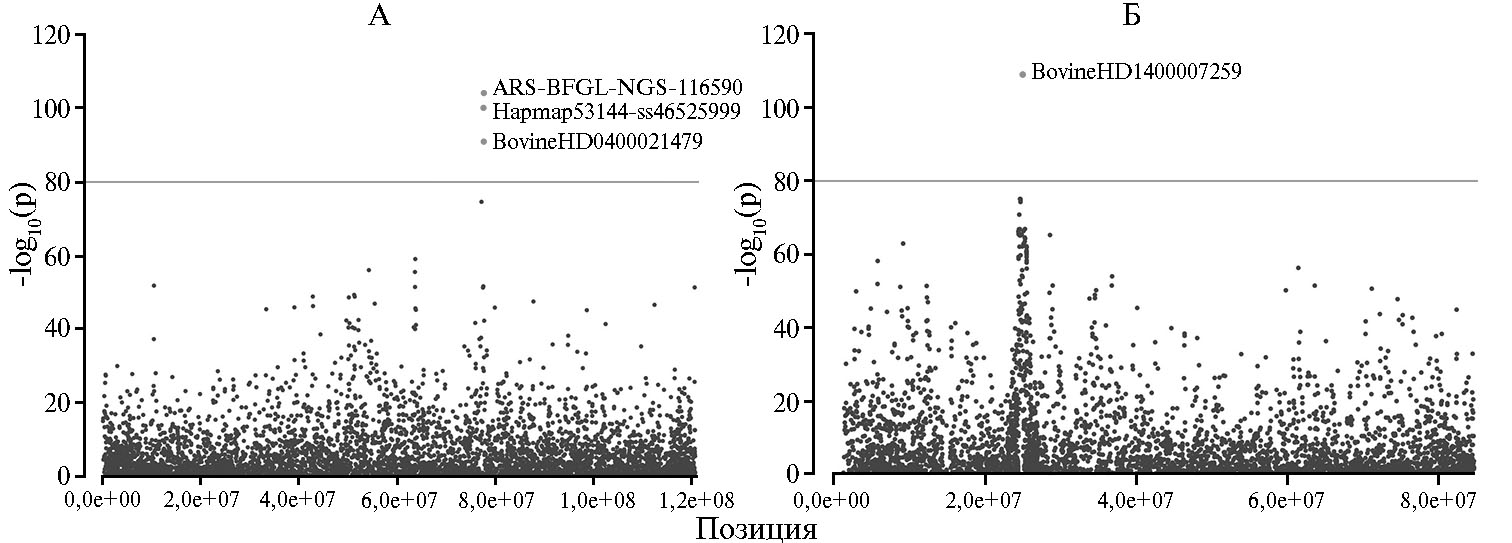
Рис. 2. Локализация однонуклеотидных полиморфизмов, значимо ассоциированных с показателем высоты в холке у российских локальных и трансграничных пород крупного рогатого скота (Bos taurus taurus): А — 4-я хромосома, Б — 14-я хромосома; горизонтальные линии — пороговые значения отрицательного логарифма.
ЛИТЕРАТУРА
- Петрова М.Ю., Чернигов Ю.В., Кузнецова Т.Ш. Подбор быков-производителей и оценка их дочерей по типу телосложения. Вестник ОмГАУ, 2019, 2(34): 120-125.
- Goddard M., Hayes B. Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nature Reviews Genetics,2009, 10: 381-391 (doi: 10.1038/nrg2575).
- Сермягин А.А., Быкова О.А., Лоретц О.Г., Костюнина О.В., Зиновьева Н.А. Оценка геномной вариабельности продуктивных признаков у животных голштинизированной черно-пестрой породы на основе GWAS анализа и ROH паттернов. Сельскохозяйственная биология, 2020, 55(2): 257-274 (doi: 10.15389/agrobiology.2020.2.257rus).
- Matukumalli L.K., Lawley C.T., Schnabel R.D., Taylor J.F., Allan M.F., Heaton M.P., O'Connell J., Moore S.S., Smith T.P.L., Sonstegard T.S., Van Tassell C.P. Development and characterization of a high density SNP genotyping assay for cattle. PLoS ONE, 2009, 4(4): e5350 (doi: 10.1371/journal.pone.0005350).
- Pant S.D., Schenkel F.S., Verschoor C.P., You Q., Kelton D.F., Moore S.S., Karrow N.A. A principal component regression based genome wide analysis approach reveals the presence of a novel QTL on BTA7 for MAP resistance in Holstein cattle. Genomics, 2010, 95(3): 176-182 (doi: 10.1016/j.ygeno.2010.01.001).
- Crispim A.C., Kelly M.J., Guimarães S.E., e Silva F.F., Fortes M.R., Wenceslau R.R., Moore S. Multi-trait GWAS and new candidate genes annotation for growth curve parameters in Brahman cattle. PLoS ONE, 2015, 10(10): e0139906 (doi: 10.1371/journal.pone.0139906).
- Hoshiba H., Setoguchi K., Watanabe T., Kinoshita A., Mizoshita K., Sugimoto Y., Takasuga A. Comparison of the effects explained by variations in the bovine PLAG1 and NCAPG genes on daily body weight gain, linear skeletal measurements and carcass traits in Japanese Black steers from a progeny testing program. Animal Science Journal, 2013, 84(7): 529-534 (doi: 10.1111/asj.12033).
- Snelling W.M., Allan M.F., Keele J.W., Kuehn L.A., McDaneld T., Smith T.P.L., Sonstegard T.S., Thallman R.M., Bennett G.L. Genome-wide association study of growth in crossbred beef cattle. Journal of Animal Science, 2010, 88(3): 837-848 (doi: 10.2527/jas.2009-2257).
- Bolormaa S., Hayes B.J., Savin K., Hawken R., Barendse W., Arthur P.F., Herd R.M., Goddard M.E. Genome-wide association studies for feedlot and growth traits in cattle. Journal of Animal Science, 2011, 89(6): 1684-1697 (doi: 10.2527/jas.2010-3079).
- Barendse W. Haplotype analysis improved evidence for candidate genes for intramuscular fat percentage from a genome wide association study of cattle. PLoS ONE, 2011, 6(12): e29601 (doi: 10.1371/journal.pone.0029601).
- Dang C.G., Cho S.H., Sharma A., Kim H.C., Jeon G.J., Yeon S.H., Hong S.K., Park B.Y., Kang H.S., Lee S.H. Genome-wide association study for Warner-Bratzler shear force and sensory traits in Hanwoo (Korean cattle). Asian-Australasian Journal of Animal Sciences, 2014, 27(9): 1328-1335 (doi: 10.5713/ajas.2013.13690).
- Wu Y., Fan H., Wang Y., Zhang L., Gao X., Chen Y., Li J., Ren H., Gao H. Genome-wide association studies using haplotypes and individual SNPs in Simmental cattle. PLoS ONE, 2014, 9(10): e109330 (doi: 10.1371/journal.pone.0109330).
- Weng Z.Q., Su H.L., Saatchi M., Lee J., Thomas M.G., Dunkelberger J.R., Garrick D.J. Genome-wide association study of growth and body composition traits in Brangus beef cattle. Livestock Science, 2016, 183: 4-11 (doi: 10.1016/j.livsci.2015.11.011).
- Littlejohn M., Grala T., Sanders K., Walker C., Waghorn G., Macdonald K., Coppieters W., Georges M., Spelman R., Hillerton E., Davis S., Snell R. Genetic variation in PLAG1 associates with early life body weight and peripubertal weight and growth in Bos taurus. Animal Genetics, 2012, 43(5): 591-594 (doi: 10.1111/j.1365-2052.2011.02293.x).
- Bolormaa S., Pryce J.E., Kemper K., Savin K., Hayes B.J., Barendse W. Zhang Y., Reich C. M., Mason B.A., Bunch R.J., Harrison B.E., Reverter A., Herd R.M., Tier B., Graser H.-U., Goddard M.E. Accuracy of prediction of genomic breeding values for residual feed intake and carcass and meat quality traits in Bos taurus, Bos indicus, and composite beef cattle. Journal of Animal Science, 2013, 91(7): 3088-3104 (doi: 10.2527/jas.2012-5827).
- Sasaki S., Ibi T., Matsuhashi T., Takeda K., Ikeda S., Sugimoto M., Sugimoto Y. Genetic variants in the upstream region of activin receptor IIA are associated with female fertility in Japanese Black cattle. BMC Genetics, 2015, 16: 123.
- Galton F. Regression towards mediocrity in hereditary stature. The Journal of the Anthropological Institute of Great Britain and Ireland, 1886, 15: 246-263. Режим доступа: http://www.jstor.org/stable/2841583. Дата обращения 10.10.2021.
- Fisher R.A. The correlation between relatives on the supposition of Mendelian inheritance. Earth and Environmental Science Transactions of the Royal Society of Edinburgh, 1918, 52(2): 399-433 (doi: 10.1017/S0080456800012163).
- Денискова Т.Е., Петров С.Н., Сермягин А.А., Доцев А.В., Форнара М.С., Reyer H., Wimmers K., Багиров В.А., Brem G., Зиновьева Н.А. Поиск геномных вариантов, ассоциированных с живой массой у овец, на основе анализа высокоплотных SNP генотипов. Сельскохозяйственнаябиология, 2021, 56(2): 279-291 (doi: 10.15389/agrobiology.2021.2.279rus).
- Visscher P.M. , Macgregor S., Benyamin B., Zhu G., Gordon S., Medland S., Hill W.G., Hottenga J.-J., Willemsen G., Boomsma D.I., Liu Y.-Z., Deng H.-W., Montgomery G.W., Martin N.G. Genome partitioning of genetic variation for height from 11,214 sibling pairs. The American Journal of Human Genetics, 2007, 81: 1104-1110 (doi: 10.1086/522934).
- Ajmone-Marsan P., Garcia J.F., Lenstra J.A. On the origin of cattle: how aurochs became cattle and colonized the world. Evolutionary Anthropology, 2010, 19(4): 148-157 (doi: 10.1002/evan.20267).
- Nelsen T.C., Short R.E., Urick J.J., Reynolds W.L. Heritabilities and genetic correlations of growth and reproductive measurements in Hereford bulls. Journal of Animal Science, 1986, 63(2): 409-417 (doi: 10.2527/jas1986.632409x).
- Northcutt S.L., Wilson D.E. Genetic parameter estimates and expected progeny differences for mature size in Angus cattle. Journal of Animal Science, 1993, 71(5): 1148-1153 (doi: 10.2527/1993.7151148x).
- McClure M.C., Morsci N.S., Schnabel R.D., Kim J.W., Yao P., Rolf M.M., McKay S.D., Gregg S.J., Chapple R.H., Northcutt S.L., TaylorJ.F. A genome scan for quantitative trait loci influencing carcass, post-natal growth and reproductive traits in commercial Angus cattle. Animal Genetics, 2010, 41(6): 597-607 (doi: 10.1111/j.1365-2052.2010.02063.x).
- Karim L., Takeda H., Lin L., Druet T., Arias J.A., Baurain D., Cambisano N., Davis S.R., Farnir F., Grisart B., Harris B.L., Keehan M.D., Littlejohn M.D., Spelman R.J., Georges M., Coppieters W. Variants modulating the expression of a chromosome domain encompassing PLAG1 influence bovine stature. Nature Genetics, 2011, 43: 405-413 (doi: 10.1038/ng.814).
- Randhawa I.A.S., Khatkar M.S., Thomson P.C., Raadsma H.W. Composite selection signals for complex traits exemplified through bovine stature using multibreed cohorts of European and African Bos taurus. G3 Genes|Genomes|Genetics, 2015, 5(7): 1391-1401 (doi: 10.1534/g3.115.017772).
- Zimin A.V., Delcher A.L., Florea L., Kelley D.R., Schatz M.C., Puiu D., Hanrahan F., Pertea G., Van Tassell C.P., Sonstegard T.S., Marçais G., Roberts M., Subramanian P., Yorke J.A., Salzberg S.L. A whole-genome assembly of the domestic cow, Bos taurus. Genome Biology, 2009, 10: R42 (doi: 10.1186/gb-2009-10-4-r42).
- Boitard S., Boussaha M., Capitan A., Rocha D., Servin B. Uncovering adaptation from sequence data: lessons from genome resequencing of four cattle breeds. Genetics, 2016, 203(1): 433-450 (doi: 10.1534/genetics.115.181594).
- Kas K., Voz M.L., Röijer E., Åström A.K., Meyen E., Stenman G., Van de Ven W.J. Promoter swapping between the genes for a novel zinc finger protein and β-catenin in pleiomorphic adenomas with t(3;8)(p21;q12) translocations. Nature Genetics, 1997, 15: 170-174 (doi: 10.1038/ng0297-170).
- Wood A., Esko T., Yang J. et al. Defining the role of common variation in the genomic and biological architecture of adult human height. Nature Genetics, 2014, 46: 1173-1186 (doi: 10.1038/ng.3097).
- Pryce J.E., Hayes B.J., Bolormaa S., Goddard M.E. Polymorphic regions affecting human height also control stature in cattle. Genetics, 2011, 187(3): 981-984 (doi: 10.1534/genetics.110.123943).
- Fortes M.R.S., Kemper K., Sasazaki S., Reverter A., Pryce J.E., Barendse W., Bunch R., McCulloch R., Harrison B., Bolormaa S., Zhang Y.D., Hawken R.J., Goddard M.E., Lehnert S.A. Evidence for pleiotropism and recent selection in the PLAG1 region in Australian Beef cattle. Animal Genetics, 2013, 44: 636-647 (doi: 10.1111/age.12075).
- Pausch H., Flisikowski K., Jung S., Emmerling R., Edel C., Götz K.U., Fries R. Genome-wide association study identifies two major loci affecting calving ease and growth-related traits in cattle. Genetics, 2011, 187(1): 289-297 (doi: 10.1534/genetics.110.124057).
- Nishimura S., Watanabe T., Mizoshita K., Tatsuda K., Fujita T., Watanabe N., Sugimoto Y., Takasuga A. Genome-wide association study identified three major QTL for carcass weight including the PLAG1-CHCHD7 QTN for stature in Japanese Black cattle. BMC Genetics, 2012, 13: 40 (doi: 10.1186/1471-2156-13-40).
- Utsunomiya Y.T., Do Carmo A.S., Carvalheiro R., Neves H.H., Matos M.C., Zavarez L.B., Pérez O’Brien A.M., Sölkner J., McEwan J.C, Cole J.B., Van Tassell C.P., Schenkel F.S., da Silva M.V.G.B., Porto Neto L.R., Sonstegard T.S., Garcia J.F. Genome-wide association study for birth weight in Nellore cattle points to previously described orthologous genes affecting human and bovine height. BMC Genetics, 2013, 14: 52 (doi: 10.1186/1471-2156-14-52).
- Zhong J.-L., Xu J.-W., Wang J., Wen Y.-F., Niu H., Zheng L., He H., Peng K., He P., Shi S.Y., Huang Y.-Q., Lei C.-Z., Dang R.-H., Lan X.-Y., Qi X.-L., Chen H., Huang Y.-Z. A novel SNP of PLAG1 gene and its association with growth traits in Chinese cattle. Gene, 2019, 689: 166-171 (doi: 10.1016/j.gene.2018.12.018).
- Abdelmanova A.S., Kharzinova V.R., Volkova V.V., Mishina A.I., Dotsev A.V., Sermyagin A.A., Boronetskaya O.I., Petrikeeva L.V., Chinarov R.Y., Brem G., Zinovieva N.A. Genetic diversity of historical and modern populations of russian cattle breeds revealed by microsatellite analysis. Genes, 2020, 11(8): 940 (doi: 10.3390/genes11080940).
- Abdelmanova A.S., Kharzinova V.R., Volkova V.V., Dotsev A.V., Sermyagin A.A., Chinarov R.Y., Zinovieva N.A., Boronetskaya O.I., Lutshikhina E.M., Sölkner J., Brem G. Comparative study of the genetic diversity of local steppe cattle breeds from Russia, Kazakhstan and Kyrgyzstan by microsatellite analysis of museum and modern samples. Diversity, 2021, 13(8): 351 (doi: 10.3390/d13080351).
- Зиновьева Н.А., Сермягин А.А., Доцев А.В., Боронецкая О.И., Петрикеева Л.В., Абдельманова А.С., Brem G. Генетические ресурсы животных: развитие исследований аллелофонда российских пород крупного рогатого скота – миниобзор. Сельскохозяйственная биология, 2019, 54(4): 631-641 (doi: 10.15389/agrobiology.2019.4.631rus).
- Sermyagin A.A., Dotsev A.V., Gladyr E.A., Traspov A.A., Deniskova T.E., Kostyunina O.V., Reyer H., Wimmers K., Barbato M., Paronyan I.A., Plemyashov K.V., Sölkner J., Popov R.G., Brem G., Zinovieva N.A. Whole-genome SNP analysis elucidates the genetic structure of Russian cattle and its relationship with Eurasian taurine breeds. Genetics Selection Evolution, 2018, 50(1): 37 (doi: 10.1186/s12711-018-0408-8).
- Chang C.C., Chow C.C., Tellier L.C., Vattikuti S., Purcell S.M., Lee J.J. Second-generation PLINK: rising to the challenge of larger and richer datasets. GigaScience, 2015, 4(1): s13742-015-0047-8 (doi: 10.1186/s13742-015-0047-8).
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Deniskova T.E., Abdelmanova A.S., Kharzinova V.R., Sölkner J., Reyer H., Wimmers K., Brem G. Selection signatures in two oldest Russian native cattle breeds revealed using high-density single nucleotide polymorphism analysis. PLoS ONE, 2020, 15(11): e0242200 (doi: 10.1371/journal.pone.0242200).
- Pryce J.E., Arias J., Bowman P.J., Davis S.R., Macdonald K.A., Waghorn G.C., Wales W.J., Williams Y.J., Spelman R.J., Hayes B.J. Accuracy of genomic predictions of residual feed intake and 250-day body weight in growing heifers using 625,000 single nucleotide polymorphism markers. Journal of Dairy Science, 2012, 95(4): 2108-2119 (doi: 10.3168/jds.2011-4628).
- de Las Heras-Saldana S., Clark S.A., Duijvesteijn N., Gondro C., van der Werf J.H.J., Chen Y. Combining information from genome-wide association and multi-tissue gene expression studies to elucidate factors underlying genetic variation for residual feed intake in Australian Angus cattle. BMC Genomics, 2019, 20(1): 939 (doi: 10.1186/s12864-019-6270-4).
- Cheruiyot E.K., Bett R.C., Amimo J.O., Zhang Y., Mrode R., Mujibi F.D.N. Signatures of Selection in admixed dairy cattle in Tanzania. Frontiers in Genetics, 2018, 9: 607 (doi: 10.3389/fgene.2018.00607).
- Taye M., Yoon J., Dessie T., Cho S., Oh S.J., Lee H.K., Kim H. Deciphering signature of selection affecting beef quality traits in Angus cattle. Genes & Genomics, 2018, 40(1): 63-75 (doi: 10.1007/s13258-017-0610-z).
- Lee S.H., Zhu C., Peng Y., Johnson D. T., Lehmann L., Sun Z. Identification of a novel role of ZMIZ2 protein in regulating the activity of the Wnt/β-catenin signaling pathway. The Journal of biological chemistry, 2013, 288(50): 35913-35924 (doi: 10.1074/jbc.M113.529727).
- Sanchez M.-P., Ramayo-Caldas Y., Wolf V, Laithier C., El Jabri M., Michenet A., Boussaha M., Taussat S., Fritz S., Delacroix-Buchet A., Brochard M., Boichard D. Sequence-based GWAS, network and pathway analyses reveal genes co-associated with milk cheese-making properties and milk composition in Montbéliarde cows. Genetics Selection Evolution, 2019, 51: 34 (doi: 10.1186/s12711-019-0473-7).
- Sanchez M.P., Govignon-Gion A., Croiseau P., Fritz S., Hozé C., Miranda G., Martin P., Barbat-Leterrier A., Letaïef R., Rocha D., Brochard M., Boussaha M., Boichard D. Within-breed and multi-breed GWAS on imputed whole-genome sequence variants reveal candidate mutations affecting milk protein composition in dairy cattle. Genetics Selection Evolution, 2017, 49(1): 68 (doi: 10.1186/s12711-017-0344-z).
- Huo N., Yu M., Li X., Zhou C., Jin X., Gao X. PURB is a positive regulator of amino acid-induced milk synthesis in bovine mammary epithelial cells. Journal of Cellular Physiology, 2019, 234(5): 6992-7003 (doi: 10.1002/jcp.27452).
- Lu D., Miller S., Sargolzaei M., Kelly M., Vander Voort G., Caldwell T., Wang Z., Plastow G., Moore S. Genome-wide association analyses for growth and feed efficiency traits in beef cattle. Journal of Animal Science, 2013, 91(8): 3612-3633 (doi: 10.2527/jas.2012-5716).
- Ghoreishifar S.M., Eriksson S., Johansson A.M., Khansefid M., Moghaddaszadeh-Ahrabi S., Parna N., Davoudi P., Javanmard A. Signatures of selection reveal candidate genes involved in economic traits and cold acclimation in five Swedish cattle breeds. Genetics Selection Evolution, 2020, 52(1): 52 (doi: 10.1186/s12711-020-00571-5).
- Setoguchi K., Furuta M., Hirano T., Nagao T., Watanabe T., Sugimoto Y., Takasuga A. Cross-breed comparisons identified a critical 591-kb region for bovine carcass weight QTL (CW-2) on chromosome 6 and the Ile-442-Met substitution in NCAPG as a positional candidate. BMC Genetics, 2009, 10: 43 (doi: 10.1186/1471-2156-10-43).
- Vanvanhossou S., Scheper C., Dossa L.H., Yin T., Brügemann K., König S. A multi-breed GWAS for morphometric traits in four Beninese indigenous cattle breeds reveals loci associated with conformation, carcass and adaptive traits. BMC Genomics, 2020, 21(1): 783 (doi: 10.1186/s12864-020-07170-0).
- Bouwman A.C., Daetwyler H.D., Chamberlain A.J., Ponce C.H., Sargolzaei M., Schenkel F.S., Sahana G., Govignon-Gion A., Boitard S., Dolezal M., Pausch H., Brøndum R.F., Bowman P.J., Thomsen B., Guldbrandtsen B., Lund M.S., Servin B., Garrick D.J., Reecy J., Vilkki J., Bagnato A., Wang M., Hoff J.L., Schnabel R.D., Taylor J.F., Vinkhuyzen A.A.E., Panitz F., Bendixen C., Holm L.E., Gredler B., Hozé C., Boussaha M., Sanchez M.P., Rocha D., Capitan A., Tribout T., Barbat A., Croiseau P., Drögemüller C., Jagannathan V., Vander Jagt C., Crowley J.J., Bieber A., Purfield D.C., Berry D.P., Emmerling R., Götz K.U., Frischknecht M., Russ I., Sölkner J., Van Tassell C.P., Fries R., Stothard P., Veerkamp R.F., Boichard D., Goddard M.E., Hayes B.J. Meta-analysis of genome-wide association studies for cattle stature identifies common genes that regulate body size in mammals. Nature Genetics, 2018, 50: 362-367 (doi: 10.1038/s41588-018-0056-5).
- Doyle J.L., Berry D.P., Veerkamp R.F., Carthy T.R., Walsh S.W., Evans R.D., Purfield D.C. Genomic regions associated with skeletal type traits in beef and dairy cattle are common to regions associated with carcass traits, feed intake and calving difficulty. Frontiers in Genetics, 2020, 11: 20 (doi: 10.3389/fgene.2020.00020).
- An B., Xu L., Xia J., Wang X., Miao J., Chang T., Song M., Ni J., Xu L., Zhang L., Li J., Gao H. Multiple association analysis of loci and candidate genes that regulate body size at three growth stages in Simmental beef cattle. BMC Genetics, 2020 21(1): 32 (doi: 10.1186/s12863-020-0837-6).
- Hou J., Qu K., Jia P., Hanif Q., Zhang J., Chen N., Dang R., Chen H., Huang B., Lei C. A SNP in PLAG1 is associated with body height trait in Chinese cattle. Animal Genetics, 51(1): 87-90 (doi: 10.1111/age.12872).