doi: 10.15389/agrobiology.2022.5.921rus
УДК 631.524.5:57.084.1
Исследование выполнено при поддержке гранта Министерства науки и высшего образования РФ № FZWG-2021-0018 в рамках государственного задания по теме «Разработка и внедрение в практику комплексных физико-химических методов оценки состояния растений для решения задач направленного формирования устойчивых фитоценозов культур различного функционального назначения в условиях промышленных и аграрных предприятий» для создания лаборатории физико-химических методов исследования растений.
ОЦЕНКА ТОЧНОСТИ ФЕНОТИПИРОВАНИЯ МОРФОЛОГИЧЕСКИХ ПРИЗНАКОВ Syringa vulgaris L. С ПОМОЩЬЮ ЛАЗЕРНОГО 3D-СКАНЕРА PlantEye F500 В ЗАВИСИМОСТИ ОТ РАСПОЛОЖЕНИЯ РАСТЕНИЙ НА СКАНИРУЕМОЙ ПОВЕРХНОСТИ
М.Ю. ТРЕТЬЯКОВ1 ✉, В.К. ТОХТАРЬ1, Е.В. ЖУРАВЛЕВА2, Д.В. БИРЮКОВ1
Так как методические приемы прямой генетики применимы только для моногенных признаков, свойства полученного селекционного материала, линии или сорта должны быть проверены в полевых условиях, поскольку присутствие в геноме желательного гена, подтвержденное молекулярными методами, не всегда приводит к формированию ценного для селекции признака. Системы на основе технологий 3D изображений позволяют получить модель растения, а также информацию о морфологических параметрах. Однако крайне мало внимания уделяется составлению протоколов проведения феноскрининга. Целью настоящего исследования была сравнительная оценка точности определения морфологических характеристик у растений сирени традиционными методами и с использованием технологии машинного зрения в зависимости от расположения объекта на сканируемой поверхности. В качестве модели для отработки методики феноскрининга выбрали микроклоны сирени (Syringa vulgaris L.) сорта Милосердие. Микроклоны характеризуются высокой степенью морфологической однородности и небольшие по размеру, что позволяет проводить измерения и сопоставлять полученные данные в достаточно больших выборках, нормализуя их к средним значениям. Учеты выполняли после окончания адаптации растений и культивирования в течение 1 мес в тепличных боксах. При традиционной морфометрии выборка составляла 10 микроклонов, у которых измеряли высоту мерной линейкой, площадь поверхности каждого листа — контурным методом. При сканировании (3D-сканер PlantEye F500, «Phenospex B.V.», Нидерланды) каждое из 10 отобранных растений располагали в пяти разных точках сканируемой поверхности и в одном и том же положении проводили не менее пяти повторных сканирований. При применении технологии машинного зрения определяли 3D площадь листьев, проецируемую площадь листьев, цифровую биомассу, высоту, максимальную высоту, наклон листьев, угол наклона листьев, глубину проникновения света. Установлено, что для получения объективных и сопоставимых результатов при использовании 3D-сканера оптимально располагать растения в центре сканируемой поверхности в одном и том же положении, а в качестве морфологических параметров для идентификации сортов и фиксации приростов можно рекомендовать следующие параметры: площадь листа, проекционная площадь листа, высота, угол наклона листа. Для каждого вида растений необходимо проводить первичные морфологические исследования традиционными методами, а затем сравнивать полученные данные с результатами сканирования, чтобы внести поправочные коэффициенты и подтвердить информативность используемого набора признаков, тем самым повышая точность данных, полученных в результате использования технологии машинного зрения.
Ключевые слова: фенотипирование, морфология, Syringa vulgaris L., машинное зрение, 3D-сканирование.
В современных исследованиях огромное внимание уделяется проблеме структуры генома растений (1, 2). Проведение таких исследований позволяет выявить генетические детерминанты, определяющие хозяйственно ценные признаки, понять механизмы работы генов (3-5), определить популяционную изменчивость (6), проводить на ранних стадиях развития растений идентификацию сортов (7, 8), а также расширить представления об организации генетического материала, путях и закономерностях его эволюции (9, 10).
Широко используемые в настоящее время подходы прямой классической генетики, при которых гены идентифицируются по кодируемым ими признакам, сменяются методами обратной генетикой, когда анализируют не фенотип и его генетический контроль, а саму последовательность ДНК и выявляют ее фенотипические эффекты (11-14). Смена парадигмы обусловлена тем, что методические приемы прямой генетики применимы только для моногенных признаков. Однако в большинстве случаев свойства биологических объектов полигенны и формируются в результате совокупного действия нескольких генов, или же фенотипические проявления могут быть следствием мутации разных генов (15). В связи с этим признаки полученного селекционного материала, линии или сорта должны быть проверены в полевых условиях, так как присутствие в геноме желательного гена, подтвержденное молекулярными методами, не всегда приводит к формированию ценного для селекции признака (16-18). Кроме того, при анализе качественных и количественных морфологических признаков необходимо учитывать модификационную изменчивость, обусловленную действием разнообразных факторов внешней среды (19).
Таким образом, морфологическая оценка растений — обязательный этап селекционно-генетических исследований (15, 17). Современные методы фенотипирования, основанные на технологиях машинного зрения, обладают высокой производительностью, позволяют получать данные в режиме реального времени по нескольким морфологическим параметрам (20, 21). Автоматизация процессов фенотипирования существенно ускоряет анализ и повышает его точность, исключая человеческий фактор как источник субъективной оценки результатов, а также позволяет исследовать параметры, которые не использовались при традиционных морфометрических измерениях (22).
В качестве важнейших морфологических признаков при фенотипировании растений можно выделить размер растения, тип расположения листьев, форму и площадь листовой пластинки. Существуют автоматизированные платформы, позволяющие проводить определение видов растений по фотографиям, например INaturalist (https://www.inaturalist.org/) и PlantNet (https://plantnet.org/). Однако точность фенотипирования зависит от объема накопленного фотоматериала (количества и качества фотографий, выполненных в разные фазы вегетации растений), частоты встречаемости определяемого вида в изучаемой местности, а также фактического подтверждения его идентификации при натурных наблюдениях (23, 24). Таким образом, при использовании автоматизированных платформ можно определить виды растений при достаточной наполненности базы, но не удается оценить модификационную изменчивость морфологических признаков, а также определять сорта.
Системы на основе технологий 3D изображений позволяют получить модель растения, а также информацию о морфологических параметрах (25). При этом главную роль играет программное обеспечение обработки изображения, а не разрешающая способность сканера (26). Как следствие, современные исследования по фенотипированию в основном посвящены разработке программного обеспечения, совершенствованию системы расположения камер (27). Однако крайне мало внимания уделяется составлению протоколов проведения феноскрининга (28-30). Несомненно, что автоматизация процессов фенотипирования, проводимого как в лабораторных, так и в полевых условиях, не только значительно ускорит оценку селекционного материала, но и позволит повысить гомогенность отобранных растений при работе с однолетними культурами (31-34). Несмотря на множество публикаций об использовании 3D-сканеров для оценки морфологических параметров, в литературе достаточно поверхностно освещены вопросы точности морфологических характеристик при фенотипировании растений в зависимости от их расположения на сканируемой поверхности (35-37).
В настоящей работе мы сопоставили результаты прямых морфометрических измерений, проводимых персоналом, и косвенных, полученных с помощью технологии машинного зрения, и выявили условия, несоблюдение которых при выполнении феноскрининга может привести к недостоверности его результатов.
Целью нашего исследования была сравнительная оценка точности определения морфологических характеристик у растений сирени традиционными методами и с использованием технологии машинного зрения в зависимости от расположения объекта на сканируемой поверхности.
Методика. Растения сирени (Syringa vulgaris L.) сорта Милосердие получали методом in vitro после адаптации. Учеты выполняли после окончания прохождения этапа адаптации и культивирования растений в течение 1 мес в тепличных боксах.
При традиционной морфометрии выборка составляла 10 микроклонов, у которых высоту растений измеряли линейкой, площадь поверхности каждого листа — контурным методом.
Сканирование проводили на мультиспектральной 3D установке PlantEye F500 («Phenospex B.V.», Нидерланды) (оборудование УНУ Ботанический сад Белгородского государственного национального исследовательского университета, https://ckp-rf.ru/usu/200997/). Каждое из 10 отобранных растений при сканировании располагали в пяти разных точках сканируемой поверхности, и в одном и том же положении проводили не менее пяти повторных сканирований. При помощи установки PlantEye F500 анализировались значения следующих морфометрических параметров: 3D площадь листьев растения (3D Leaf Area), см2; проецируемая площадь листьев растения (Projected Leaf Area), см2; цифровая биомасса (Digital Biomass), см3; высота (Height), мм; максимальная высота (Height Max), мм; наклон листьев растения (Leaf Inclination), см2/см2; угол наклона листьев растения (Leaf Angle), °; глубина проникновения света в листья растения (Light Penetration Depth), мм, для обработки полученных данных использовали программное обеспечение PlantEye F500 HortControl.
Рассчитывали средние арифметические значения (M) и доверительные интервалы (±CI) при доверительном уровне р = 0,05, выполняли корреляционный анализ.
Результаты. Выбор микроклонов в качестве объекта обусловлен высокой степенью морфологической однородности и небольшими размерами растений, что позволяет проводить измерения и сопоставлять полученные данные в достаточно больших выборках, нормализуя их к средним значениям.
На первом этапе исследования мы провели морфометрические измерения высоты растения (22,7±2,3 см) и площади листовой поверхности (388,3±12,3 см2). Цифровая биомасса (произведение высоты растения на площадь листовой поверхности) составила 8814,41±325 см3.
Расположение каждого растения на сканируемой поверхности при 3D-сканировании показано на рисунке 1, А. При использовании 3D-сканера PlantEye F500 для измерения площади листьев создаются точки в облаке точек, принадлежащие одному массиву, которые триангулируются (соединяются в треугольники). Поскольку допускается неравномерное распределение точек в пространстве, размер треугольников может варьироваться (см. рис. 1, Б).
Группа треугольников, образующих однородную поверхность, представляет собой домен и соответствует одному листу. Затем общая площадь 3D-сканирования листьев растения рассчитывается как сумма площадей элементарных треугольников всех сканируемых листьев одного растения (рис. 2).
Из представленных данных видно, что местоположение растения существенно повлияло на результат. В точках 2 и 4 были получены наименьшие значения площади листьев, в точках 3 и 5 — наибольшие, а расположение растения в точке 1 соответствовало среднему значению, полученному по всем пяти точкам. Необходимо отметить и тот факт, что доверительный интервал по пяти повторным сканированиям у 10 растений оказался наибольшим именно в точке 1. То есть при проведении автоматизированных измерений на полученные данные влияет положение растения относительно области сканирования, то есть даже при статичном расположении объекта и отсутствии внешних изменяющихся факторов (изменение освещенности не влияло на результаты сканирования) в точке сканирования, расположенной ближе всего к началу области сканирования, наблюдалась высокая степень нестабильности полученных данных.
Площадь поверхности всех листьев растения при применении контурного метода в 3,2 раза превысила величину при 3D-сканировании. Такое значительное расхождение объясняется тем, что некоторые листья перекрывают друг друга, что занижает показатель. Следовательно, для использования 3D-сканера при оценке площади листьев требуется введение поправочного коэффициента, рассчитанного на основании сравнения данных, полученных разными методами (в нашем случае контурным методом и в результате сканирования с помощью PlantEye F500).
Проецируемая площадь листьев определяется как площадь проекции всех элементарных треугольников на плоскость X-Y. При этом она эквивалентна значению, которое может быть измерено с помощью обычной 2D-камеры. PlantEye F500 измеряет площадь проекции растения на плоскость X-Y и превращает 3D-объект в плоский 2D-объект (рис. 3).
Из гистограммы видно, что нахождение растения в точках 1, 2, 3 и 4 существенно не влияло на полученные данные, тогда как расположение в центре сканируемой поверхности (точка 5) привело как к достоверному увеличению площади листьев в проекции, так и к увеличению доверительного интервала. Полученные при 3D-сканировании средние значения площади всех листьев по 10 растениям и проецируемой площади листьев коррелируют (r = 0,55, p < 0,05). При этом величина проецируемой площади листьев намного меньше площади листьев, так как при 3D-сканировании листья с одинаковым положением в плоскости X-Y, но находящиеся на разной высоте над землей, не учитываются дважды. То есть проецируемая площадь листьев служит аналогом проективного покрытия, определяющего относительную площадь проекции листьев на подстилающей поверхности.
Цифровую биомассу при использовании прибора PlantEye F500 вычисляют, как произведение высоты и площади листьев при условии, что растение имеет структуру побегов, объем которых можно рассчитать с учетом высоты и длины (рис. 4).
Аналогичным образом местоположение растения влияло на результаты оценки как величины цифровой биомассы, так и площади листьев (r = 0,98, р < 0,05). Наименьшую величину цифровой биомассы получили в точках 2 и 4, наибольшую — в точках 3 и 5, а точка 1 соответствовала среднему значению, полученному по всем пяти точкам, в точке 1 значение доверительного интервала тоже было наибольшим, как и в случае площади листьев (см. рис. 2).
Для расчета высоты растения PlantEye F500 использует распределение элементарных треугольников вдоль оси Z. Для этого сначала вычисля-ется гистограмма вдоль оси Z, которая отражает число элементарных треугольников на разной высоте над землей. Далее усредняются верхние 10 % высоты растения, а сама высота рассчитывается, как расстояние от высоты горшка до части, для которого проводили усреднение (рис. 5).
Из гистограммы видно, что местоположение растения в точках 1 и 3 давало максимальные значения высоты, в точках 2 и 4 — минимальные, в то же время расположение растения в центре сканируемой поверхности (точка 5) соответствовало среднему значению по всем пяти точкам. Следует учитывать, что погрешность полученных значений определятся тем, насколько глубоко располагается растение относительно края горшка, и может составлять от 1 до 5 см.
Параметр максимальная высота разработан для определения абсолютной наивысшей точки растения в миллиметрах. Этот показатель не заменяет текущий параметр высоты, а служит его дополнением. Текущая высота ориентирована на средние значения, а не на точность измерения, сводя к минимуму влияние внешних артефактов или суточных перемещений растений. Чтобы вычислить максимальную высоту, PlantEye F500 находит самую высокую область (группу точек в 3D-файле), которая содержит необходимое число точек и находится достаточно близко к другим областям. В этом домене наивысшая точка затем задается как максимальная высота (рис. 6).
В целом для высоты и максимальной высоты растений при 3D-сканировании прослеживается одинаковая зависимость изменения показателей для разного положения растения на сканируемой поверхности (для корреляции между этими двумя параметрами r = 0,99, р < 0,05). Разница между значениями высоты и максимальной высоты в нашем эксперименте составила около 12 мм во всех точках.
Наклон листа отражает информацию о том, насколько высоко расположены листья на растении и рассчитывается как общая площадь листьев, деленная на сумму проекций каждого элементарного треугольника на плоскость X-Y (рис. 7). Доверительный интервал по полученным значениям настолько маленький, что им можно пренебречь при пяти повторных измерениях у 10 растений. Максимальные значения наклона листьев были получены в точке 3, минимальные — в точках 2 и 4.
Угол наклона листьев представляет собой среднее арифметическое значение всех углов каждой грани на основе их нормали (рис. 8).
На представленной гистограмме видно, что расположение растения на сканируемой поверхности достоверно влияет на полученные значения. Так, местонахождение в точках 2 и 4 дает максимальные значения углов наклона, в точке 3 — минимальное, в точках 1 и 5 они наиболее близки к среднему по всем пятью точкам. При этом величина угла наклона была обратно пропорциональна наклону листьев (r = -0,99, р < 0,05) и площади листьев (r = -0,92, р < 0,05) при 3D-сканировании. Чем выше находятся листья над землей и более развернуты относительно сканирующего элемента, тем больше будет общая площадь листьев (без изменения прогнозируемой площади), и, как следствие, наклон листьев увеличится.
Глубина проникновения света отражает расстояние, на которое луч лазера может проникать сквозь листовую поверхность растения (рис. 9). Из представленных данных видно, что местонахождение растений в точках 1, 2 и 3 достоверно не влияло на глубину проникновения света, в точке 4 происходило незначительное уменьшение показателя, а в точке 5 мы отмечали наименьшую степень проник-новения лазерного луча. Таким образом можно с уверенностью констатировать, что расположение в точке 5 наиболее информативно. Величина проецируемой площади листа в различных местонахождениях растения на сканируемой поверхности обратно пропорциональна глубине проникно-вения света (r = -0,95, р < 0,05).
Таким образом, проведенное нами исследование показало, что расположение растения на сканируемой поверхности значительно влияет на значения морфологических параметров, измеряемых с помощью лазерного 3D-сканера PlantEye F500. Из восьми морфологических показателей два (высота растения и максимальная высота растения) дублируют друг друга. Более предпочтительна при анализе размеров растения его высота, поскольку при пяти измерениях высоты доверительный интервал был меньше. Величина площади листа и цифровой биомассы в разных точках расположения растений на сканируемой поверхности коррелируют (r = 0,98). Цифровая биомасса — менее информативный показатель при 3D-сканировании: он имеет большие значения доверительного интервала и зависит от архитектоники растения, необходимым условием определения этого параметра служит возможность расчета объема с учетом высоты и длины растения. Два показателя с обратной зависимостью (r = -0,95) — это проецируемая площадь листа и глубина проникновения лазерного луча. Глубина проникновения света могла бы быть интересным показателем, характеризующим густоту расположения побегов и листьев, однако при исследовании небольших растений (мы использовали растения, выращенные in vitro) эти признаки не имеют существенного значения. Величина угла наклона обратно пропорциональна наклону листьев (r = -0,99). Однако угол наклона листа — более информативный показатель при 3D-сканировании, позволяющий лучше понять архитектонику растения несмотря на то, что доверительный интервал наклона листьев практически равен нулю. В любом случае оба эти значения рассчитываются на основании средней величины всех наклонов листьев и углов наклонов, следовательно, снижение тургора побегов может существенно повлиять на полученные результаты.
Анализ публикаций и полученные нами результаты практического применения технологий машинного зрения при оценке морфологических параметров у растений сирени сорта Милосердие с использованием 3D-сканера PlantEye F500 позволили выделить следующие преимущества и недостатки автоматизированного подхода. К преимуществам следует отнести то, что платформы фенотипирования позволяют за одно сканирование определять от 5 до 15 морфологических характеристик на одном или сразу нескольких растениях (38-40). Проведение измерений морфологических параметров традиционными методами требует использования различных видов оборудования, а также значительных трудозатрат. Точность полученных значений различных морфологических параметров отличается высокой степенью сходимости (см. рис. 2-9) даже несмотря на имеющиеся погрешности измерений. Полученные данные загружаются в компьютер, и существует возможность оценки динамики изменений морфологических параметров во времени. Недостатками следует считать отсутствие протоколов проведения феноскрининга морфологических параметров для разных культур (41). Следует учитывать, что при одновременном изучении нескольких растений на сканируемой поверхности увеличивается вероятность ошибки измерений, на что указывают полученные нами экспериментальные данные. Невозможность использования установки в полевых условиях при проведении экспериментов, внешние факторы (ветер) мешают растению оставаться в статичном положении и, как следствие, влияют на точность полученных данных (36). Большая часть исследуемых методом 3 D-сканирования морфометрических параметров находятся в сильной положительной либо отрицательной взаимозависимости и дублируют друг друга, например высота и максимальная высота, цифровая биомасса и площадь листа, проецируемая площадь листа и глубина проникновения света. То есть разработчикам программного обеспечения необходимо сконцентрироваться не на числе выдаваемых параметров, а на их информативности при оценке состояния растений и возможности определения динамики ростовых процессов. при использовании различных форм препаратов. Исходя из того, что при смене положения одного и того же растения на сканируемой поверхности (перемещение по пяти точкам) регистрируемые морфологические показатели значительно различались, можно с уверенностью ожидать, что расположение нескольких растений на сканируемой поверхности приведет к значительным различиям полученных данных.
Итак, наши эксперименты показали, что для получения объективных и сопоставимых результатов при использовании 3D-сканера PlantEye F500 оптимально располагать растения в центре сканируемой поверхности в одном и том же положении, а в качестве морфологических параметров для идентификации сортов и фиксации приростов можно рекомендовать использование параметров площадь листа, проекционная площадь листа, высота, угол наклона листа. Для каждого вида растений необходимо проводить первичные морфологические исследования традиционными методами, а затем сравнивать полученные данные с результатами сканирования, чтобы внести поправочные коэффициенты и подтвердить информативность используемого набора признаков, тем самым повышая точность данных, полученных в результате использования технологии машинного зрения.

Рис. 1. Точки местонахождения растения сирени (Syringa vulgaris L.) сорта Милосердие на сканируемой поверхности (А) и триангулирование точек для построения 3D-облака (Б) при фенотипировании 3D-сканированием (PlantEye F500, «Phenospex B.V.», Нидерланды).
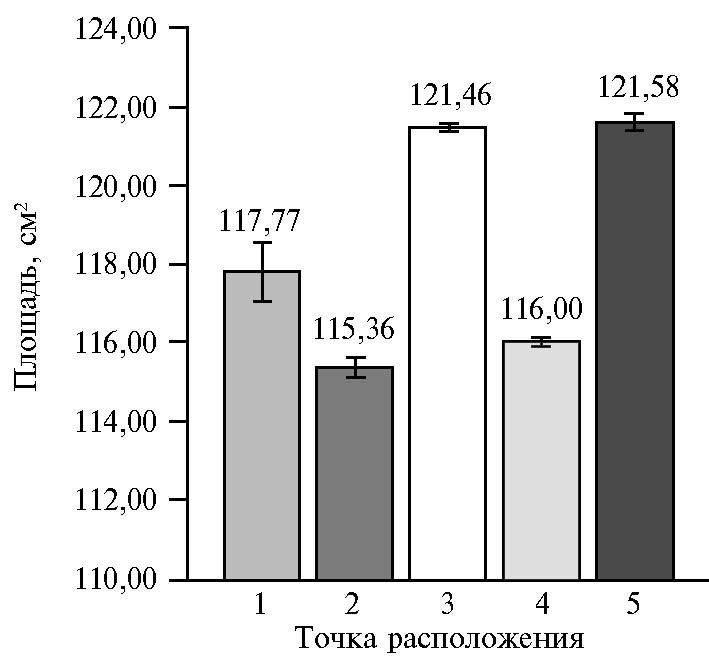
Рис. 2. Площадь листьев в зависимости от местоположения растения сирени (Syringa vulgaris L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
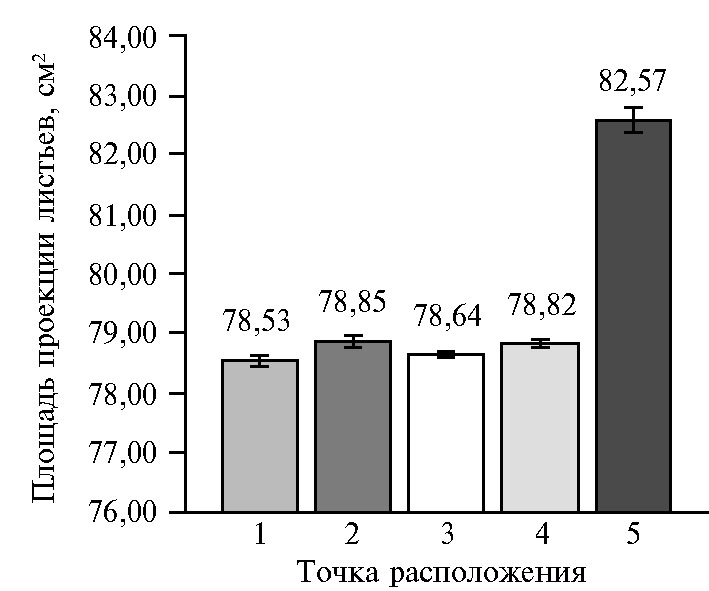
Рис. 3. Площадь проекции листьев в зависимости от местоположения растения сирени (Syringa vul-garis L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
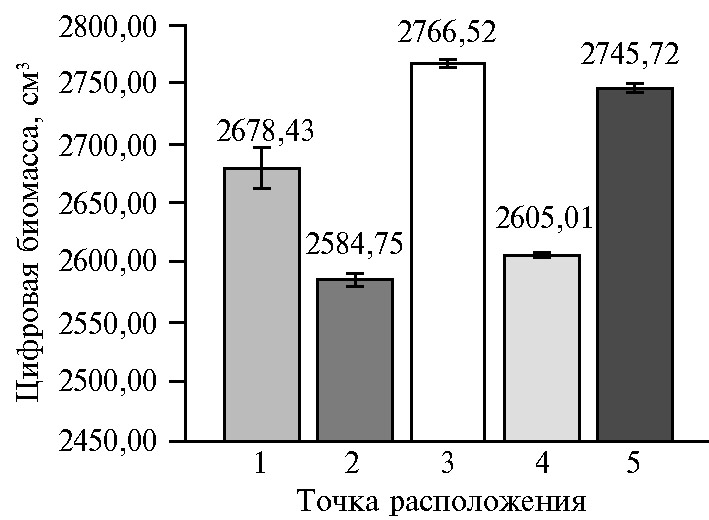
Рис. 4. Цифровая биомасса листьев в зависимости от местоположения растения сирени (Syringa vulga-ris L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
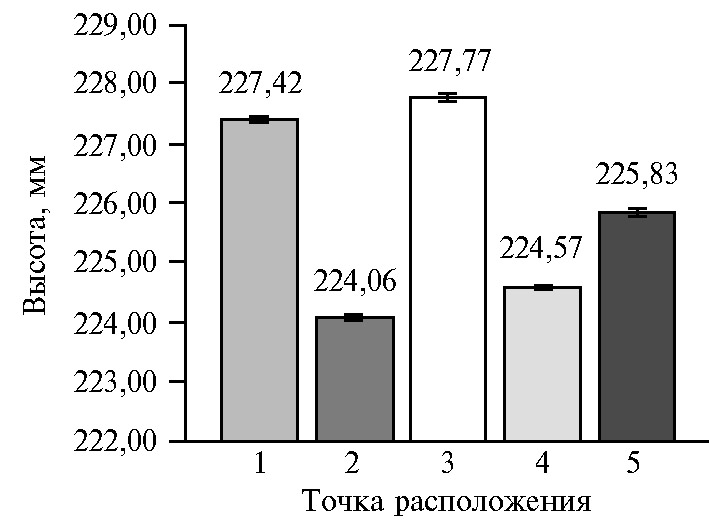
Рис. 5. Высота растения сирени (Syringa vulgaris L.) сорта Милосердие в зависимости от его местоположения на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phe-nospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
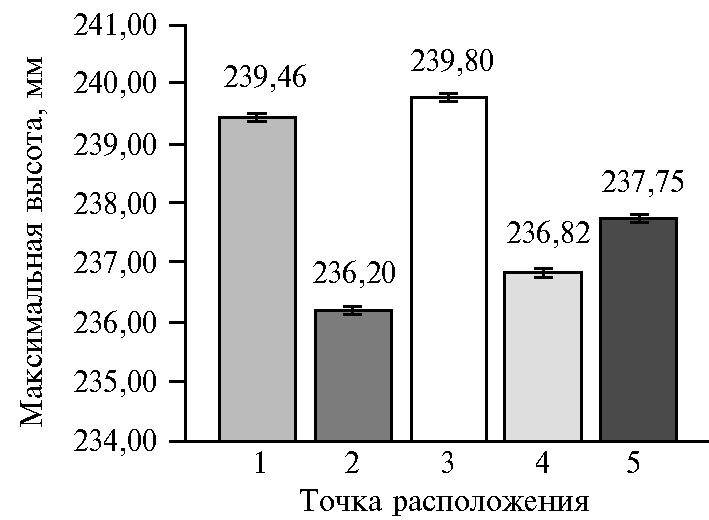
Рис. 6. Максимальная высота растения сирени (Syringa vulgaris L.) сорта Милосердие в зависимости от его местоположения на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
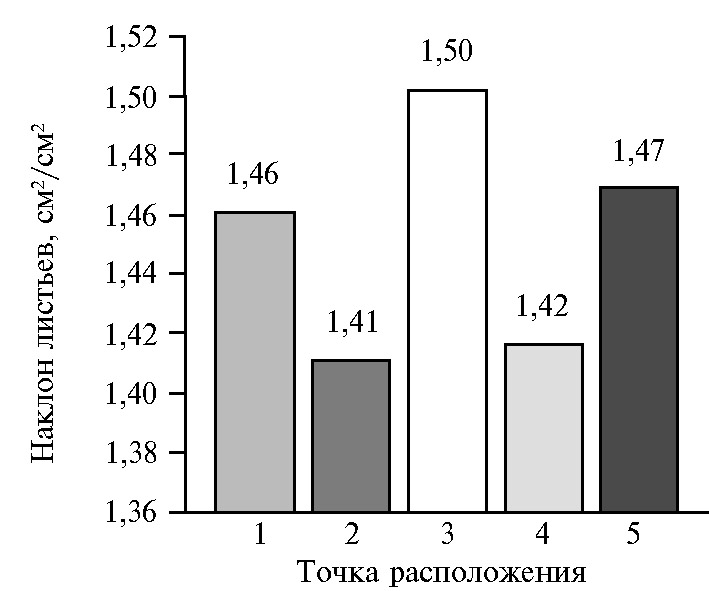
Рис. 7. Наклон листьев в зависимости от местоположения растений сирени (Syringa vulgaris L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
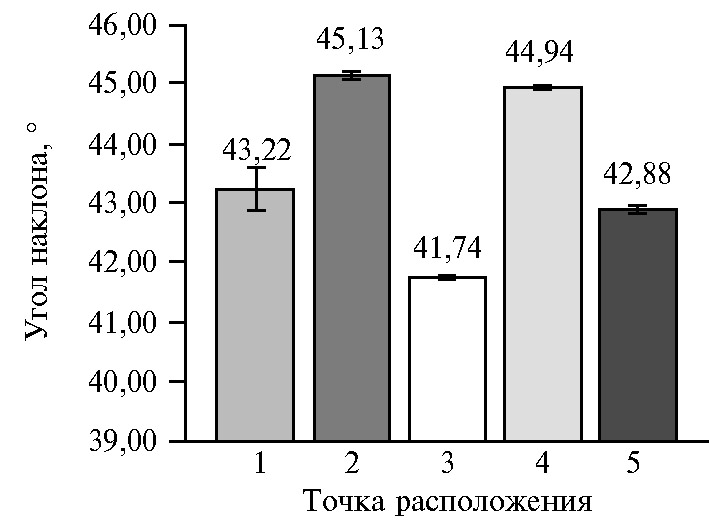
Рис. 8. Угол наклона листьев в зависимости от местоположения растений сирени (Syringa vulgaris L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, Plant-Eye F500, «Phenospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
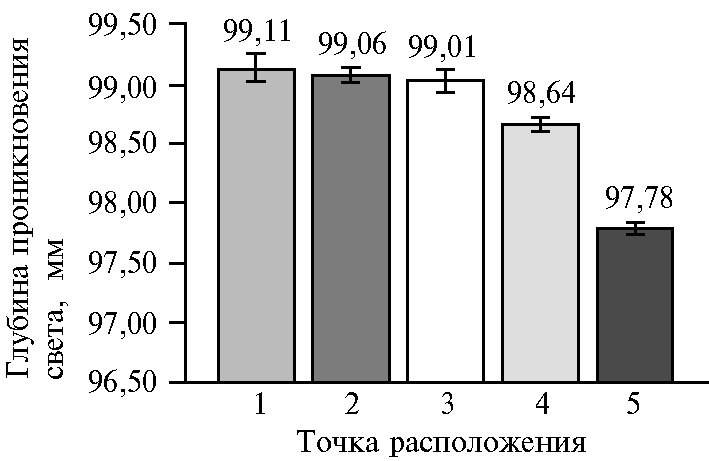
Рис. 9. Глубина проникновения света сквозь листовую поверхность в зависимости от местоположения растений сирени (Syringa vulgaris L.) сорта Милосердие на сканируемой поверхности (n = 10, M±CI, р = 0,05; 3D-сканирование, PlantEye F500, «Phe-nospex B.V.», Нидерланды). Каждое растение располагали в 5 точках сканируемой поверхности и в одном и том же положении проводили не менее 5 повторных сканирований.
ЛИТЕРАТУРА
- Kumar J., Rai K.M. Research advances in plant genomics. All Life, 2021, 11: 1313 (doi: 10.3390/life11121313).
- Bennett M.D., Leitch I.J. Plant genome size research: a field in focus. Annals of Botany, 2005, 95(1): 1-6 (doi: 10.1093/aob/mci001).
- Генаев М.А., Шмаков Н.А., Мустафин З.С., Мухин А.М., Константинов Д.К., Дорошков А.В., Лашин С.А., Афонников Д.А. Поиск новых генов в «скрытой» части транскриптомов сельскохозяйственных растений. VII Съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы. Сборник тезисов Международного конгресса. СПб, 2019: 528.
- Кирьянова О.Ю., Кулуев Б.Р., Кулуев А.Р., Марданшин И.С., Губайдуллин И.М., Чемерис А.В. Мультиплексный IN SILICO RAPD-анализ ряда родственных растений с отличающимися размерами геномов и перспективы такого подхода для ДНК-паспортизации сортов сельскохозяйственных растений. Биомика, 2020, 12(2): 194-210 (doi: 10.31301/2221-6197.bmcs.2020-10).
- Дьяченко Е.А., Кулакова А.В., Кочиева Е.З., Щенникова А.В. Вариабельность геномных RGA-локусов современных отечественных сортов картофеля: данные NBS-маркирования. Сельскохозяйственная биология, 2021, 56(1): 32-43 (doi: 10.15389/agrobiology.2021.1.32rus).
- Пономарева М.Л., Пономарев С.Н., Тагиров М.Ш., Гильмуллина Л.Ф., Маннапова Г.С. Генотипическая изменчивость содержания пентозанов в зерне озимой ржи. Сельскохозяйственная биология, 2017, 52(5): 1041-1048 (doi: 10.15389/agrobiology.2017.5.1041rus).
- Рогозина Е.В., Терентьева Е.В., Потокина Е.К., Юркина Е.Н., Никулин А.В., Алексеев Я.И. Идентификация родительских форм для селекции картофеля, устойчивого к болезням и вредителям, методом мультиплексного ПЦР-анализа. Сельскохозяйственная биология, 2019, 54(1): 19-30 (doi: 10.15389/agrobiology.2019.1.19rus).
- Nakanwagi M.J., Sseremba G., Kabod N.P., Masanza M., Kizito E.B. Identification of growth stage-specific watering thresholds for drought screening in Solanum aethiopicum Shum. Scientific Reports, 2020, 10: 862 (doi: 10.1038/s41598-020-58035-1).
- Robles P., Quesada V. Organelle genetics in plants. International Journal of Molecular Science, 2021, 22(4): 2104 (doi: 10.3390/ijms22042104).
- Heslop-Harrison J.S.P., Schwarzacher T. Organisation of the plant genome in chromosomes. The Plant Journal, 2011. 66(1): 18-33 (doi: 10.1111/j.1365-313X.2011.04544.x).
- Белоногова Н.М. «Прямая» и «обратная» генетика. генетика количественных признаков. Вавиловский журнал генетики и селекции, 2014, 18(1): 147-157.
- Endalkachew A. Review on forward and reverse genetics in plant breeding. All Life, 2021, 14(1): 127-135 (doi: 10.1080/26895293.2021.1888810).
- Gilchrist E., Haughn G. Reverse genetics techniques: engineering loss and gain of gene function in plants. Briefings in Functional Genomics, 2010, 9(2): 103-110 (doi: 10.1093/bfgp/elp059).
- Lyu J. Reverse genetics: wheat solution. Nature Plants, 2017, 3: 17005 (doi: 10.1038/nplants.2017.5).
- Holubová K., Hensel G., Vojta P., Tarkowski P., Bergougnoux V., Galuszka P. Modification of Barley plant productivity through regulation of cytokinin content by reverse-genetics approaches. Frontiers in Plant Science, 2018, 27(9): 1676 (doi: 10.3389/fpls.2018.01676).
- Новохатин В.В., Драгавцев В.А., Леонова Т.А., Шеломенцева Т.В. Создание сорта мягкой яровой пшеницы Гренада с помощью инновационных технологий селекции на основе теории эколого-генетической организации количественных признаков. Сельскохозяйственная биология, 2019, 54(5): 905-919 (doi: 10.15389/agrobiology.2019.5.905rus).
- Crossa J., Fritsche-Neto R., Montesinos-Lopez Osval A., Costa-Neto G., Dreisigacker S., Montesinos-Lopez A., Bentley Alison R. The modern plant breeding triangle: optimizing the use of gGenomics, phenomics, and enviromics data. Frontiers in Plant Science, 2021, 12: 651480 (doi: 10.3389/fpls.2021.651480).
- Хлесткина Е.К., Хлесткин В.К. Современные подходы прямой и обратной генетики картофеля. Мат. межд. науч.-практ. конф. «Картофелеводство». М., 2017: 59-64.
- Тохтарь В.К., Мазур Н.В. Изучение морфологических признаков популяций Conyza canadensis (L.) Cronq. на юго-западе Среднерусской возвышенности. Научные ведомости Белгородского государственного университета. Серия: Естественные науки, 2011, 15/1(104): 249-253.
- Furbank R.T., Tester M. Phenomics — technologies to relieve the phenotyping bottleneck. Trends in Plant Science, 2011, 16(12): 635-644 (doi: 10.1016/j.tplants.2011.09.005).
- Li L., Zhang Q., Huang D. A review of imaging techniques for plant phenotyping. Sensors, 2014, 14(11): 20078-20111 (doi: 10.3390/s141120078).
- Афонников Д.А., Генаев М.А., Дорошков А.В., Комышев Е. Г., Пшеничникова Т.А. Методы высокопроизводительного фенотипирования растений для массовых селекционно-генетических экспериментов. Генетика, 2016, 52(7): 788-803 (doi: 10.7868/S001667581607002X).
- Серегин А.П., Бочков Д.А., Шнер Ю.В., Гарин Э.В., Майоров С.Р., Голяков П.В., Большаков Б.В., Прохоров В.Е., Маллалиев М.М., Виноградов Г.М., Эбель А.Л., Каширина Е.С., Бирюкова О.В., Курякова О.П., Мирвода С.В., Химин А.Н., Муртазалиев Р.А., Зеленкова В.Н., Дудов С.В., Горбунова М.С. и др. «Флора России» на платформе Inaturalist: большие данные о биоразнообразии большой страны. Журнал общей биологии, 2020, 81(3): 223-233 (doi: 10.31857/S0044459620030070).
- Светашева Т.Ю., Лакомов А.Ф., Привалова М.В., Смирнова Е.В., Максимова Т.В. Международная интернет-платформа Inaturalist как база наблюдений растений красной книги тульской области. Фиторазнообразие Восточной Европы, 2020, 14(4): 549-559 (doi: 10.24411/2072-8816-2020-10088).
- Liu H., Bruning B., Garnett T., Berger B. Hyperspectral imaging and 3D technologies for plant phenotyping: From satellite to close-range sensing. Computers and Electronics in Agriculture, 2020, 175: 105621 (doi: 10.1016/j.compag.2020.105621).
- Reeb R.A., Aziz N., Lapp S.M., Kitzes J., Heberling J.M., Kuebbing S.E. Using convolutional neural networks to efficiently extract immense phenological data from community science images. Frontiers in Plant Science, 2022, 12: 787407 (doi: 10.3389/fpls.2021.787407).
- Gibbs J.A., Pound M., French A.P., Wells D.M., Murchie E., Pridmore T. Plant phenotyping: an active vision cell for three-dimensional plant shoot reconstruction. Plant Physiology, 2018, 178(2): 524-534 (doi: 10.1104/pp.18.00664).
- Tomé F., Jansseune K., Saey B., Grundy J., Vandenbroucke K., Hannah M.A., Redestig H. rosettR: protocol and software for seedling area and growth analysis. Plant Methods, 2017, 13: 13 (doi: 10.1186/s13007-017-0163-9).
- Tsaftaris S.A., Minervini M., Scharr H. Machine learning for plant phenotyping needs image processing. Trends in Plant Science, 2016, 21(12): 989-991 (doi: 10.1016/j.tplants.2016.10.002).
- Ubbens J.R., Stavness I. Deep plant phenomics: a deep learning platform for complex plant phenotyping tasks. Frontiers in Plant Science, 2017, 8: 1190 (doi: 10.3389/fpls.2017.01190).
- Coppens F., Wuyts N., Inzé D., Dhondt S. Unlocking the potential of plant phenotyping data through integration and data-driven approaches. Current Opinion in Systems Biology, 2017, 4: 58-63 (doi: 10.1016/j.coisb.2017.07.002).
- Fasoula D.A., Ioannides I.M., Omirou M. Phenotyping and plant breeding: overcoming the barriers. Frontiers in Plant Science, 2020, 10: 1713 (doi: 10.3389/fpls.2019.01713).
- Carvalho L.C., Gonçalves E.F., da Silva J.M., Miguel J. Potential phenotyping methodologies to assess inter- and intravarietal variability and to select grapevine genotypes tolerant to abiotic stress. Frontiers in Plant Science, 2021, 12: 71820226 (doi: 10.3389/fpls.2021.718202).
- Naik H.S., Zhang J., Lofquist A., Assefa T., Sarkar S., Ackerman D., Singh A., Singh A.K., Ganapathysubramanian B. A real-time phenotyping framework using machine learning for plant stress severity rating in soybean. Plant Methods, 2017, 13: 23 (doi: 10.1186/s13007-017-0173-7).
- Wang Y., Wen W., Wu S., Wang C., Yu Z., Guo X., Zhao C. Maize plant phenotyping: comparing 3D laser scanning, multi-view stereo reconstruction, and 3D digitizing estimates. Remote Sensing, 2018, 11(1): 63 (doi: 10.3390/rs11010063).
- Aleynikov Y.G., Konstantinovich A.V. Creation of 3D cloud models for plants using a scanner and walking machine with dynamic stability. Bioscience Biotechnology Research Communications, 2021, 14(2): 505-508 (doi: 10.21786/bbrc/14.2/1).
- Paulus S. Measuring crops in 3D: using geometry for plant phenotyping. Plant Methods, 2019, 15: 103 (doi: 10.1186/s13007-019-0490-0).
- Yang W., Guo Z., Huang C., Duan L., Chen G., Jiang N., Fang W., Feng H., Xie W., Lian X., Wang G., Luo Q., Zhang Q., Liu Q., Xiong L. Combining high-throughput phenotyping and genome-wide association studies to reveal natural genetic variation in rice. Nature Communications, 2014. 5: 5087 (doi: 10.1038/ncomms6087).
- Klukas C., Chen D., Pape J.M. Integrated analysis platform: an open-source information system for highthroughput plant phenotyping. Plant Physiology, 2014, 165(2): 506-518 (doi: 10.1104/pp.113.233932).
- Chen D., Neumann K., Friedel S., Kilian B., Chen M., Altmann T., Klukas C. Dissecting the phenotypic components of crop plant growth and drought responses based on high-throughput image analysis. The Plant Cell, 2014, 26(12): 4636-4655 (doi: 10.1105/tpc.114.129601).
- Бондаренко В.Ю., Барковский А.В., Шашко А.Ю., Черныш М.А., Пржевальская Д.А., Колбанов Д.В., Соколик А.И., Смолич И.И., Медведев С.С., Демидчик В.В. Анализ фенотипа декоративных растений с использованием искусственных нейронных сетей: определение таксономических и физиологических характеристик. Журнал Белорусского государственного университета. Биология, 2019, 1: 25-32 (doi: 10.33581/2521-1722-2019-1-25-32).