doi: 10.15389/agrobiology.2022.5.954rus
УДК 635.64:631.52:577.21
Работа выполнена в рамках Государственного задания № 0431-2022-0004.
ОСОБЕННОСТИ ПЕРВИЧНОЙ СТРУКТУРЫ ГЕНА Ph-3, ВЫЯВЛЕННЫЕ ПРИ СОЗДАНИИ НОВОГО МАРКЕРА УСТОЙЧИВОСТИ ТОМАТА К ФИТОФТОРОЗУ
В.В. МАРТЫНОВ1 ✉, Е.Г. КОЗАРЬ2, И.А. ЕНГАЛЫЧЕВА2
Фитофтороз, вызываемый оомицетом Phytophthora infestans (Mont.) de Bary, — одно из самых вредоносных заболеваний томатов. Наиболее перспективным методом борьбы с ним остается выведение устойчивых сортов, при создании которых широко используется интрогрессия генов устойчивости из дикорастущих родственных видов. В частности, несколько генов устойчивости к фитофторозу, идентифицированных у дикого вида томата Solanum pimpinellifolium, были интрогрессированы в культурные сорта. Наиболее сильным геном считается Ph-3, поскольку он обеспечивает устойчивость к множеству изолятов P. infestans. На сегодняшний день известны ДНК-маркеры, так или иначе ассоциированные с этим геном. Однако в геноме томата были обнаружены гомологи этого гена, которые не обладают функциональной активностью. В настоящей работе впервые показано, что в сортах томата отечественной селекции при наличии гена Ph-3 отсутствуют другие его гомологи. Также впервые установлено, что в последовательности гена Ph-3присутствует вставка ретротранспозона, которая может приводить к потере геном своей функциональной активности. Нашей целью было создание простого в использовании высокоспецифичного ДНК-маркера гена Ph-3, с помощью которого можно отличить Ph-3 от его структурных гомологов, и валидация этого маркера в сравнении с уже известными маркерами на основе анализа коллекции отечественных сортов и линий томата и оценки связи маркеров с полевой устойчивостью к фитофторозу. В работе использовали 24 образца томата (Solanum lycopersicum L.). Исследования проводили в 2021 году на опытном поле ФГБНУ ФНЦО (Московская обл., Одинцовский р-н). Рассаду высаживали в грунт в I декаде июня. Поражение фитофторозом учитывали в динамике через каждые 7 сут, начиная с появления первых симптомов (III декада июля). Тотальную ДНК выделяли из молодых листьев 2-недельных растений при помощи набора реагентов Сорб-ГМО-Б («Синтол», Россия). Дизайн праймеров для специфичной амплификации гена Ph-3 осуществляли на основе множественного выравнивания нуклеотидной последовательности гена Ph-3 (GenBank no. KJ563933) и его структурных гомологов SlRGA1, SlRGA2, SlRGA3 и SlRGA4. Были подобраны праймеры, амплифицирующие фрагмент гена Ph-3 размером 412 п.н.: прямой 5′-AATATTGAAAATAGCTGCACTGA-3′ и обратный 5′-CGAGATTTGGAGGGAATGTAA-3′. Созданный маркер получил название Ph3-412. Кроме того, для сравнительного анализа использовали праймеры маркера NC-LB-9-6678, амплифицирующие фрагменты размером 601 и 907 п.н.: 5′-CCTTAATGCAATAGGCAAAT-3′ и 5′-ATTT-GAATGTTCTGGATTGG-3′, последовательности которых абсолютно консервативны для гена Ph-3 и его гомологов. Для определения нуклеотидных последовательностей полученных ампликонов их клонировали в вектор pAL-TA («Евроген», Россия), которым трансформировали компетентные клетки Escherichia coli DH5a, и секвенировали по методу Сэнгера. Осуществляли множественное выравнивание нуклеотидных последовательностей с последующим анализом результатов выравнивания. Для построения дендрограммы использовали программу TREECON (http://bioinfor-matics.psb.ugent.be/software/details/Treecon). Производные аминокислотные последовательности были получены с помощью программы EditSeq (https://macdownload.informer.com/editseq/down-load/). Для поиска гомологовполученных последовательностей в базе данных NCBI использовали программу BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). При сравнении результатов молекулярного анализа с данными фенотипической оценки полевой устойчивости к фитофторозу ни один из маркеров не показал однозначной связи с полевой устойчивостью. Мы подтвердили, что амплифицируемый с помощью праймеров Ph3-412 фрагмент принадлежит гену Ph-3, в то время как фрагмент размером 601 п.н., который получают с праймерами NC-LB-9-6678, соответствует гомологу SlRGA4. Показано, что фрагмент размером 907 п.н., полученный с теми же праймерами, гомологичен гену Ph-3, но при этом содержит вставку LTR ретротранспозона семейства Ty1-copia размером 306 п.н. У всех сортов, у которых был обнаружен ген Ph-3, он содержал вышеуказанную вставку. Наличие такой вставки может приводить к потере функциональной активности, что необходимо учитывать при маркировании гена Ph-3. Исходя из этого, набольшую селекционную ценность представляют генотипы, у которых ген Ph-3 не имеет вставки ретротранспозона.
Ключевые слова: томаты, фитофтороз, ген Ph-3, ДНК-маркеры, гены устойчивости.
Фитофтороз, вызываемый оомицетом Phytophthora infestans (Mont.) de Bary, — одно из самых вредоносных заболеваний томатов, которое может уничтожать до 100 % урожая (1). Борьба с фитофторозом остается сложной задачей из-за высокой генетической изменчивости и сложного расового состава P. infestans. Применение фунгицидов, в частности металаксила, недостаточно эффективно, поскольку расы патогена быстро мутируют и приобретают устойчивость к этому препарату (2-6). Кроме того, обработка фунгицидами — дорогостоящее мероприятие, а сами фунгициды наносят вред окружающей среде и опасны для здоровья человека. Наиболее перспективный метод борьбы с фитофторозом — выведение устойчивых сортов томата.
При создании устойчивых сортов широко используется интрогрессия генетического материала из дикорастущих родственных видов с целью пирамидирования имеющихся у них генов устойчивости (7-9). В частности, несколько генов устойчивости к фитофторозу, идентифицированных у дикого вида томата Solanum pimpinellifolium, были интрогрессированы в культурные сорта (10, 11). Прежде всего это гены Ph-1, Ph-2 и Ph-3. Ген Ph-1, картированный на 7-й хромосоме, придает устойчивость к расе T0 P. infestans (12). Ген Ph-2, первоначально идентифицированный у S. pimpinellifolium West Virginia 700 (WV700) на 10-й хромосоме, обеспечивает устойчивость к расе T0 и частичную устойчивость к расе T1 (13). Однако устойчивость, определяемая этими генами, быстро преодолевается новыми расами P. in-festans. Ген Ph-3 был идентифицирован в образце L3708 S. pimpinellifolium и картирован на длинном плече 9-й хромосомы. Он обеспечивает устойчивость ко многим расам P. infestans, которые преодолевают устойчивость, придаваемую генами Ph-1 и Ph-2 (14). Также было показано, что Ph-2 и Ph-3 действуют синергически и вместе определяют устойчивость к более широкому кругу изолятов патогенов, чем каждый ген в отдельности (15).
В настоящее время наиболее сильным геном устойчивости к фитофторозу томата считается ген Ph-3. На его изучение направлены немалые усилия с целью включения в селекционные программы и введения в новые коммерческие сорта и линии (16). Ген Ph-3 был клонирован и охарактеризован на молекулярном уровне у образца L3708 S. pimpinellifolium. Установлено, что Ph-3 кодирует белок, содержащий домен типа «суперспираль», связывающий нуклеотиды домен и богатые лейцином повторы (CC-NBS-LRR). При трансгенозе он может придавать восприимчивому сорту томата устойчивость к фитофторозу (17). При этом в геноме томата сорта Heinz1706 в локусе, соответствующем локусу Ph-3 S. pimpinellifolium, были обнаружены четыре структурных гомолога этого гена — SlRGA1, SlRGA2, SlRGA3 и SlRGA4 (17).
Также предпринимаются попытки маркировать ген Ph-3 для более эффективного переноса в представляющий селекционный интерес материал с помощью маркер-опосредованной селекции (marker assistant selection, MAS) и пирамидирования с другими генами устойчивости к фитофторозу. На сегодняшний день известно несколько ДНК-маркеров, так или иначе ассоциированных с этим геном, в том числе три SCAR (sequence characterised amplified region) маркера (18-20) и один CAPS (cleaved amplified polymorphic sequences) маркер (21). Однако эти маркеры обладают рядом недостатков.
В частности, с помощью SCAR маркеров, описанных Y. Park с соавт. (18), можно различить гомологи генаPh-3, но не сам ген. Кроме того, анализ предлагается проводить с использованием набора из трех пар праймеров, что увеличивает затраты труда и времени и затрудняет интерпретацию полученных результатов. Маркер, описанный H.T.H. Truong с соавт. (19), был получен из RAPD маркера, и его связь с последовательностью гена Ph-3 неизвестна. Анализ множественного выравнивания нуклеотидных последовательностей гена Ph-3 и его известных гомологов показал, что праймеры, используемые D.R. Panthee с соавт. (20) для амплификации этого гена, находятся в консервативных областях упомянутых последовательностей и с их помощью невозможно специфично амплифицировать ген. CAPS маркер, описанный Y.-Y. Wang с соавт. (21), предполагает использование эндонуклеаз рестрикции и разделение продуктов рестрикции в полиакриламидном геле, что делает этот анализ относительно дорогим и трудоемким. Также в случае CAPS маркеров могут возникать проблемы с воспроизводимостью результатов, поскольку на эффективность рестрикции влияет активность фермента и количество ДНК, а эти параметры трудно четко контролировать. Кроме того, валидация всех этих маркеров проводилась на сегрегирующих популяциях, полученных в результате скрещивания восприимчивой и устойчивой родительских форм.
В настоящей работе впервые показано, что в сортах томата отечественной селекции при наличии гена Ph-3 отсутствуют другие его гомологи. Также впервые установлено, что в последовательности гена Ph-3 присутствует вставка ретротранспозона, которая может приводить к потере геном своей функциональной активности.
Нашей целью было создание простого в использовании высокоспецифичного ДНК-маркера гена Ph-3, с помощью которого можно отличить Ph-3 от его структурных гомологов, и валидация этого маркера в сравнении с уже известными маркерами на основе анализа коллекции отечественных сортов и линий томата и оценки связи маркеров с полевой устойчивостью к фитофторозу.
Методика. Исследование выполняли на 24 образцах томата (Solanum lycopersicum L.) селекции Федерального научного центра овощеводства (ФГБНУ ФНЦО), внесенных в Государственный реестр селекционных достижений, допущенных к использованию на территории Российской Федерации: Оттава 30 (стандарт устойчивости к фитофторозу), Талалихин (стандарт восприимчивости к фитофторозу), Фитилек, Приморец, Грот, Чаровница, Лотос, Росинка, Посьет, Топтыжка, Одиссей, Патрокл, Благодатный, Викинг, Дубок, Реванш, Талисман, Монах, Перст, Камея, Северянка и Восход ВНИИССОКа, линии л-ДВot30-2/19 и л-Ft5/20. Тридцать растений каждого сорта выращивали в условиях лаборатории до 4-недельного возраста при температуре 23-25 °C, влажности воздуха 70-80 % и 16-часовом искусственном освещении (с 700 до 2300), после чего пересаживали в открытый грунт.
Эксперименты проводили в 2021 году на опытном поле ФГБНУ ФНЦО (Московская обл., Одинцовский р-н) на провокационном инфекционном фоне (изолированный участок с монокультурой томата). Рассаду высаживали в грунт в I декаде июня в фазу 4-5 настоящих листьев с применением двухстрочной посадки по схеме 70×40 см для детерминантных сортов, 70×35 см — для штамбовых. Агротехника выращивания рассады была стандартной для культуры томата.
Для фенотипической оценки томата на устойчивость к P. infestans высаживали по 10 растений каждого образца в 3-кратной повторности по схеме рандомизированных блоков так, чтобы каждый образец имел одинаковые шансы на заражение. Для контроля выравненности инфекционного фона и динамики развития болезни использовали восприимчивый сорт Талалихин и устойчивый сорт Оттава 30, которые высаживали через пять изучаемых образцов. Учет поражения фитофторозом проводили в динамике через каждые 7 сут, начиная с появления первых симптомов (III декада июля). Поражение растений оценивали методом визуальной диагностики по характерным симптомам на листьях и плодах по модифицированной десятибалльной шкале, где 0 — нет симптомов; 0,1 — поражено 1-5 % площади листа, мелкие поражения (< 2 мм), отсутствие поражений стебля; 0,5 — поражено 6-10 % площади листа, отсутствие повреждений стеблей; 1 — поражено 11-20 % площади листа, отсутствие повреждений стеблей; 1,5 — поражено 21-30 % площади листа, сливающиеся поражения листьев или крошечные водянистые поражения стеблей; 2 — поражено 31-40 % площади листьев, расширяющиеся по краям поражения листьев или несколько небольших стеблевых поражений (< 5 мм); 2,5 — поражено 41-50 % площади листьев, стеблевые поражения (< 30 мм); 3 — поражено 51-60 % площади листьев, засыхающие повреждения листьев или поражения стебля с расширением краев, 20 % пораженных плодов; 3,5 — поражено 61-70 % площади листьев, засыхающие повреждения листьев и поражения стебля с расширением краев, 40 % пораженных плодов; 4 — поражено 71-100 % площади листьев, стеблей и плодов. Устойчивость каждого образца оценивали по индексу поражения (I, средний балл). По совокупности всех оценок образцы дифференцировали на группы устойчивости: У — устойчивые (I = 0), ОУ — относительно устойчивые (0 < I ≤ 1), СВ — средневосприимчивые (1 < I ≤ 2), В — восприимчивые (2 < I ≤ 3), ВВ — высоковосприимчивые (I > 3).
Тотальную ДНК выделяли из молодых листьев 2-недельных растений при помощи набора реагентов Сорб-ГМО-Б («Синтол», Россия) по протоколу фирмы-производителя. Для каждого сорта ДНК выделяли из всех 8 растений, после чего препараты ДНК объединяли в одну общую пробу.
Дизайн праймеров для специфичной амплификации гена Ph-3 осуществляли на основе множественного выравнивания нуклеотидной последовательности гена Ph-3 (GenBank no. KJ563933) и его структурных гомологов SlRGA1, SlRGA2, SlRGA3 и SlRGA4, нуклеотидные последовательности которых были взяты из нуклеотидной последовательности 9-й хромосомы томата, зарегистрированной в базе данных GenBank NCBI (номер EF647605.1). Были подобраны прямой (5'-AATATTGAAAATAGCTGCACTGA-3') и обратный (5'-CGAGATTTGGAGGGAATGTAA-3') праймеры, у которых последовательности 3'-концов были строго специфичны для гена Ph-3 и отличались от последовательностей гомологов гена в этом положении. Указанные праймеры располагались в LRR-домене гена Ph-3, и ожидаемый размер ампликона составлял 412 п.н. Кроме того, для сравнительного анализа использовали праймеры маркера NC-LB-9-6678 5'-CCTTAATGCA-ATAGGCAAAT-3' и 5'-ATTTGAATGTTCTGGATTGG-3' (11), последовательности которых были абсолютно консервативны для гена Ph-3 и его гомологов.
Программа для амплификации была следующей: 3 мин при 94 °С; 30 с при 94 °С, 30 с при 60 °С, 1 мин при 72 °С (35 циклов); 5 мин при 72 °С (финальный синтез). Объем реакционной смеси составлял 25 мкл. На одну реакцию брали 50 нг тотальной ДНК. Для амплификации использовали прибор GeneAmp PCR System 2700 («Applied Biosystems, Inc.», США). Электрофоретическое разделение продуктов амплификации проводили в 1 % агарозном геле в 1× TAE буфере. Условия амплификации с праймерами NC-LB-9-6678 были такими, как описано в работе D.R. Panthee с соавт. (20): 3 мин при 92 °С; 30 с при 92 °С, 1 мин при 52 °С, 30 с при 72 °С (35 циклов); 8 мин при 72 °С (финальный синтез).
Для определения нуклеотидных последовательностей полученных ампликонов их клонировали в вектор pAL-TA («Евроген», Россия), которым трансформировали компетентные клетки Escherichia coli DH5a, и секвенировали по методу Сэнгера с помощью набора реактивов Big Dye Terminator v.3.1 («Applied Biosystems, Inc.», США) на автоматическом секвенаторе ABI PRIZM 3730 («Applied Biosystems, Inc.», США) согласно инструкциям производителя.
Множественное выравнивание нуклеотидных последовательностей осуществляли в программе Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clust-alo/), с последующим анализом результатов выравнивания при помощи программы GeneDoc 2.7 (https://genedoc.software.informer.com/2.7/). Для построения дендрограммы использовали программу TREECON (22). Производные аминокислотные последовательности были получены с помощью программы EditSeq (https://macdownload.informer.com/editseq/download/). Для поиска гомологов полученных последовательностей в базе данных NCBI использовали программу BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Результаты. При ПЦР-амплификации препаратов тотальной ДНК, выделенных из 24 образцов томата, с праймерами, специфичными к гену Ph-3, созданный нами маркер Ph3-412 был обнаружен у всех проанализированных образцов, кроме трех (л-Ft5-19, Викинг и Реванш) (рис. 1, А). При этом по результатам анализа с праймерами маркера NC-LB-9-6678 три указанных образца имели маркер, который в работе H.L. Merk с соавт. (11) был связан с устойчивостью к фитофторозу томата (600 п.н.), а остальные — маркер, связанный с восприимчивостью (900 п.н.) (см. рис. 1, Б). Иными словами, маркер Ph3-412 отсутствовал у сортов, у которых был обнаружен ранее известный маркер, связанный с устойчивостью, и присутствовал у сортов, в которых был выявлен фрагмент размером 900 п.н., связанный с восприимчивостью к фитофторозу. То есть анализ с новым и ранее известным маркером дал диаметрально противоположные результаты.
Погодные условия 2021 года характеризовались жарким и засушливым вегетационным периодом: осадков за весь период выпало на 29,4 мм меньше среднемноголетних значений, температура воздуха была на 2,5 °С выше климатической нормы. Учет поражения сортов-дифференциаторов оомицетом P. infestans показал наличие расы Т1, характеризующейся высокой агрессивностью и вирулентностью в условиях открытого грунта 2021 года.
При сравнении результатов молекулярного анализа с данными фенотипической оценки полевой устойчивости к фитофторозу установили, что у стандарта устойчивости сорта Оттава 30 присутствовал маркер Ph3-412, а для маркера NC-LB-9-6678 был отмечен только фрагмент 900 п.н., в то время как связанный с устойчивостью фрагмент 600 п.н. отсутствовал (табл.). Из девяти средневосприимчивых образов маркер Ph3-412 был обнаружен у семи, маркер устойчивости 600 п.н. NC-LB-9-6678 — у двух. В группе восприимчивых образцов маркер Ph3-412 выявили у 13, а известный маркер устойчивости — у одного образца. Следовательно, ни один из маркеров не показал однозначной связи с полевой устойчивостью.
Чтобы выяснить, с чем могут быть связаны такие неоднозначные результаты, мы выполнили клонирование и секвенирование ПЦР-продуктов размером 412 п.н., полученных с праймерами Ph3-412, и ПЦР продуктов размером 600 п.н. и 900 п.н., полученных с праймерами NC-LB-9-6678. Полученные нуклеотидные последовательности сравнили с последовательностью гена-прототипа Ph-3 и его структурных гомологов SlRGA1, SlRGA2, SlRGA3 и SlRGA4. Результаты сравнения представлены в виде дендрограмм (рис. 2).
Фрагмент, амплифицированный с праймерами Ph3-412, кластеризовался вместе с геном-прототипом Ph-3, и его нуклеотидная последовательность была на 99,3 % гомологична последовательности Ph-3 (см. рис. 2, А). То есть можно с уверенностью утверждать, что созданные нами праймеры специфично амплифицируют именного ген Ph-3, а не его гомологи. Точный размер фрагментов, амплифицированных с праймерами NC-LB-9-6678, составил 601 п.н. и 907 п.н. При этом оказалось, что фрагмент размером 601 п.н. принадлежит гомологу SlRGA4:на дендрограмме он кластеризовался вместе с SlRGA4 и имел с ним 99,5 % гомологии по последовательности (см. рис. 2, Б).
Наиболее интересные результаты были получены при анализе нуклеотидной последовательности фрагмента размером 907 п.н. Оказалось, что различие в длине между фрагментами, полученными с праймерами NC-LB-9-6678, обусловлено наличием вставки фрагмента LTR ретротранспозона семейства Ty1-copia, размер которой составлял 306 п.н (рис. 3). При этом остальная часть последовательности фрагмента размером 907 п.н. была гомологична гену Ph-3 на 99,7 % и кластеризовалась с ним на дендрограмме (см. рис. 3, Б). То есть фрагмент размером 907 п.н. принадлежит генуPh-3, но со вставкой ретротранспозона, которая нарушает рамку считывания и трансляцию функционального белка. Таким образом, нами впервые было обнаружено, что в геноме томата ген Ph-3 может иметь вставку ретротранспозона, которая, по всей видимости, делает этот ген нефункциональным.
Совокупный анализ полученных результатов подтверждает высокую специфичность созданного нами маркера гена Ph-3 Ph3-412. Большинство проанализированных образцов томата (21 из 24) имели ген Ph-3, на что указывало наличие у них маркера Ph3-412 и фрагмента размером 907 п.н., получаемого с праймерами NC-LB-9-6678. Эти образцы, скорее всего, не имели гомологов гена Ph-3, поскольку у них отсутствовал фрагмент размером 601 п.н., который, по нашим данным, принадлежит гомологу SlRGA4, и ни один из образцов не имел одновременно маркеры 412 п.н./907 п.н. и 601 п.н. По-видимому, при интрогрессии генетического материала S. pim-pinellifolium в геном томата локус, содержащий Ph-3, в процессе гомологичной рекомбинации заменял собой локус, содержащий все гомологи этого гена в геноме S. lycopersicum.
Однако образцы, имевшие маркер Ph3-412, по-видимому, содержали неактивный ген Ph-3 со вставкой ретротранспозона семейства Ty1-copia. Ретротранспозоны этого типа очень распространены в геноме томата (23). Наличием вставки ретротранспозона можно объяснить тот факт, что созданный нами маркер Ph3-412 не показал четкой связи с полевой устойчивостью проанализированных образцов к фитофторозу: все они содержали инактивированную форму гена Ph-3.
Устойчивость образцов к фитофторозу была обусловлена другими генетическими факторами. Например, устойчивый сорт Оттава 30, имеющий, по нашим данным, ген Ph-3 внеактивной форме, содержит гены устойчивости Ph-1 и Ph-2 (24, 25). Поскольку источником этих генов также служил S. pimpinellifolium, можно предположить, что у донорных форм, включенных в селекционный процесс, ген Ph-3 исходно имел вставку ретротранспозона. Впоследствии генетический материал именно этих доноров, а не образца L3708, широко распространился среди сортов томата отечественной селекции.
Согласно нашим результатам, по той же причине маркер NC-LB-9-6678 не показал связи с полевой устойчивостью к фитофторозу, поскольку его верификацию авторы проводили в расщепляющихся популяциях растений томата, полученных от скрещивания с первоначальным донором гена Ph-3 — образцом L3708 S. pimpinellifolium (20). В результате, несмотря на свою неспецифичность, маркер NC-LB-9-6678 позволил авторам отличить неактивную форму (фрагмент 907 п.н.), содержащую вставку у восприимчивых генотипов, от функциональной формы Ph-3 устойчивых образцов, которая при ПЦР амплификации дает фрагмент размером 601 п.н., как и другие гомологи этого гена. Поскольку в работе D.R. Panthee с соавт. (20) в качестве донора гена Ph-3 выступал не содержащий других гомологов образец L3708, в полученных устойчивых формах такие гомологи также отсутствовали и фрагмент размером 601 п.н. соответствовал функциональному гену Ph-3.
Интересно, что D.R. Panthee с соавт. (20) также отмечали наличие устойчивых форм, которые по результатам анализа имели оба фрагмента — 907 п.н. и 601 п.н. и, наряду с геном Ph-3, содержали ген Ph-2. То есть неактивная форма гена Ph-3 может попадать в геном томатов при введении в него других активных генов устойчивости S. pimpinellifolium, так как эти гены находятся на разных хромосомах, наследуются не сцеплено и отбор по ним идет независимо. Можно предположить, что в качестве доноров устойчивости к фитофторозу отечественные селекционеры могли использовать формы S. pimpinellifolium, содержащие ген Ph-3 со вставкой, поэтому в наших экспериментах маркер NC-LB-9-6678 не сработал как маркер устойчивости к фитофторозу.
Таким образом, нами создан высокоспецифичный маркер Ph3-412 гена устойчивости томата к фитофторозу Ph-3. Также мы показали, что в сортах томата отечественной селекции при наличии гена Ph-3 отсутствуют другие гомологи этого гена. У проанализированных нами образцов, в которых был обнаружен ген Ph-3,в его последовательности присутствовала вставка ретротранспозона. Наличие такой вставки может приводить к потере функциональной активности, что необходимо учитывать при маркировании гена Ph-3 при проведении маркер-опосредованной селекции на фитофтороустойчивость. В качестве доноров устойчивости к фитофторозу следует привлекать формы томата, у которых ген Ph-3 не имеет вставки ретро-транспозона. Созданный нами маркер Ph3-412 позволяет выявлять таких доноров при совместном применении с маркером NC-LB-9-6678 и обеспечивает возможность их использования в селекции.
Сравнение результатов молекулярного анализа с использованием ДНК-маркеров гена устойчивости к фитофторозу Ph-3 |
||||
Сорт |
Группа устойчивости по фенотипу |
Маркер |
||
Ph3-412 |
NC-LB-9-6678 |
NC-LB-9-6678 |
||
Оттава 30 (стандарт устойчивости) |
ОУ |
+ |
- |
+ |
Фитилек |
СВ |
+ |
- |
+ |
Лотос |
СВ |
+ |
- |
+ |
Патрокл |
СВ |
+ |
- |
+ |
Приморец |
СВ |
+ |
- |
+ |
Благодатный |
СВ |
+ |
- |
+ |
Талисман |
СВ |
+ |
- |
+ |
Монах |
СВ |
+ |
- |
+ |
л-ДВot30-2/19 |
СВ |
+ |
- |
+ |
Реванш |
СВ |
- |
+ |
- |
Викинг |
СВ |
- |
+ |
- |
л-Ft5/20 |
В |
- |
+ |
- |
Талалихин (стандарт восприимчивости) |
В |
+ |
- |
+ |
Росинка |
В |
+ |
- |
+ |
Чаровница |
В |
+ |
- |
+ |
Топтыжка |
В |
+ |
- |
+ |
Дубок |
В |
+ |
- |
+ |
Перст |
В |
+ |
- |
+ |
Камея |
В |
+ |
- |
+ |
Восход ВНИИССОК |
В |
+ |
- |
+ |
Посьет |
ВВ |
+ |
- |
+ |
Грот |
ВВ |
+ |
- |
+ |
Одиссей |
ВВ |
+ |
- |
+ |
Северянка |
ВВ |
+ |
- |
+ |
Примечание. ОУ — относительно устойчив, СВ — средневосприимчив, В — восприимчив, ВВ — высоковосприимчив; |
||||
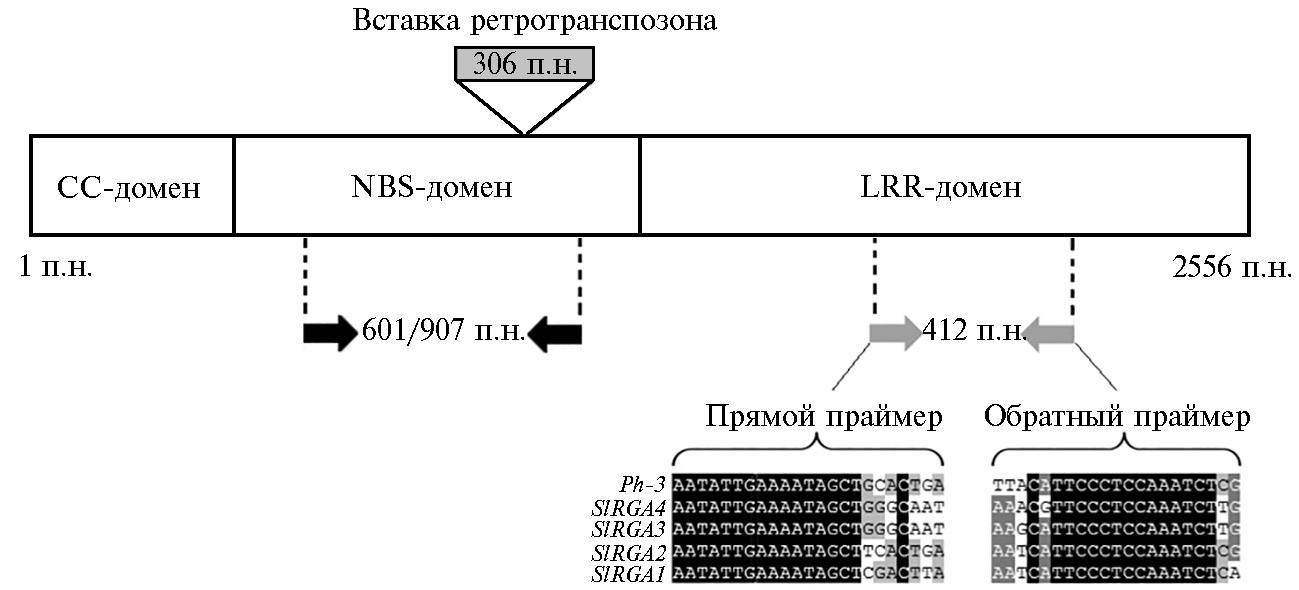
Рис. 1. Электрофореграмма продуктов ПЦР-амплификации тотальной ДНК образцов томата (Solanum lycopersicum L.) с праймерами маркеров Ph3-412 (А) и NC-LB-9-6678 (Б), специфичными к гену устойчивости к фитофторозу Ph-3: М – маркер молекулярной массы, 1 — Оттава 30, 2 — л-ДВot30-2/19, 3 — Фитилек, 4 — л-Ft5/20, 5 — Приморец, 6 — Грот, 7 — Чаровница, 8 — Лотос, 9 — Талалихин, 10 — Росинка, 11 — Посьет, 12 — Топтыжка, 13 — Одиссей, 14 — Патрокл, 15 — Благодатный, 16 — Викинг, 17 — Дубок, 18 — Реванш, 19 — Талисман, 20 — Монах, 21 — Перст, 22 — Камея, 23 — Северянка, 24 — Восход ВНИИССОКа.
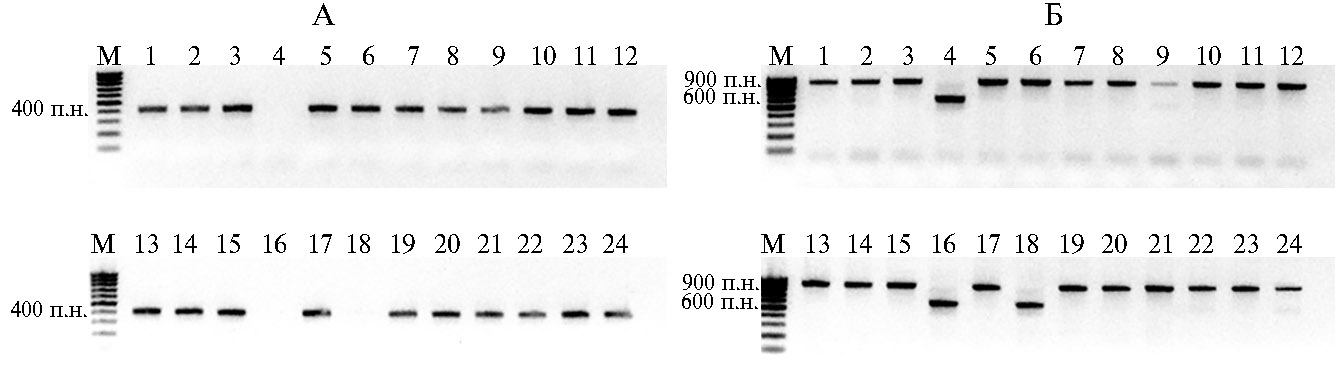
Рис. 2. Дендрограммы, построенные по результатам сравнения нуклеотидных последовательностей гена устойчивости томата к фитофторозу Ph-3 и его гомологов SlRGA1, SlRGA2, SlRGA3, SlRGA4 с нуклеотидной последовательностью ПЦР-продукта размером 412 п.н., полученного с праймерами маркера Ph3-412 (А), и нуклеотидными последовательностями ПЦР-продуктов размером 601 п.н. и 907 п.н., полученных с праймерами маркера NC-LB-9-6678 (Б).
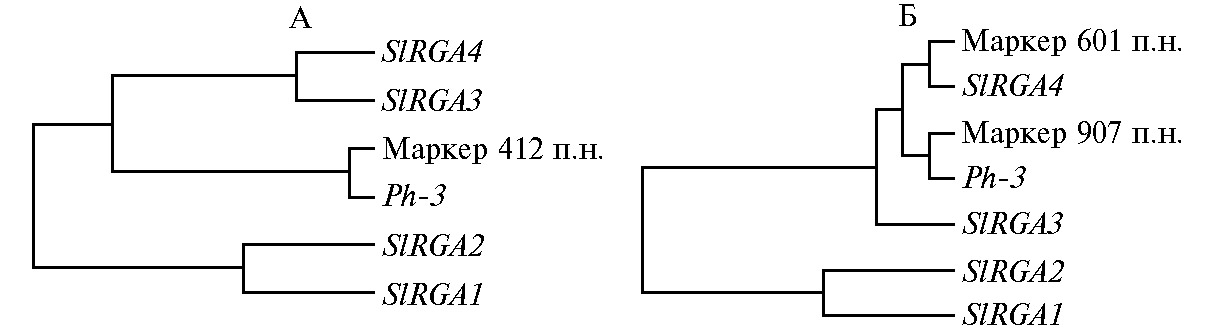
Рис. 3. Схематическое изображение гена устойчивости томата к фитофторозу Ph-3, расположение праймеров и вставка ретротранспозона. CC-домен, NBS-домен и LRR-домен обозначают участки гена, кодирующие соответствующие домены белка Ph-3; серыми стрелками обозначено расположение праймеров маркера Ph3-412, их нуклеотидные последовательности приведены в сравнении с нуклеотидными последовательностями соответствующих участков гомологов гена, черными стрелками обозначено расположение праймеров маркера NC-LB-9-6678; числа 1 и 2556 обозначают размер гена Ph-3 в п.н.
ЛИТЕРАТУРА
- Judelson H.S., Blanco F.A. The spores of Phytophthora: weapons of the plant destroyer. Nature Reviews Microbiology, 2005, 3(1): 47-58 (doi: 10.1038/nrmicro1064).
- Deahl K.L., Cooke L.R., Black L.L., Wang T.C., Perez F. M., Moravec B.C., Quinn M., Jones R.W. Population changes in Phytophthora infestans in Taiwan associated with the appearance of resistance to metalaxyl. Pest Management Science, 2002, 58(9): 951-958 (doi: 10.1002/ps.559).
- Fry W.E. Phytophthora infestans: the plant (and R gene) destroyer. Molecular Plant Pathology, 2008, 9(3): 385-402 (doi: 10.1111/j.1364-3703.2007.00465.x).
- Randall E., Young V., Sierotzki H., Scalliet G., Birch P.R.J., Cooke D.E.L., Csukai M., Whisson S.C. Sequence diversity in the large subunit of RNA polymerase I contributes to mefenoxam insensitivity in Phytophthora infestans. Molecular Plant Pathology, 2014, 15(7): 664-676 (doi: 10.1111/mpp.12124).
- Saville A., Graham K., Grünwald N.J., Myers K., Fry W.E., Ristaino J.B. Fungicide sensitivity of US genotypes of Phytophthora infestans to six oomycete-targeted compounds. Plant Disease, 2015, 99(5): 659-666 (doi: 10.1094/PDIS-05-14-0452-RE).
- Montes M.S., Nielsen B.J., Schmidt S.G., Bødker L., Kjøller R., Rosendahl S. Population genetics of Phytophthora infestans in Denmark reveals dominantly clonal populations and specific alleles linked to metalaxyl-M resistance. Plant Pathology Journal, 2016, 65(5): 744-753 (doi: 10.1111/ppa.12462).
- Brouwer D.J., Jones E.S., St Clair D.A. QTL analysis of quantitative resistance to Phytophthora infestans (late blight) in tomato and comparisons with potato. Genome, 2004, 47(3): 475-492 (doi: 10.1139/g04-001).
- Foolad M.R., Merk H.L., Ashrafi H. Genetics, genomics and breeding of late blight and early blight resistance in tomato. Critical Reviews in Plant Sciences, 2008, 27(2): 75-107 (doi: 10.1080/07352680802147353).
- Li J., Liu L., Bai Y., Finkers R., Wang F., Du Y., Yang Y., Xie B., Visser R.G.F., van Heusden A.W. Identification and mapping of quantitative resistance to late blight (Phytophthora infestans) in Solanum habrochaites LA1777. Euphytica, 2011, 179(3): 427-438 (doi: 10.1007/s10681-010-0340-7).
- Nowicki M., Foolad M.R., Nowakowska M., Kozik E.U. Potato and tomato late blight caused by Phytophthora infestans: an overview of pathology and resistance breeding. Plant Disease, 2012, 96(1): 4-17 (doi: 10.1094/PDIS-05-11-0458).
- Merk H.L., Ashrafi H., Foolad M.R. Selective genotyping to identify late blight resistance genes in an accession of the tomato wild species Solanum pimpinellifolium. Euphytica, 2012, 187(1): 63-75 (doi: 10.1007/s10681-012-0729-6).
- Peirce L.C. Linkage tests with Ph conditioning resistance to race 0 Phytophthora infestans. Report of the Tomato Genetics Cooperative, 1971, 21: 30.
- Moreau P., Thoquet P., Olivier J., Laterrot H., Grimsley N. Genetic mapping of Ph-2, a single locus controlling partial resistance to Phytophthora infestans in tomato. Molecular Plant-Microbe Interactions, 1998, 11(4): 259-269 (doi: 10.1094/MPMI.1998.11.4.259).
- Chunwongse J., Chunwongse C., Black L., Hanson P. Molecular mapping of the Ph-3 gene for late blight resistance in tomato. The Journal of Horticultural Science and Biotechnology, 2002, 77(3): 281-286 (doi: 10.1080/14620316.2002.11511493).
- Chen C.H., Sheu Z.M., Wang T.C. Host specificity and tomato-related race composition of Phytophthora infestans isolates in Taiwan during 2004 and 2005. Plant Disease, 2008, 92(5): 751-755 (doi: 10.1094/PDIS-92-5-0751).
- Kim M.J., Mutschler M.A. Transfer to processing tomato and characterization of late blight resistance derived from Solanum pimpinellifoium L. L3708. Journal of the American Society for Horticultural Science, 2005, 130(6): 877-884 (doi: 10.21273/JASHS.130.6.877).
- Zhang C., Liu L., Wang X., Vossen J., Li G., Li T., Zheng Z., Gao J., Guo Y., Visser R.G.F., Li J., Bai Y., Du Y. The Ph-3 gene from Solanum pimpinellifolium encodes CC-NBS-LRR protein conferring resistance to Phytophthora infestans. Theoretical and Applied Genetics, 2014, 127(6): 1353-1364 (doi: 10.1007/s00122-014-2303-1).
- Park Y., Hwang J., Kim K., Kang J., Kim B., Xu S., Ahn Y. Development of the gene-based SCARs for the Ph-3 locus, which confers late blight resistance in tomato. Scientia Horticulturae, 2013, 164: 9-16 (doi: 10.1016/j.scienta.2013.08.013).
- Truong H.T.H., Tran H.N., Choi H.S., Park P.H., Lee H.E. Development of a co-dominant SCAR marker linked to the Ph-3 gene for Phytophthora infestans resistance in tomato (Solanum lycopersicum). European Journal of Plant Pathology, 2013, 136(2): 237-245 (doi: 10.1007/s10658-012-0157-4).
- Panthee D.R., Gardner R.G., Ibrahem R., Anderson C. Molecular markers associated with Ph-3 gene conferring late blight resistance in tomato. American Journal of Plant Sciences, 2015, 6(13): 2144-2150 (doi: 10.4236/ajps.2015.613216).
- Wang Y.Y., Chen C.H., Hoffmann A., Hsu Y.C., Lu S.F., Wang J.F., Hanson P. Evaluation of the Ph-3 gene-specific marker developed for marker-assisted selection of late blight-resistant tomato. Plant Breeding, 2016, 135(5): 636-642 (doi: 10.1111/pbr.12395).
- Van de Peer Y., de Wachter R. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the microsoft windows environment. Computer Applications in the Biosciences, 1994, 10(5): 569-570 (doi: 10.1093/bioinformatics/10.5.569).
- Cheng X., Zhang D., Cheng Z., Keller B., Ling H.Q. A new family of Ty1-copia-like retrotransposons originated in the tomato genome by a recent horizontal transfer event. Genetics, 2009, 181(4): 1183-1193 (doi: 10.1534/genetics.108.099150).
- Michalska A.M., Pazio M. A new method for evaluating tomato leaf resistance to Phytophthora infestans using a seedling test. Plant Breeding and Seed Science, 2002, 46(1): 3-21.
- Michalska A.M., Sobkowiak S., Flis B., Zimnoch-Guzowska E. Virulence and aggressiveness of Phytophthora infestans isolates collected in Poland from potato and tomato plants identified no strong specificity. European Journal of Plant Pathology, 2016, 144(2): 325-336 (doi: 10.1007/s10658-015-0769-6).