doi: 10.15389/agrobiology.2021.5.885rus
УДК 635.64:581.192:575:577.2
СТРУКТУРНО-ФУНКЦИОНАЛЬНЫЙ АНАЛИЗ ГЕНОВ-ГОМОЛОГОВ GME1 И НАКОПЛЕНИЕ АСКОРБАТА В ПЛОДАХ У КУЛЬТУРНЫХ И ДИКОРАСТУЩИХ ВИДОВ ТОМАТА
Д.Ю. ТЯПКИНА1, 2 ✉, М.А. СЛУГИНА2, Е.З. КОЧИЕВА2
Аскорбиновая кислота (аскорбат, витамин С) играет важную роль в различных метаболических процессах и у растений, и у человека, поэтому увеличение содержания аскорбата в клетках растений при помощи селекционных подходов важно для повышения как пищевой ценности плодов, так и устойчивости растений к стрессу. Известно, что томат обладает высоким потенциалом как источник аскорбата в рационе человека. К сожалению, у современных сортов и гибридов томата овощного (Solanum lycopersicum L.) содержание аскорбата в спелых плодах мало в сравнении с его дикорастущими родственными видами. Исследования показали, что селекционные программы с привлечением дикорастущих видов томата могут значительно увеличить содержание аскорбата в спелых плодах. Однако для эффективной селекции по этому признаку необходимо более детальное изучение генетических детерминант, ответственных за накопление аскорбата в спелых плодах. В представляемой работе нами впервые амплифицированы, клонированы и секвенированы гомологи гена GME1, играющего ключевую роль в биосинтезе аскорбата, у томата овощного и 11 дикорастущих видов томата. Структурный анализ показал низкую степень вариабельности гена GME1 у изученных образцов. В кодирующих последовательностях GME1 выявлено 28 однонуклеотидных замен (single nucleotide polymorphisms, SNPs), из которых только две (G2E и E281D) приводили к несинонимичным заменам у видов S. neorickii и S. peruvianum var. dentatum. Анализ мотивов и доменов белка GME1 томатов не выявил каких-либо специфических мотивов ни на межвидовом уровне, ни на более отдаленных таксономических уровнях. Наблюдаемый высокий консерватизм GME1 у достаточно эволюционно отдаленных видов томата, по всей видимости, служит свидетельством функциональной значимости этого фермента для синтеза аскорбата и, опосредованно, для защиты от стрессовых факторов, прежде всего фотостресса. Корреляцию между аминокислотными (нуклеотидными) заменами и содержанием аскорбата в плодах мы не обнаружили. При сравнительном межвидовом органоспецифическом анализе и анализе зависимости количества аскорбата в зрелых плодах у сортов и дикорастущих образцов от уровня экспрессии GME1 мы также не выявили связи между транскрипционной активностью GME1 и концентрацией аскорбата. Можно также предположить, что определяющее влияние на конечное содержание аскорбата в созревшем плоде томата, вероятно, оказывает не интенсивность экспрессии GME1 на завершающей стадии созревания плода, а то, насколько ген GME1 был активен на более ранних стадиях созревания.
Ключевые слова: GME1, уровень экспрессии, однонуклеотидные замены, SNP, Solanum lycopersicum L., томат, дикие виды, культивируемые виды, зрелые плоды, содержание аскорбата.
Аскорбиновая кислота (аскорбат, витамин С) играет важную роль во многих метаболических процессах у растений — в фотосинтезе, фотозащите, устойчивости к стрессу, контроле роста клеток, биосинтезе гормонов и компонентов клеточной стенки (1-5). В растительной клетке аскорбиновая кислота выполняет функцию основного антиоксиданта благодаря способности снижать содержание активных форм кислорода, которые образуются при фотосинтезе и абиотических стрессах (например, при высокой интенсивность света, высоких температурах и сильном ультрафиолетовом излучении) (6, 7). Человек и некоторые приматы не способны синтезировать аскорбат, поэтому растительная пища, богатая витамином С, необходима для поддержания нормальной жизнедеятельности (8), и поиск новых источников аскорбата имеет важнейшее значение (9).
Томат овощной (Solanum lycopersicum L.) — одна из наиболее часто употребляемых в пищу овощных культур с широким ареалом возделывания (10, 11). Сорта томата могут служить дополнительным источником витамина С при круглогодичном потреблении (12). Однако у современных сортов и гибридов культурного томата содержание аскорбата в спелых плодах невелико в сравнении с таковым у родственных дикорастущих видов (13). Исследования показали, что селекционные программы с привлечением дикорастущих видов томата, в частности S. pennellii, могут значительно увеличить содержание аскорбата в спелых плодах (14-18). Однако для эффективной селекции по этому признаку необходимо более детальное изучение генетических детерминант, ответственных за накопление аскорбата в спелых плодах.
Одной из важнейших реакций биосинтеза аскорбата в клетках высших растений считается превращение GDP-D-маннозы в GDP-1-галактозу, которое катализирует фермент GDP-манноза-3´,5´-эпимераза (GME; EC 5.1.3.18). В отличие от большинства растений, которые имеют лишь одну копию гена GME, в геноме томата идентифицированы две копии — GME1 и GME2 на 1-й и 9-й хромосомах (17, 19). Анализ колокализации QTL (quantitative trait loci) указывает на то, что GME может служить генетической детерминантой повышенного уровня аскорбата в плодах томата (17, 19). Это предположение подтверждает работа L. Gilbert с соавт. (20), где для подавления экспрессии обоих генов — SlGME1 и SlGME2 использовался метод РНК сайленсинга. Позднее C. Zhang с соавт. (21) продемонстрировали, что сверхэкспрессия обоих генов значительно увеличила содержание аскорбата в листьях и спелых плодах томата. Это напрямую влияло на стрессоустойчивость модифицированных растений. Положительную корреляцию между экспрессией GME и содержанием аскорбата показали также для яблони и черники (22, 23). Анализ эволюционных паттернов гена GME на основе сравнения 59 геномов разных видов высших растений и зеленых водорослей выявил высокую степень консервативности последовательности и структурной организации GME у высших растений (24). Однако структуру генов-гомологов GME у дикорастущих видов томата до настоящего времени не исследовали.
В представляемой работе мы впервые выявили и детально охарактеризовали гены-гомологи GME1 у сортов культивируемого томата S. lycopersicumи образцов дикорастущих видов, характеризующихся высоким содержанием аскорбата в плодах. Определена вариабельность нуклеотидных и аминокислотных последовательностей, состав функциональных мотивов у генов-гомологов GME1. Анализ экспрессии таких генов-гомологов у образцов томата с разным содержанием аскорбата в товарной продукции показал отсутствие связи между этим признаком и транскрипционной активностью GME1 в зрелых плодах.
Целью исследования стала идентификация новых генов-гомологов GME1 и сравнительный анализ их структуры и транскрипционной активности у разных видов томата для выявления возможных корреляций между различиями этих генов и накоплением аскорбата в плодах.
Методика. Растительный материал включал 15 образцов томата (секция Lycopersicon, род Solanum) — 11 дикорастущих видов S. chmielewskii (VIR13725), S. neorickii (VIR5033), S. chilense (VIR4300), S. corneliomulleri (VIR4367), S. peruvianum (VIR4361), S. peruvianum var. dentatum (VIR3966), S. arcanum (VIR13958), S. habrochaites (VIR13964), S. cheesmaniae (VIR3969), S. galapagense (VIR3970), S. pimpinellifolium var. racemigerum (VIR1018), дикорастущий образец томата овощного S. lycopersicum var. humboldtii (VIR2912), сорта томата овощного S. lycopersicum Silvestre recordo (VIR1580), Бычье сердце и Желтый белорус (предоставлены ФГБНУ ФИЦ Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Пе-тербург, ФНЦО ФНЦ овощеводства и ООО «Гавриш», Московская обл.). Растения выращивали в теплице (23 °С, длина светового дня 16 ч, освещенность 5 тыс. лк/м2).
При определении содержания аскорбата в плодахдля его экстракции использовали метод G. Giovanelli с соавт. (25) с модификациями. К свежим томатам добавляли водный раствор метафосфорной кислоты (1 г/100 мл) в соотношении 1:10 (масса/объем) и гомогенизировали в блендере Waring 8011 («Waring», США). Полученный гомогенат быстро переносили в полипропиленовые пробирки и центрифугировали при 11000 g в течение 20 мин при 4 °С. Анализ выполняли методом высокоэффективной жидкостной хроматографии (ВЭЖХ, жидкостный хроматограф с масс-детектором LC-QQQ Agilent 6460, «Agilent Technologies», США; колонка ACE® 5 C18, «Advanced Chromatography Technologies, Ltd.», Великобритания) (ЦКП «Промышленные биотехнологии ФИЦ Биотехнологии РАН», г. Москва). Подвижная фаза — сверхчистая вода (рH 3), изократическое элюирование (скорость потока 0,5 мл/мин, температура 35 °С).Супернатанты предварительно фильтровали через мембранные фильтры с размером пор 0,45 мкм («Amersham», США). Объем анализируемых проб 20 мл. Концентрацию аскорбиновой кислоты выражали в расчете на 100 г гомогенизированной мякоти плода. Содержание аскорбата определяли в двух биологических повторностях.
Геномную ДНК выделяли из свежесобранных листьев томата по методу K. Edwards с соавт. (26). Оценивали чистоту и определяли концентрацию полученных препаратов ДНК (спектрофотометр DU 530, «Beckman», США).
Для выделения РНК с последующим синтезом кДНК использовали листья, корни, цветки и плоды (на стадии полной спелости) дикорастущих образцов видов томата S. lycopersicum var. humboldtii, S. peruvianum, S. habrochaites итрех сортов томата овощного S. lycopersicum Silvestre recordo, Бычье сердце и Желтый белорус. РНК выделяли с помощью набора реактивов RNeasy Plant Mini Kit («Qiagen», Германия) в соответствии с рекомендациями производителя. Полученные препараты РНК обрабатывали ДНКазой I (10 ед.) («Invitrogen», США) в соответствии с прилагаемым описанием. Препараты кДНК получали с использованием набора GoScript™ Reverse Trans-cription System («Promega», США) согласно методике производителя. Концентрацию РНК и кДНК определяли на флуориметре Qubit 4 («Thermo Fisher Scientific», США) с помощью соответствующих реактивов («Invitrogen», США). Дополнительно качество РНК проверяли методом электрофореза в 1,5 % агарозном геле.
ПЦР-амплификацию проводили на приборе Veriti™ 96-well Thermal Cycler («Applied Biosystems», США) в реакционной смеси объемом 15 мкл, содержащей 1,5 мкл Dream™ Taq буфера 10×, 20 мМ dNTP, 10 мкМ специально разработанных нами праймеров GME F (5´-CACTGTATTAGTGCC-TCATC-3´) и GME R (5´-CAATTACCAGAATCTAACACATC-3´), 0,25 мкл Dream™ Taq DNA Polymerase (5 U/мкл) («Fermentas», Литва) и ~ 100 нг геномной ДНК. Условия амплификации: 5 мин при 95 °С; 15 с при 95 °С, 10 с при 58 °С, 1 мин при 72 °С (40 циклов); конечная элонгация — 4 мин при 72 °С.
Полученные продукты ПЦР анализировали методом электрофореза в 1,5 % агарозном геле в 1× ТВЕ-буфере, окрашенном бромистым этидием. Результаты документировали в системе Gel Doc™ XR+ Imaging System («Bio-Rad», США). Использовали коммерческие стандарты для определения размеров амплифицированных фрагментов ДНК 1 Kb DNA Ladder и 100 bp DNA Ladder («Fermentas», Литва).
ПЦР-продукты клонировали с помощью системы плазмидного вектора pGEM→-T Easy Vector («Promega», США) согласно методике, предложенной производителем, с дополнительной депротеинизацией смесью фенол:хлороформ (1:1).
Анализ экспрессии генов-гомологов GME1 проводили в четырех органах томата — корень, лист, цветок, плод в биологической спелости методом количественной ПЦР в реальном времени (РВ-ПЦР, RT-qPCR) с использованием набора «Реакционная смесь для проведения РВ-ПЦР в присутствии SYBR GreenI и ROX» (ООО «Синтол», Россия) на амплификаторе CFX96 Real-Time PCR Detection System («Bio-Rad Laboratories», США). Реакции проводили в двух биологических и трех аналитических повторах. Для анализа экспрессии использовался следующий протокол: 5 мин при 95 °С; 15 с при 95 °С, 30 с при 60 °С (40 циклов), чтение результатов. Для получения кривых плавления продуктов амплификации увеличивали температуру от 55 °С до 95 °С (с шагом 0,5 °С каждые 5 с) с последующим чтением результатов. Для анализа экспрессии генов-гомологов GME1 была разработана пара праймеров GMErnaF (5´-AGAATGGGAAGCTCTGGTGG-3´) и GMErnaR (5´-GGCTTCCAATTGAAATGATGACAG-3´), позволяющая амплифицировать фрагмент длиной 185 п.н. При нормализации использовали величину экспрессии референсных генов Expressed и Actin 2/7 согласно описанию (27, 28).
Биоинформатический анализ нуклеотидных последовательностей проводили с помощью программы MEGA 7.0 (29) (https://www.kent.ac.uk/soft-ware/mega-7). Анализ последовательностей аминокислот и предсказание наличия сайтов замен, оказывающих значительное влияние на функциональность фермента, выполняли в программе PROVEAN (30) (http://pro-vean.jcvi.org/index.php). Поиск общих скрытых мотивов проводили в программе MEME 5.3.2 (31) (https://meme-suite.org/meme/tools/meme). Для дополнительного анализа аминокислотных последовательностей использовали ресурс NCBI-CDD (https://www.ncbi.nlm.nih.gov/cdd/) и базу данных UniProt (https://www.uniprot.org). Для сравнительного анализа нуклеотидных и аминокислотных последовательностей также были использованы последовательности генов-гомологов GME из базы данных NCBI — S. lycopersicum сорт Heinz 1706; S. pimpinellifolium (LA0480), S. arcanum (LA2157); Oryza sativa и Arabidjpsis thaliana.
При определении содержания аскорбата рассчитывали среднеарифметические значения.
Результаты. Н а к о п л е н и е а с к о р б а т а в з р е л ы х п л о д а х. Распределение изученных образцов по содержанию витамина С в плодах иллюстрирует таблица 1. Биохимический анализ показал, что у эволюционно более молодых красноплодных видов томата S. lycopersicum и S. pimpinellifolium накопление аскорбата происходит равномерно по мере созревания плода с максимум в плоде биологической спелости. У большинства зеленоплодных перекрестноопыляемых томатов наблюдается обратная динамика с максимальным содержанием аскорбата на ранних стадиях развития плода. Были выявлены образцы с наименьшим и наибольшим содержанием аскорбата в плоде на стадии биологической спелости. Среди представителей S. lycopersicum наименьшее количество аскорбата детектировали в образцах Желтый белорус (28,1 мг/100 г гомогенизированной мякоти плода) и Бычье сердце (42,8 мг/100 г), наибольшее — у S. lycopersicum var. humboldtii (120,1 мг/100 г). Полученные значения несколько превышали данные, полученные ранее для сортов и образцов дикорастущих видов S. pennellii и S. pimpinellifolium (32), что может быть результатом специфичности проявления этого признака у изученных сортов и образцов, а также условий выращивания растений.
И д е н т и ф и к а ц и я и х а р а к т е р и с т и к а в а р и а б е л ь н о с т и г е н о в - г о м о л о г о в GME1. При дизайне праймеров для амплификации ДНК генов GME — фермента, вовлеченного в синтез аскорбата, использовали представленные в базе данных NCBI геномы томата овощного S. lycopersicum сорта Heinz 1706 (NM_001247914.2), дикорастущего вида томата S. pennellii (XM_015214839.2) и картофеля S. tuberosum (GCA_000226075.1) и последовательности мРНК. В результате для амплификации гена GME1 была разработана пара фланкирующих праймеров GME F–GME R (табл. 2). Кроме того, для секвенирования были разработаны внутренние праймеры с локализацией в последовательностях экзонов. В итоге для амплификации и секвенирования генов GME1 получили семь внутренних праймеров (см. табл. 2, рис. 1).
С помощью разработанных праймеров GME F—GME R мы амплифицировали последовательности генов-гомологов GME1 у 11 дикорастущих видов томата и сорта томата овощного Silvestre recordo как представителя вида lycopersicum.Полученные фрагменты были клонированы и секвенированы с использованием внутренних праймеров.
Сравнение структуры генов-гомологов GME1 у изученных образцов томатапоказало, что у всех анализируемых видов этот ген состоит из шести экзонов (см. рис. 1), что совпало с данными по анализу экзон-интронной структуры GME1 гена S. lycopersicum, полученными ранее (21). Длина гена варьировала от 1962 п.н. (S. galapagense) до 1985 п.н. (S. peruvianum). Размеры гена не зависели от того, был ли вид эволюционно более древним (зеленоплодным) или более молодым (красноплодным). Сравнение с ранее полученными данными по гену GMEу культивируемого томата сорта Ailsa Craig также не выявило разницы в нуклеотидных последовательностях (21).
В полногеномной последовательности GME1 мы обнаружили 139 однонуклеотидных замен (single nucleotide polymorphisms, SNPs), что составило 6,9 %. Из них 134 SNPs были найдены у зеленоплодных видов томатов и всего лишь 5 — у красноплодных видов. Подавляющее большинство (108 SNPs) выявили в интронных последовательностях. Ранее при анализе генома томата было показано, что частота встречаемости нуклеотидных замен в интронных последовательностях составляет в среднем 8±2 % (33). Описанный нами в представляемой работе уровень полиморфизма интронов у генов-гомологов GME1 в целом соответствует приведенному значению. Также в результате проекта The 100 Tomato Genome Sequencing Consortium было показано, что вариабельность последовательностей генов у красноплодных томатов в 20 раз ниже, чем у зеленоплодных видов (33). В нашем исследовании это разница для GME1 оказалась 26,8-кратной, что также согласуется с ранее опубликованными данными (33).
В кодирующей области были обнаружены 28 однонуклеотидных замен — два SNPs у красноплодных видов и 26 SNPs у зеленоплодных (рис. 2, см. на сайте http://www.agrobiology.ru). Число синонимичных замен в области экзонов составляло от 7-8 SNPs у образцов S. peruvianum до 12 и 13 соответственно у S. arcanum и S. neorickii. Только две замены — G2E и E281D, выявленные у зеленоплодных видов S. neorickii и S. peruvianum var. dentatum, оказались несинонимичными.
У томата, помимо нуклеотидных замен в анализируемых последовательностях генов-гомологов GME1, мы обнаружили 22 индела размером от 1 до 10 п.н. Все инделы, как и ожидалось, локализовались в интронах, максимальное число инделов (семь) находилось в V интроне. Интересно, что в IV интроне (в положении 1237 от стартового кодона) был выявлен 9-нуклеотидный индел (CCCTTGTA), присутствующий только у самоопыляемых видов, включая красноплодные виды S. lycopersicum (сорта Heinz 1706 и Silvestre recordo),S. pimpinellifolium (LA0480), S. pimpinellifolium var. racemigerum, S. cheesmaniae, S. galapagenseи зеленоплодный вид S. chmielewskii. Кроме инделей, в анализируемых последовательностях GME1 мы обнаружили пять SNPs, также характерных для самоопыляемых видов томата (рис. 3).
Кодирующие последовательности генов-гомологов GME1 были транслированы. Полученные белковые последовательности имели одинаковую длину — 376 а.о., что соответствовало молекулярной массе 42 кДа (табл. 3). В целом аминокислотные последовательности белков оказались сходными. В белках GME1 у зеленоплодных видов S. neorickii и S. peruvianum var. dentatum присутствовали две аминокислотные замены — G2E и E281D. Проведенный анализ этих замен с использованием программы PROVEAN показал, что они не являются радикальными и, предположительно, не могут влиять на функциональность GME1. Полученные результаты подтверждают данные о том, что GME относится к числу наиболее консервативных белков, вовлеченных в биосинтез аскорбата (24, 34).
Следует отметить, что оба образца,у которых быливыявлены несинонимичные замены, — S. neorickii и S. peruvianum var. dentatum существенно не отличались от других образцов по содержанию аскорбиновой кислоты (см. табл. 1) и динамике ее накопления.
У всех изученных видов томата анализ аминокислотных последовательностей GME1 с использованием базы данных UniProt (35) выявил эпимеразный домен (позиции 30-269 а.о.). Внутри домена у всех анализируемых белков были идентифицированы пять сайтов связывания с субстратом GME — GDP-маннозой (G105, N204, K226, R307 и S357), а также четыре сайта связывания с NAD+ — кофактором GME (D60, D80, Y175 и K179).
Ранее была определена структура и функции GMEу Arabidopsis thaliana (36). Проведенный нами сравнительный анализ с использованием пакета программ MEGA 7.0 показал высокую степень гомологии этого белка у арабидопсиса и томата, что позволяет предположить и функциональное сходство аминокислотной последовательности GME1 у томата и арабидопсиса. Так, на основе данных сайт-направленного мутагенеза гена GME у арабидопсиса было показано, что за активность окислительной и восстановительной эпимеризации отвечают именно аминокислоты С145 и К217 (36). Эти аминокислотные остатки совпадают с таковыми в белке GME1 томата и, следовательно, тоже могут быть определяющими для его функциональной активности. Аминокислоты С145 и К217 были инвариантны во всех проанализированных нами последовательностях GME1 у изученных образцов томата (рис. 4).
Анализ также показал, что GME1 является членом семейства удлиненной короткоцепочечной дегидратазы/редуктазы (short chain dehydratase/reductase, SDR), хотя и с модифицированным нуклеотид-связывающим мотивом, богатым глицином (37) (GAGGFIA вместо GXXGXXG) (36). У изученных нами видов томата в GME1 также был выявлен консервативный мотив GAGGFIA в положении 34-40 а.о. Столь высокая консервативность указанного мотива у белков GME, скорее всего, определяется его критической функцией, которая обусловлена участием в образовании водородных связей с NAD+.
Кроме того, мы проанализировали аминокислотные последовательности, полученные нами для GME1 у изученных видов томата и у гомологов других видов (O. sativa, A. thaliana) в программе MEME 5.3.2, что позволило выявить 10 консервативных мотивов. Все обнаруженные мотивы (см. дополнительные материалы на сайте http://www.agrobiology.ru) показывали высокую консервативность в сравнении с гомологами у других представителей рода Solanum и с гомологом GME арабидопсиса. Полученные нами результаты подтвердили данные о сходстве как гомологовGME1, так и GME2 у растений (24). Во втором мотиве (12-61 а.о.) выявлена последовательность GAGGFIA, нуклеотид-связывающие функции которой определены ранее для A. thaliana (36).
Также мы сравнили выявленные мотивы с использованием ресурса NCBI-CDD, что позволило отнести их к NAD-связывающим доменам — NAD(P)(+)-binding Rossmann-fold (NADB_Rossmann). Домен NADB характерен для многих дегидрогеназ метаболических путей и различных окислительно-восстановительных ферментов. Считается, что присутствием этого домена обусловлена эпимеразная активность GME1 (24).
Таким образом, проведенный анализ мотивов и доменов белка GME1 у изученных образцов томата и сравнение с гомологичными белками риса и арабидопсиса не выявил каких-либо специфических мотивов ни на межвидовом уровне, ни на более отдаленных таксономических уровнях. Наблюдаемый низкий уровень вариабельности белков GME1 у эволюционно достаточно отдаленных видов томата, например S. neorickii и S. lycopersicum (сорт Silvestre recordo), и наличие лишь двух радикальных замен подтверждает его консервативность, показанную ранее (34), и, по всей видимости, свидетельствует о функциональной значимости этого фермента (а также катализируемой им биохимической реакции) для синтеза аскорбата и, опосредованно, для защиты от стрессовых факторов, прежде всего от фотостресса.
А н а л и з э к с п р е с с и и г е н о в - г о м о л о г о в GME1. Ввиду того, что структурный анализ последовательностей генов-гомологов GME1 не выявил полиморфизмов, коррелировавших с накоплением аскорбата, мы предположили, что влияние фермента GME1 на биосинтез аскорбата может осуществляться на уровне транскрипции.
При изучении органоспецифичности транскрипционной активности генов-гомологов GME1 мы сравнили активность транскрипции этого гена в четырех органах (корень, лист, цветок и плод в стадии биологической спелости) у растений S. lycopersicum сорта Silvestre recordo и образца дикорастущего вида S. peruvianum, которые характеризовались контрастным содержанием аскорбата в плодах (см. табл. 1, рис. 5).
Проведенный анализ выявил наибольшую транскрипционную активность GME1 в листьях независимо от вида томата и накопления аскорбата в плодах. Подобный результат ожидаем, так как листья более всего подвержены фотоокислительному стрессу. При этом самый низкий уровень экспрессии гена GME1 отмечали в плодах (см. рис. 5).
Однако именно содержание аскорбата в плодах (и в частности, выяснение механизмов, обеспечивающих формирование этого признака) представляет наибольший практический интерес. Для более детального изучения возможной связи между накоплением аскорбата в товарной продукции и количеством транскриптов GME1 мы определили уровень экспрессии GME1 в плодах биологической спелости у пяти образцов — томата овощного S. lycopersicum сортов Silvestre recordo, Бычье сердце, Желтый белорус и образцов дикорастущих видов S. lycopersicum var. humboldtii и S. peruvianum (рис. 6, А). Отобранные образцы были контрастными по содержанию аскорбата в плодах и представляли как сорта и дикорастущий образец красноплодного культивируемого вида, так и образец дикорастущего зеленоплодного вида. У тех же пяти образцов мы проаннотировали экспрессию GME1 в зрелых плодах (см. рис. 6, Б).
Сопоставление результатов анализа экспрессии генов GME1 и биохимического анализа (см. рис. 6) не обнаруживает четкой корреляции между полученными показателями, допустимо лишь говорить о слабой обратной зависимости экспрессии гена в зрелых плодах и содержания в них аскорбата. Можно также предположить, что на конечное содержание аскорбата в созревшем плоде у анализируемых видов томата определяющее влияние может оказывать не интенсивность экспрессии GME1 на последней стадии созревания плода, а то, насколько этот ген был активен на более ранних стадиях созревания, что было показано ранее для S. lycopersicum сорта West Virginia 106(38). Для подтверждения или опровержения такого предположения необходимо изучить активность GME1 в процессе созревания плодов. Кроме того, следует рассматривать возможные альтернативные метаболические пути накопления аскорбата в спелых плодах томата, например L-галактозный путь, в котором в качестве субстрата используются полисахариды из клеточной стенки плода.
Проведенный биохимический анализ содержания аскорбата в зрелых плодах у изученных видов томата не показал корреляции с уровнями транскрипции GME1 или выявленными SNPs (см. рис. 6). Возможное объяснение в дополнение к сформулированному выше предположению, что транскрипционная активность гена GME1 может влиять на содержание аскорбата в плодах на стадиях, предшествующих полной зрелости, состоит в том, что GME1, помимо биосинтеза аскорбата, активно вовлечен в синтез клеточной стенки во время роста плода (38). Также следует учитывать, что помимо L-галактозного пути, для образования аскорбата в плодах томата могут быть использованы другие альтернативные пути биосинтеза, например мио-инозитольный (39).
Итак, в представленной работе нами впервые получены и проанализированы последовательности генов-гомологов GME1 в 11 видах культивируемого и дикорастущего томата. Проведенный структурно-функциональный анализ показал крайне высокую консервативность генов-гомологов GME1 и кодируемых ими белков как у эволюционно более древних зеленоплодных видов, так и у эволюционно более молодых красноплодных томатов. В генах-гомологах GME1 найдены 28 SNPs, характерные для самоопыляемых видов томата, что может быть использовано в селекционной работе. Не выявлена корреляция между содержанием аскорбата в зрелых плодах, с одной стороны, и обнаруженными SNPs или уровнями транскрипции GME1 — с другой. Результаты проведенного исследования показывают, что у изученных нами видов томата белок GME1 не имеет значимого влияния на накопление аскорбата в спелых плодах и косвенно подтверждают, что биосинтез аскорбата в плодах томата могут обеспечивать другие альтернативные пути, такие как мио-инозитольный или L-галактозный.
1. Скрининг изученных видов томата (Solanum L.) по содержанию аскорбата в плодах (М, мг/100 г гомогенизированной мякоти плода) в фазу полной спелости |
|||||
Генотип |
IG |
MG |
Br |
Спелый плод
|
|
S. lycopersicum Silvestre recordo (VIR1580) К/О |
0,2 |
11,9 |
23,2 |
68,5 |
|
S. pimpinellifolium var. racemigerum (VIR1018) К/Д |
36,5 |
77,4 |
99,3 |
150,3 |
|
S. lycopersicum var. humboldtii (VIR2912) К/Д |
19,3 |
14,7 |
17,1 |
120,1 |
|
S. lycopersicumсортБычье сердце К/О |
2,5 |
6,8 |
12,3 |
42,8 |
|
S. lycopersicum сорт Желтый белорус К/О |
0,4 |
13,1 |
16,8 |
28,1 |
|
S. cheesmaniae (VIR3969) К/Д |
9,5 |
32,4 |
23,3 |
130,1 |
|
S. galapagense (VIR3970) К/Д |
34,2 |
46,4 |
65,7 |
84,2 |
|
S. chmielewskii (VIR13725) З/Д |
63,3 |
74,8 |
94,1 |
132,7 |
|
S. chilense (VIR4300) З/Д |
21,8 |
43,7 |
63,2 |
93,3 |
|
S. corneliomulleri (VIR4367) З/Д |
14,9 |
18,7 |
27,2 |
104,8 |
|
S. peruvianum (VIR4361) З/Д |
18,8 |
17,6 |
36,4 |
85,0 |
|
S. peruvianum var. dentatum (VIR3966) З/Д |
50,0 |
84,7 |
87,3 |
119,7 |
|
S. arcanum (VIR13958) З/Д |
66,9 |
65,2 |
37,8 |
44,7 |
|
S. habrochaites (VIR13964) З/Д |
0,2 |
0,2 |
0,3 |
0,3 |
|
S. neorickii (VIR5033) З/Д |
58,0 |
78,4 |
98,1 |
127,3 |
|
Примечание. K — красноплодные, З — зеленоплодные, Д — дикорастущие, О — культивируемые. IG — завязавшийся плод, MG — зеленый плод финального размера, Br — бланжевый плод. Измерения проводились в двух биологических повторностях (в обобщенных пробах). |
|||||
2. Праймеры, разработанные для амплификации и секвенирования генов-гомологов GME1 у исследованных сортов и дикорастущих видов томата (Solanum L.) |
|
Праймер |
Последовательность (5´→ 3´) |
GME F |
CACTGTATTAGTGCCTCATC |
GME R |
CAATTACCAGAATCTAACACATC |
GME1exF |
CAGCTTCAATATGATGGAAGCT |
GME2exF |
CAGCCTGAAAGAAGCTGATG |
GME3exR |
ACCTTTCCAGGTTCCAAAAGG |
GME4exF |
GCACAAACTGCAACGGATAAG |
GME5exR |
ATTGTTGGAGCCCAACCAAG |
GME5exF |
GCCAGTAAACATTGGAAGTGA |
GME4inR |
CTCCAGTAATTCAGAATAAGTGG |
3. Характеристика идентифицированных генов-гомологов GME1 у изученных сортов и дикорастущих видов томата (Solanum L.) |
|||||||||
Вид |
Номер в базе данных Gen-Bank NCBI |
Длина гена, п.н. |
Длина |
1 |
2 |
Изоэлектрическая точка pI |
Молекулярная масса, кДа |
||
интронов общая, п.н. |
кДНК, п.н. |
белка, а.о. |
|||||||
Красноплодные |
|||||||||
Самоопыляемые |
|||||||||
S. lycopersicum сорт Silvestre recordo (VIR1580) |
MK895092 |
1963 |
832 |
1131 |
376 |
0 |
0 |
5,65 |
42,456 |
S. pimpinellifolium var. racemigerum (VIR1018) |
MK895094 |
1963 |
832 |
1131 |
376 |
1 |
0 |
5,65 |
42,456 |
S. cheesmaniae (VIR3969) |
MK895095 |
1963 |
832 |
1131 |
376 |
0 |
0 |
5,65 |
42,456 |
S. galapagense (VIR3970) |
MK895096 |
1962 |
831 |
1131 |
376 |
0 |
0 |
5,65 |
42,456 |
Зеленоплодные |
|||||||||
Самоопыляемые |
|||||||||
S. chmielewskii (VIR13725) |
MK895097 |
1966 |
836 |
1131 |
376 |
0 |
0 |
5,65 |
42,456 |
Перекрестноопыляемые |
|||||||||
S. chilense (VIR4300) |
MK895098 |
1973 |
842 |
1131 |
376 |
10 |
0 |
5,65 |
42,456 |
S. corneliomulleri (VIR4367) |
MK895099 |
1981 |
850 |
1131 |
376 |
10 |
0 |
5,65 |
42,456 |
S. peruvianum (VIR4361) |
MK895100 |
1985 |
854 |
1131 |
376 |
7 |
0 |
5,65 |
42,456 |
S. peruvianum var. dentatum (VIR3966) |
MK895101 |
1982 |
851 |
1131 |
376 |
8 |
1 |
5,64 |
42,515 |
S. arcanum (VIR13958) |
MK895102 |
1982 |
851 |
1131 |
376 |
12 |
0 |
5,65 |
42,456 |
S. habrochaites (VIR13964) |
MK895103 |
1975 |
844 |
1131 |
376 |
10 |
0 |
5,65 |
42,456 |
S. neorickii (VIR5033) |
MK895093 |
1981 |
850 |
1131 |
376 |
13 |
1 |
5,56 |
42,601 |
Примечание. 1 и 2 — число SNPs и аминокислотных замен по сравнению с референсной последовательностью (S. lycopersicum, сорт Heinz 1706; GeneBank NCBI). Данные для сорта Silvestre recordo приведены как типичные для изученных сортов. Описание видов и сортов см. в разделе «Методика» и в таблице 1. |
|||||||||
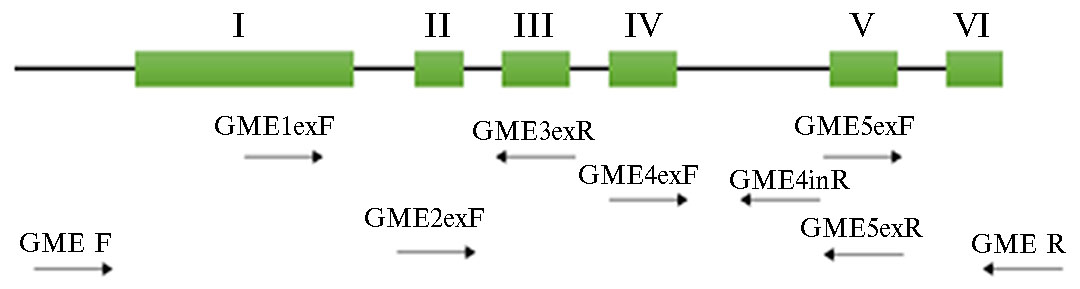
Рис. 1. Схематичное изображение структуры гена GME1, полученной при анализе генов-гомологов GME1 у 11 дикорастущих видов томата и сорта томата овощного Silvestre recordoс использованием разработанных праймеров. Отмечено расположение фланкирующих и внутренних праймеров с локализацией в экзонах (зеленые прямоугольники) и интроне (линии). Римскими цифрами отмечены экзоны. Описание видов и сортов см. в разделе «Методика» и в таблице 1.
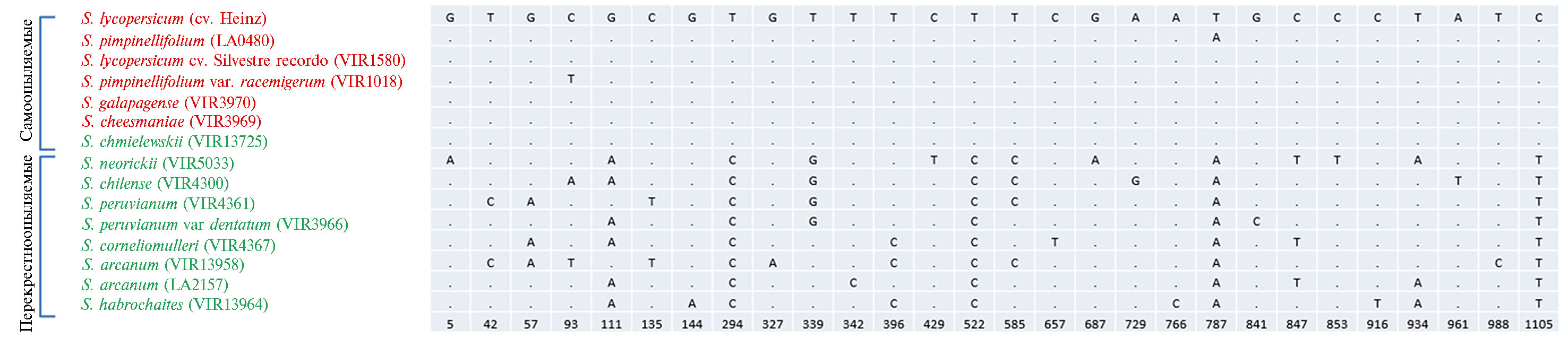
Рис. 2. Распределение SNPs в кодирующей области генов-гомологов GME1 у изученных перекрестно- и самоопыляемых дикорастущих видов и сортов томата (Solanum L.). Числовыми значениями обозначено расположение однонуклеотидных замен. Описание видов и сортов см. в разделе «Методика» и в таблице 1.

Рис. 3. Распределение SNPs в генах-гомологах GME1 у изученных перекрестно- и самоопыляемых дикорастущих видов и сортов томата (Solanum L.). Цветом обозначены красно(оранжево)- и зеленоплодные виды. Числовые значения соответствуют положению выявленных однонуклеотидных замен. Описание видов и сортов см. в разделе «Методика» и в таблице 1.
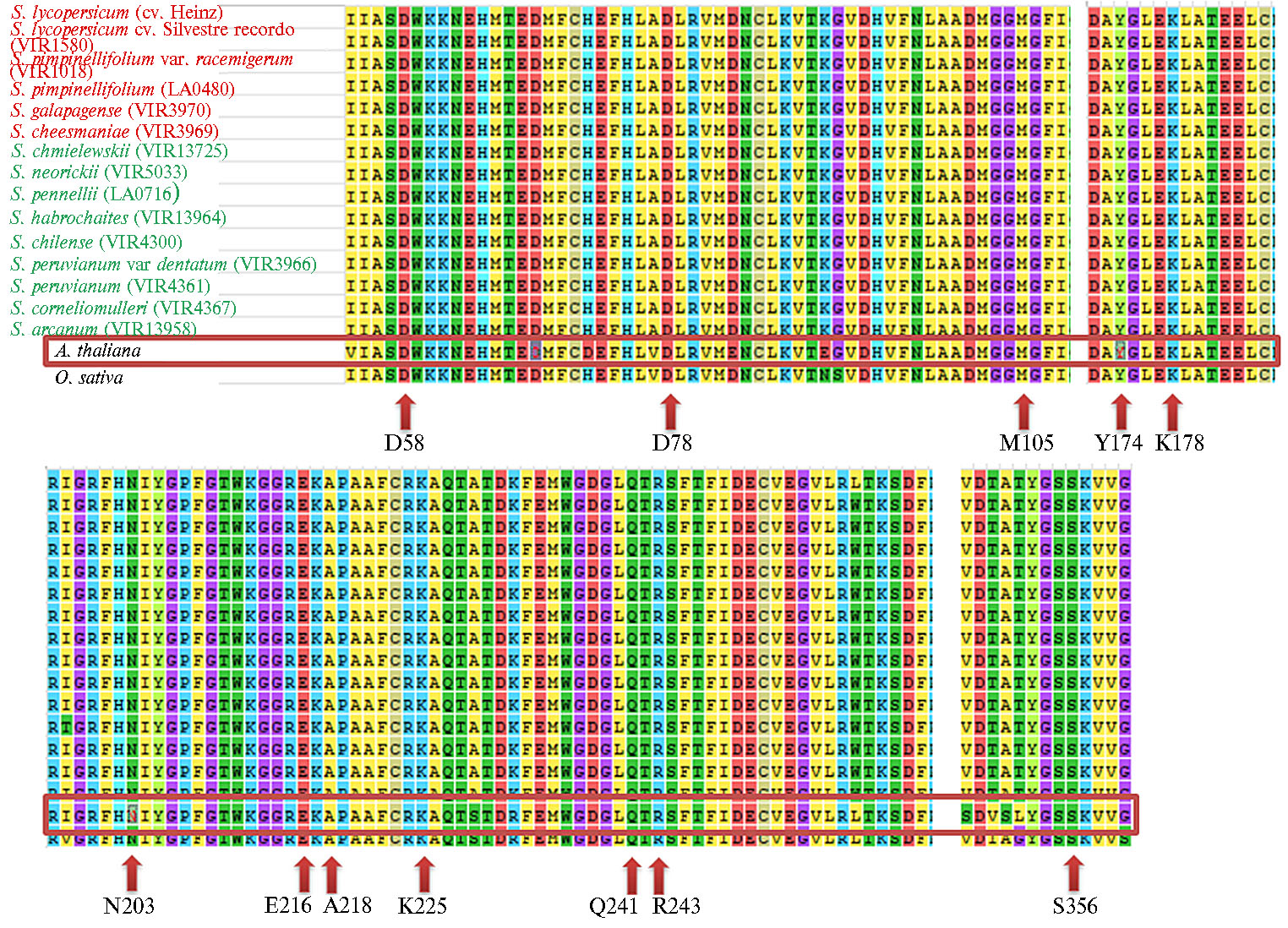
Рис. 4. Консервативные аминокислотные остатки в белках GME1 у изученных сортов и дикорастущих видов томата (Solanum L.), имеющие критическое значение для функциональности фермента GME (GDP-манноза-3´,5´-эпимераза) (указаны стрелками). Для сравнения взяты аминокислотные последовательности гомологов GME риса и арабидопсиса из базы данных NCBI. Описание видов и сортов см. в разделе «Методика» и в таблице 1.
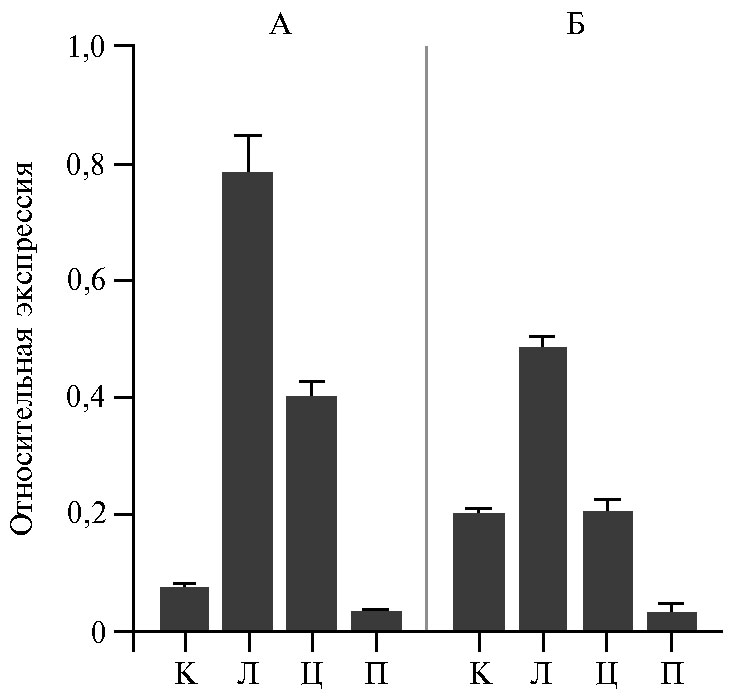
Рис. 5. Экспрессия генов-гомологов GME1 в органах растений томата двух видов с контрастным содержанием аскорбата в плодах: A — Solanum lycopersicum(сорт Silvestre recordo), Б — S. peruvianum; К — корень, Л — лист, Ц — цветок, П — плод в фазу биологической спелости. Нормализацию экспрессии проводили по двум стандартным референсным генам Expressed и Actin 2/7.
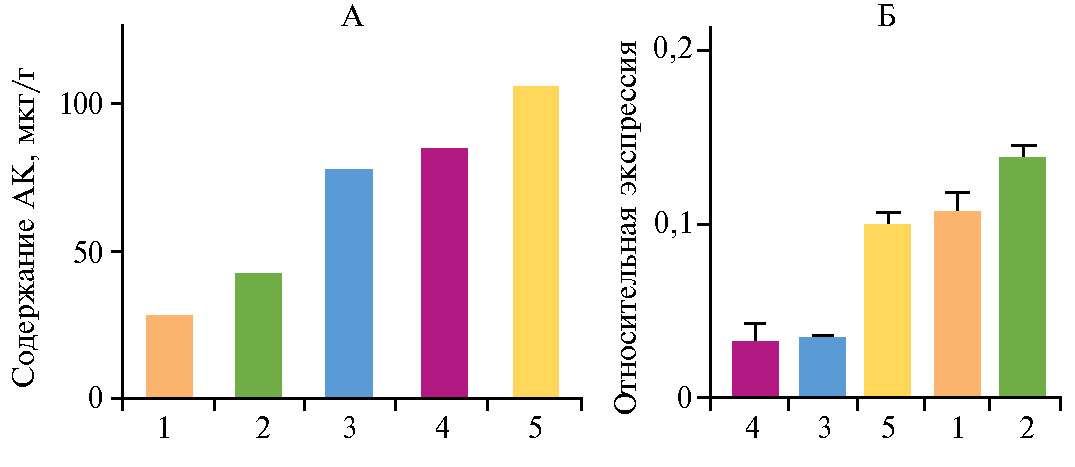
Рис. 6. Содержание аскорбата (А, АК) в сопоставлении с экспрессией гена GME1 (Б) в плодах в фазу биологической спелости у линиии сортов томата овощного Solanum lycopersicum Желтый белорус (1), Бычье сердце (2), Silvestre recordo (3) и образцов дикорастущих видов S. peruvianum (4), S. lycopersicum var. humboldtii (5). Нормализацию экспрессии проводили по двум стандартным референсным генам Expressed и Actin 2/7.
ЛИТЕРАТУРА
- Barth C., De Tullio M., Conklin P. The role of ascorbic acid in the control of flowering time and the onset of senescence. Journal of Experimental Botany, 2006, 57(8): 1657-1665 (doi: 10.1093/jxb/erj198).
- Fotopoulos V., De Tullio M.C., Barnes J., Kanellis K. Altered stomatal dynamics in ascorbate oxidase over-expressing tobacco plants suggest a role for dehydroascorbate signalling. Journal of Experimental Botany, 2008, 59(4): 729-737 (doi: 10.1093/jxb/erm359).
- Foyer C.H., Noctor G. Ascorbate and glutathione: the heart of the redox hub. Plant Physiology, 2011, 155(1): 2-18 (doi: 10.1104/pp.110.167569).
- Fotopoulos V., Kanellis A.K. Altered apoplastic ascorbate redox state in tobacco plants via ascorbate oxidase overexpression results in delayed dark-induced senescence in detached leaves. Plant Physiology and Biochemistry, 2013, 73: 154-160 (doi: 10.1016/j.plaphy.2013.09.002).
- Smirnoff N. Ascorbic acid metabolism and functions: a comparison of plants and mammals. Free Radical Biology and Medicine, 2018, 122: 116-129 (doi: 10.1016/j.freeradbiomed.2018.03.033).
- Conklin P.L., Barth C. Ascorbic acid, a familiar small molecule intertwined in the response of plants to ozone, pathogens, and the onset of senescence. Plant Cell and Environment, 2004, 27(8): 959-970 (doi: 10.1111/j.1365-3040.2004.01203.x).
- Müller-Moulé P., Golan T., Niyogi K.K. Ascorbate-deficient mutants of Arabidopsis grow in high light despite chronic photooxidative stress. Plant Physiology, 2004, 134(3): 1163-1172 (doi: 10.1104/pp.103.032375).
- Linster C.L., Van Schaftingen E. Vitamin C. Biosynthesis, recycling and degradation in mammals. FEBS Journal, 2007, 274(1): 1-22 (doi: 10.1111/j.1742-4658.2006.05607.x).
- Granger M., Eck P. Dietary vitamin C in human health. Advances in Food and Nutrition Research, 2018, 83: 281-310 (doi: 10.1016/bs.afnr.2017.11.006).
- Celma A.R., Cuadros F., López-Rodríguez F. Characterisation of industrial tomato by-products from infrared drying process. Food and Bioproducts Processing, 2009, 87(4): 282-291 (doi: 10.1016/J.FBP.2008.12.003).
- FAOSTAT. Food and Agriculture Organization of the United Nations Statistical Databases, 2019. Режим доступа: http://faostat.fao.org/. Без даты.
- Wang F., Kang S.Z., Du T.S., Li F.S., Qiu R.J. Determination of comprehensive quality index for tomato and its response to different irrigation treatments. Agricultural Water Management, 2011, 98(8): 1228-1238 (doi: 10.1016/j.agwat.2011.03.004).
- Di Matteo A., Sacco A., Anacleria M., Pezzotti M., Delledonne M., Ferrarini A., Frusciante L., Barone A. The ascorbic acid content of tomato fruits is associated with the expression of genes involved in pectin degradation. BMC Plant Biology, 2010, 10(1): 1-11 (doi: 10.1186/1471-2229-10-163).
- Rousseaux M.C., Jones C.M., Adams D., Chetelat R., Bennett A., Powell, A. QTL analysis of fruit antioxidants in tomato using Lycopersicon pennellii introgression lines. Theoretical and Applied Genetics, 2005, 111(7): 1396-1408 (doi: 10.1007/s00122-005-0071-7).
- Schauer N., Semel Y., Roessner U., Gur A., Balbo I., Carrari F., Pleban T., Perez-Melis A., Bruedigam C., Kopka J., Willmitzer L., Zamir D., Fernie A.R. Comprehensive metabolic profiling and phenotyping of interspecific introgression lines for tomato improvement. Nature Biotechnology, 2006, 24(4): 447-454 (doi: 10.1038/nbt1192).
- Schauer N., Semel Y., Balbo I., Steinfath M., Repsilber D., Selbig J., Pleban T., Zamir D., Fernie A.R. Mode of inheritance of primary metabolic traits in tomato. The Plant Cell, 2008, 20(3): 509-523 (doi: 10.1105/tpc.107.056523).
- Stevens R., Buret M., Duffe P., Garchery C., Baldet P., Rothan C., Causse M. Candidate genes and quantitative trait loci affecting fruit ascorbic acid content in three tomato populations. Plant Physiology, 2007, 143(4): 1943-1953 (doi: 10.1104/pp.106.091413).
- Stevens R., Page D., Gouble B., Garchery C., Zamir D., Causse M. Tomato fruit ascorbic acid content is linked with monodehydroascorbate reductase activity and tolerance to chilling stress. Plant, Cell and Environment, 2008, 31(8): 1086-1096 (doi: 10.1111/j.1365-3040.2008.01824.x).
- Zou L.P., Li H.X., Ouyang B., Zhang J.H., Ye Z.B. Cloning, expression, and mapping of GDP-D-mannose pyrophosphorylase cDNA from tomato (Lycopersicon esculentum). Yi Chuan Xue Bao, 2006, 33(8): 757-764 (doi: 10.1016/S0379-4172(06)60108-X).
- Gilbert L., Alhagdow M., Nunes-Nesi A., Quemener B., Guillon F., Bouchet B., Faurobert M., Gouble B., Page D., Garcia V., Petit J. GDP-D-mannose 3,5-epimerase (GME) plays a key role at the intersection of ascorbate and non-cellulosic cell-wall biosynthesis in tomato. The Plant Journal, 2009, 60(3): 499-508 (doi: 10.1111/j.1365-313X.2009.03972.x).
- Zhang C., Liu J., Zhang Y., Cai X., Gong P., Zhang J., Wang T., Li H., Ye Z. Overexpression of SlGMEs leads to ascorbate accumulation with enhanced oxidative stress, cold, and salt tolerance in tomato. Plant Cell Rep., 2011, 30(3): 389-398 (doi: 10.1007/s00299-010-0939-0).
- Li M., Ma F., Liang D., Li J., Wang Y. Ascorbate biosynthesis during early fruit development is the main reason for its accumulation in kiwi. PLoS ONE, 2010, 5(12): 14281 (doi: 10.1371/journal.pone.0014281).
- Liu F., Wang L., Gu L., Zhao W., Su H., Cheng X. Higher transcription levels in ascorbic acid biosynthetic and recycling genes were associated with higher ascorbic acid accumulation in blueberry. Food Chemistry, 2015, 188: 399-405 (doi: 10.1016/j.foodchem.2015.05.036).
- Tao J., Wu H., Li Z., Huang C., Xu X. Molecular evolution of GDP-D-mannose epimerase (GME), a key gene in plant ascorbic acid biosynthesis. Frontiers in Plant Science, 2018, 9: 1293 (doi: 10.3389/fpls.2018.01293).
- Giovanelli G., Zanoni B., Lavelli V., Nani R. Water sorption, drying and antioxidant properties of dried tomato products. Journal of Food Engineering, 2002, 52(2): 135-141 (doi: 10.1016/S0260-8774(01)00095-4).
- Edwards K., Johnstone C., Thompson C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Research, 1991, 19(6): 1349 (doi: 10.1093/nar/19.6.1349).
- González-Aguilera K.L., Saad C.F., Chávez Montes R.A., Alves-Ferreira M., de Folter S. Selection of reference genes for quantitative real-time RT-PCR studies in tomato fruit of the genotype MT-Rg1. Frontiers in Plant Science, 2016, 7: 1386 (doi: 10.3389/fpls.2016.01386).
- Slugina M.A., Filyushin M.A., Shchennikova A.V., Kochieva E.Z., Skryabin K.G. FAS, YABBY2, and YABBY5 gene expression profile correlates with different fruit locule number in tomato. Russian Journal of Genetics, 2020, 56(4): 401-407 (doi: 10.31857/S0016675820030157).
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874 (doi: 10.1093/molbev/msw054).
- Choi Y., Sims G.E., Murphy S., Miller J.R., Chan A.P. Predicting the functional effect of amino acid substitutions and indels. PLoS ONE, 2012, 7(10): e46688 (doi: 10.1371/journal.pone.0046688).
- Bailey T.L., Boden M., Buske F.A. “MEME SUITE: tools for motif discovery and searching”. Nucleic Acids Research, 2009, 37(2): 202-208 (doi: 10.1093/nar/gkp335).
- Mellidou I., Koukounaras A., Kostas S., Patelou E., Kanellis A.K. Regulation of vitamin C accumulation for improved tomato fruit quality and alleviation of abiotic stress. Genes, 2021, 2(5): 694 (doi: 10.3390/genes12050694).
- The 100 Tomato Genome Sequencing Consortium, 2014. Режим доступа: https://solgenomics.net/projects/tomato100/. Без даты.
- Wolucka B.A., Van Montagu M. The VTC2 cycle and the de novo biosynthesis pathways for vitamin C in plants: an opinion. Phytochemistry, 2007, 68(21): 2602-2613 (doi: 10.1016/j.phytochem.2007.08.034).
- Bairoch A., Apweiler R., Wu C.H., Barker W.C., Boeckmann B., Ferro S., Gasteiger E., Huang H., Lopez R., Magrane M., Martin M.J., Natale D.A., O'Donovan C., Redaschi N., Yeh L.S. The Universal Protein Resource (UniProt). Nucleic Acids Research, 2004, 33(1): 154-159 (doi: 10.1093/nar/gki070).
- Major L.L., Wolucka B.A., Naismith J.H. Structure and function of GDP-mannose-3',5'-epimerase: an enzyme which performs three chemical reactions at the same active site. Journal of the American Chemical Society, 2005, 127(51): 18309-18320 (doi: 10.1021/ja056490i).
- Wierenga R.K., De Maeyer M.C.H., Hol W.G.J. Interaction of pyrophosphate moieties with α-helixes in dinucleotide binding proteins. Biochemistry, 1985, 24(6): 1346-1357 (doi: 10.1021/bi00327a012).
- Mounet-Gilbert L., Dumont M., Ferrand C., Bournonville C., Monier A., Jorly J., Lemaire-Chamley M., Mori K., Atienza I., Hernould M., Stevens R. Two tomato GDP-D-mannose epimerase isoforms involved in ascorbate biosynthesis play specific roles in cell wall biosynthesis and development. Journal of Experimental Botany, 2016, 67(15): 4767-4777 (doi: 10.1093/jxb/erw260).
- Munir S., Mumtaz M.A., Ahiakpa J.K., Liu G., Chen W., Zhou G., Zheng W., Ye Z., Zhang Y. Genome-wide analysis of Myo-inositol oxygenase gene family in tomato reveals their involvement in ascorbic acid accumulation. BMC Genomics, 2020, 21(1): 1-15 (doi: 10.1186/s12864-020-6708-8).