doi: 10.15389/agrobiology.2021.5.823rus
УДК 631.522/.524:575:577.21
Работа выполнена при поддержке РНФ (грант № 21-16-0008), РФФИ (грант № 18-29-07007) и Министерства науки и высшего образования РФ.
ФАКТОРЫ ТРАНСКРИПЦИИ СЕМЕЙСТВА MADS РАСТЕНИЙ: СВЯЗЬ С ПРИЗНАКАМИ ДОМЕСТИКАЦИИ И ПЕРСПЕКТИВЫ ДЛЯ СЕЛЕКЦИИ (обзор)
А.В. НЕЖДАНОВА, А.В. ЩЕННИКОВА✉
Признаки доместикации, которые подразделяются на три группы (продуктивность, адаптивность и воспроизводство) и составляют в совокупности доместикацонный синдром, сближающий таксономически удаленные одомашненные формы, остаются хозяйственно значимыми и у современных возделываемых культур. Значительная часть генов, контролирующих у растений признаки доместикации, представлена генами факторов регуляции транскрипции, в частности принадлежащих семейству белков с MADS-доменом. MADS-белки служат ключевыми регуляторами практически всех аспектов репродуктивного развития растений, включая определение сроков цветения, строения соцветий, идентичности цветковых органов, развития корней, плодов и семян, а также адаптивной и стрессовой реакции растений на неблагоприятные условия окружающей среды. В представленном обзоре показано возможное участие MADS-box генов в процессах, происходивших при одомашнивании растений. Обсуждается роль MADS-box генов в реакции растений на длительное воздействие холодом (яровизацию), в регуляции состояния физиологического покоя почек, в формировании структуры соцветия и цветка, изменения фертильности растения и качественных признаков плода (процесс созревания, синтез каротиноидов и антоцианов, число семян, способность к растрескиванию, сроки хранения), а также в ответе растений на стрессы (засоление, засуха, изменение температуры). Рассмотрено явление плейотропии и избыточности функций MADS-box генов (за счет существования паралогов). Высказываетcя предположение, что высокий структурно-функциональный консерватизм может свидетельствовать о высоком потенциале MADS-box генов как инструментов для предсказуемой тонкой настройки фенотипов сельскохозяйственных культур посредством комбинирования (в том числе, дозозависимого) различных аллелей и паралогов MADS-box генов. Еще один возможный способ такой настройки — разделение плейотропных функций MADS-box гена посредством введения мутаций в его кодирующую или цис-регуляторную последовательность для изменения взаимодействий белок-белок или белок-ДНК, а также профиля и(или) уровня экспрессии, в том числе в ответ на различные внешние и внутренние сигналы. Сделано заключение о том, что фундаментальные и прикладные исследования MADS-box генов у различных видов растений (как дикорастущих, так и культурных) не только приведут к более глубокому пониманию эволюции и развития современных растений, но также внесут большой вклад в улучшение сельскохозяйственных культур, в том числе с помощью CRISPR/Cas и других современных технологий.
Ключевые слова: регуляция транскрипции, факторы транскрипции, MADS-box гены, консерватизм, плейотропный эффект, признаки одомашнивания, продуктивность, адаптация, воспроизводство, хозяйственно ценные признаки, целевые гены.
Сельскохозяйственные культуры появились в результате одомашнивания, при котором дикие виды растений адаптировались к выращиванию человеком в процессе совместной эволюции с ним (1). При этом у доместицированных форм развились признаки, полезные для объема, качества, сбора и сроков хранения урожая, а также для адаптации к влиянию окружающей среды (1-4). В совокупности они составляют доместикационный синдром, сближающий таксономически удаленные одомашненные формы, и подразделяются на три группы — признаки продуктивности, адаптивности и воспроизводства (5, 6), которые остаются хозяйственно значимыми и у современных возделываемых культур. По разным оценкам, в настоящее время известно от 1000 до 2500 полу- и полностью одомашненных видов растений из 120-160 семейств (2, 7). Благодаря интенсивным исследованиям в области генетики, геномики и археологии, постепенно формируется взгляд на то, как происходила доместикация (2, 3, 8-10), что представляет интерес как для углубления понимания эволюционных событий, так и для современных селекционных программ, основанных на знании молекулярно-генетических особенностей регуляции хозяйственно ценных признаков. Более того, предполагается, что понимание эволюционного происхождения и регуляции ключевых признаков доместикации может помочь не только в усовершенствовании существующих и выведении новых сортов, но даже при одомашнивании новых видов растений (10).
Наблюдения, основанные на археоботанических исследованиях, популяционном геномном анализе и изучении древней ДНК, показали, что формирование фенотипа различных культур с закреплением ключевых признаков занимает около 2-3 тыс. лет (3, 10-14). Основными причинами такого длительного процесса считают поток генов между популяциями зарождающихся домашних растений и их диких предков (15, 16), а также полигенную природу многих признаков (17). Кроме того, предполагается, что, хотя некоторые характеристики (например, окраска и вкус) обусловлены, скорее всего, осознанным выбором человека, большая часть признаков доместикации (устойчивость к осыпанию семян, синхронное прорастание и др.) была сначала результатом бессознательного отбора, происходившего подобно естественному (10, 17). В качестве механизма одомашнивания растений рассматривается интрогрессивная гибридизация между дикими родственниками (18), благодаря которой и сегодня происходит диверсификация сельскохозяйственных культур (19), например банана Musa spp. (20), пшеницы Triticum aestivum (21), риса Oryza sativa (22), кукурузы Zea mays (23), ячменя Hordeum vulgare (24), яблони Mlus domestica (25) и других многолетных плодовых культур (26).
Интересно, что гены, лежащие в основе признаков одомашнивания и диверсификации у различных видов растений, во многих случаях одни и те же или близкородственные (4, 5, 9, 27). Это составляет основу для использования эволюционной гомологии с целью передачи желаемых признаков множеству видов, в том числе с помощью новых технологий (например, CRISPR/Cas), которые позволяют повторять генетические этапы доместикации (28-30).
Значительную часть идентифицированных генов, связанных с признаками одомашнивания, составляют гены регуляторов транскрипции. Хотя в геноме растений на них приходится только около 5 % белок-кодирующих генов, изменения в них могут повлиять на целый набор свойств в относительно короткие сроки (31-33).
В качестве эволюционных мишеней часто рассматривают обнаруженные практически у всех эукариот гены факторов транскрипции с доменом MADS, который кодирует консервативная последовательность нуклеотидов MADS-box (MADS-box гены). Считается, что дупликация предшественников MADS-box генов и последующая диверсификация и нео- и субфункционализация дупликатов играли и играют одну из ключевых ролей в эволюции и разнообразии растений (33-35). В то время как дупликаты большинства генов теряют свои функции, гены транскрипционных факторов, в том числе факторов регуляции транскрипции с MADS доменом, сохраняют и обновляют функции после дупликации, что способствует расширению генетических возможностей для возникновения эволюционных новшеств (36). Более того, анализ геномов трех видов перца — C. baccatum, C. chinense и C. annuum показал, что MADS-box гены входят в десятку генных семейств с наибольшей массовой дупликацией (37), что свидетельствует о ключевых позициях, занимаемых этими генами в эволюции и диверсификации растений. У растений MADS-факторы транскрипции служат ключевыми регуляторами практически всех аспектов репродуктивного развития, включая определение сроков цветения, архитектуры соцветий и цветков, формирования пыльцы, семян, плодов, а также развития корней (38) и ответа растений на различные стрессы (39). Все это является еще одним подтверждением важности MADS-box генов как объектов отбора во время доместикации культур. Таким образом, данные функционального анализа MADS-box генов в сочетании с имеющимися биоресурсами могут быть использованы для улучшения различных репродуктивных признаков сельскохозяйственных культур с помощью современных молекулярных технологий селекции.
Настоящий обзор сфокусирован на семействе MADS-факторов транскрипции и их участии в формировании признаков продуктивности, адаптивности и воспроизводства у растений.
С е м е й с т в о MADS-ф а к т о р о в р е г у л я ц и и т р а н с к р и п ц и и. Аббревиатура MADS происходит от названий основателей семейства: MINICHROMOSOME MAINTENANCE 1 (MCM1) (Saccharomyces cerevisiae), AGAMOUS (AG) (Arabidopsis thaliana), DEFICIENS (DEF) (Antirrhinum majus) и SERUM RESPONSE FACTOR (SRF) (Homo sapiens) (38). MADS-факторы транскрипции (MADS-domain transcription factors) характеризуются присутствием высококонсервативного ДНК-связывающего N-концевого MADS-домена (38).
В растениях MADS-факторы транскрипции представлены двумя структурными типами: тип I — в состав белка входит MADS-домен MADS_SRF_like (NCBI: cd00266); тип II, или MIKC, — в состав белка входят MADS-домен MADS_MEF2_like (NCBI: cd00265), междоменный I-участок, консервативный кератин-подобный К-домен K-box (NCBI: pfam01486) и вариабельная С-область, расположенные последовательно (40). Первыми были выделены MIKC-гены, при нокауте которых происходит полное или частичное гомеотическое превращение одних органов цветка в другие. Так, потеря функции DEF или его ортолога APETALA3 (AP3) у Arabidopsis приводит к развитию чашелистиков вместо лепестков и плодолистиков вместо тычинок, а мутация agamous-1 вызывает превращение тычинок в лепестки (41, 42).
Всего в геноме модельного растения A. thaliana насчитывается 107 MADS-box генов (40); в геномах других видов растений, включая сельскохозяйственные культуры, их от 23 до 367 (табл.). MADS-box семейство делится на подсемейства, большая часть которых сохраняется на протяжении всей эволюции семенных растений (рис.), а функции генов внутри каждого подсемейства у разных видов растений часто гомологичны (40, 43). К примеру, в геноме всех видов цветковых растений присутствуют ортологи генов AP3/DEF и AGAMOUS (AG), участвующие в развитии репродуктивных органов (35, 40, 43), а также гена времени цветения SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1 (SOC1, или AGL20) (44).
Присутствие в геноме растения дупликатов MADS-box генов, претерпевших субфункционализацию, приводит к избыточности функции (45). Так, AG участвует в репродуктивном развитии цветка, а его паралоги SHATTERPROOF1 (SHP1) и SHP2 — в развитии семяпочек (46). Это обусловлено различиями в паттернах экспрессии указанных генов, так как сверхэкспрессия SHP1 и SHP2 в растениях Arabidopsis с мутацией ag способна восстанавливать развитие тычинок и плодолистиков (46).
Два гена, участвующие в контроле идентичности цветковой меристемы, — APETALA1 (AP1) и CAULIFLOWER (CAL), напротив, имеют сходные паттерны экспрессии, но различаются функционально, частично дублируя друг друга (47). В то время как растения с мутацией ap1 демонстрируют сильные дефекты идентичности цветковой меристемы и органов цветка, фенотип растений cal подобен дикому типу, а соцветия с двойной мутацией ap1 cal имеют фенотип «цветной капусты» (47, 48). Показано, что функциональное различие между CAL и AP1 отчасти связано с несколькими замещениями аминокислотных остатков, которые изменяют паттерн белок-белковых взаимодействий (49).
Результаты интенсивных исследований эволюции MADS-box генов и их вклада в эволюцию и диверсификацию цветковых растений позволяют сделать предположения о роли MADS-box генов в одомашнивании цветковых растений (33-35). Далее мы рассмотрим хозяйственно ценные признаки современных культур и их связь с MADS-box генами.
В р е м я ц в е т е н и я р а с т е н и й. Я р о в и з а ц и я. При переходе растения от вегетативного роста к репродуктивному развитию апикальная меристема побега приобретает статус меристемы соцветия, на периферии которой формируются цветковые меристемы. Контроль этого процесса — одна из мишеней адаптационных механизмов (50). Анализ естественных вариаций, мутаций и трансгенных растений A. thaliana, которые цветут позже или раньше, чем дикий тип, выявил генные локусы, вовлеченные в регуляцию времени цветения (50).
На сегодня известны шесть основных сигнальных путей, под влиянием которых происходит переход к цветению. Из них три (автономный путь, возрастной путь и путь, опосредованный гиббереллинами) в основном не зависят от внешних сигналов, а четвертый (путь фотопериода) запускает или отменяет цветение, реагируя на изменения длины дня. Оставшиеся два пути — температурозависимые. Совместно сигнальные пути управляют основными регуляторами времени цветения — MADS-box генами FLOWERING LOCUST (FT), FLOWERING LOCUSD (FD) и SOC 1, продукты которых активируют транскрипцию генов идентичности меристем соцветия и цветка (44, 51).
Температурозависимый сигнальный путь яровизации, отражающий восприимчивость растения к длительному воздействию холода, эффективно задействует MADS-box гены (51-54). Синдром яровизации у растений, вероятно, возник как адаптация к сезонным холодам и местным климатическим условиям (52) и важен при выращивании сельскохозяйственных культур: нечувствительные к яровизации яровые сорта высевают весной, тогда как чувствительные озимые сорта — осенью (55). Таким образом, яровизация, очевидно, была целью искусственного отбора при доместикации однодольных и двудольных культур, а ключевыми мишенями при этом оказались MADS-box гены.
Один из них — репрессор цветения FLOWERING LOCUS C (FLC, или FLF): у Arabidopsis фактор FLC подавляет транскрипцию генов центральных стимуляторов цветения SOC1, FT и FD (44, 51, 53). Длительный холод (яровизация) препятствует экспрессии FLC, включая эпигенетические механизмы, в частности модификацию гистонов в локусе FLC, что, в свою очередь, позволяет активировать гены, способствующие цветению (54). Генетическая вариабельность FLC, определяющая количество и активность синтезируемого белка, способна изменять потребность в яровизации у различных экотипов Arabidopsis (54). Следовательно, вариации в ортологах FLC могли сыграть важную роль в адаптации сельскохозяйственных культур к различным климатическим условиям (56).
Яркий пример важности генов FLC — виды рода Brassica L. (семейство Brassicaceae — Капустные) (57). Так, раннее цветение у китайской капусты Brassica rapa ssp. pekinensis (листовые овощи) снижает качество урожая. У масличных культур (масличный рапс Brassica napus и полевая горчица Brassica rapa ssp. oleifera) существуют сорта озимого и ярового типа, пригодные для адаптации репродуктивного развития к различным условиям окружающей среды. Корнеплоды (репа Brassica rapa ssp. rapa) и, наконец, капуста огородная (Brassicaoleracea, разновидности — кочанная капуста var. oleracea,брокколи var. italica, цветная капуста var. botrytis) также подвержены температурозависимой регуляции времени цветения.
В геноме Brassica идентифицированы четыре ортолога FLC (BrFLC1, BrFLC2, BrFLC3 и BrFLC5), вариации которых определяют различия во времени цветения сортов репы (56, 58-60). Ген BrFLC1 ассоциирован с поздним цветением китайской капусты (58), а мутация в последовательности BrFLC2 — с ускоренным цветением рапса (61). Вариации гена FLC отвечают за различия между яровыми и озимыми сортами рапса (56) и изменение времени цветения у брокколи (62). Отметим, что в геноме B. napus, который возник в результате аллоплоидии между палеополиплоидными предками B. rapa и B. oleracea, гены времени цветения представлены избыточно; в частности, у FLC имеется девять идентифицируемых копий (57). Очевидно, что в процессе образования видов Brassica и их доместикации ряд молекулярных изменений в ортологах FLCи присутствие нескольких паралогов FLC способствовали различиям в чувствительности к яровизации и времени цветения.
Ортологи FLC идентифицированы и у злаков — ячменя (H. vulgare), пшеницы (T. aestivum), риса (O. sativum) и кукурузы (Z. mays) (63). Внутри клады FLC однодольных есть субклады OsMADS51 и OsMADS37; субклада OsMADS51 разделена на две группы — ODDSOC1 и ODDSOC2 (63). У пшеницы гомологи ODDSOC2 — TaAGL42 и TaAGL33 характеризуются разными профилями экспрессии у яровых и озимых сортов (63, 64). Это предполагает, что члены группы ODDSOC2 были частью адаптивного механизма, с помощью которого разные популяции злаков приобретали различные потребности в яровизации (63, 64).
В геноме A. thaliana имеется пять паралогов FLC: MADS AFFECTING FLOWERING2 (MAF2, или AGL31), MAF3 (AGL70), MAF4 (AGL69), MAF5 и FLOWERING LOCUS M (FLM, или MAF1, AGL27) (63). Исследование разных популяций A. thaliana, представляющих генетическое разнообразие вида, подтвердило, что локусы количественных признаков (QTL) цветения включают все три вида FLC-подобных генов (FLC, FLM и MAF2-5) (65). Кроме того, в температурозависимой регуляции цветения активно участвуют MADS-box гены клады StMADS11, которая у A. thaliana представлена генами AGAMOUS-like 24 (AGL24) и SHORTVEGATATIVEPHASE (SVP) (34, 44).
В зависимости от температуры ген FLMимеет различные формы сплайсинга, две из которых генерируют два разных белка — FLM-β и FLM-δ (34). FLM-β считается основной функциональной формой FLM, отвечающей за отклик на температуру (34). Предполагается, что комплекс SVP/FLM-β связывается с промоторами генов-мишеней, таких как индуктор цветения SOC1, репрессируя цветение, а комплекс SVP/FLM-δ связываться с ДНК не может и, конкурируя с SVP/FLM-β, выступает как непрямой индуктор цветения (34). Количество FLM-β увеличивается при низкой температуре, а повышенные температуры дестабилизируют белок SVP, из чего следует, что более высокие температуры благоприятствуют цветению из-за уменьшения образования комплекса SVP/FLM-β (34).
Тандемно расположенные гены MAF2-5 служат репрессорами цветения (34). Ген MAF2 предотвращает раннее цветение в ответ на короткие периоды холода, что позволяет избежать индукции цветения в теплый осенний период перед зимними холодами (66). Для MAF2 и MAF3 подобно FLM свойственен температурозависимый альтернативный сплайсинг (67, 68). Низкотемпературная форма MAF2 кодирует белок, который взаимодействует с SVP, подавляя цветение; при повышенных температурах сплайсинг смещается в сторону варианта, который кодирует белок, не взаимодействующий с SVP; таким образом, при более низких температурах MAF2 и SVP подавляют цветение одновременно с FLM и SVP (68). Это также может быть справедливо для других генов MAF, однако активность генов MAF не является избыточной, что подтверждается анализом мутантных фенотипов по отдельным генам (68). Тандемные гены особенно восприимчивы к перегруппировкам последовательностей при неаллельной гомологичной рекомбинации (69). Подобные структурные отклонения в кластере MAF2-5 могли служить инструментом при адаптации видов к различным климатическим условиям.
Помимо ортологов и паралогов FLC, яровизация пшеницы в значительной степени регулируется тремя генами VERNALIZATION (VRN), два из которых — VRN1 и VRN3 представляют собой MADS-box гены (63). Яровизация приводит к росту экспрессии стимулятора цветения VRN1 (ортолог АР1), продукт которого подавляет транскрипцию гена репрессора цветения VRN2, смягчая репрессивный эффект, оказываемый этим геном на стимулятор цветения VRN3 (ортолог FT); VRN3 затем положительно регулирует экспрессию VRN1, что приводит к положительной обратной связи и вызывает цветение (63). С учетом того, что клада FLC существует у однодольных, представляется вероятным, что в геноме предковых видов двудольных и однодольных присутствовали как FLC-, так и AP1/VRN1-подобные гены, и каждая группа по-разному пополнялась в ходе развития восприимчивости к яровизации (63). Большое разнообразие ответа на яровизацию у образцов пшеницы, ячменя и райграса (Lolium perenne) вызвано различными мутациями в регуляторных областях ортологов VRN1 (70-72). Интересно, что MADS-box ген VRN4 пшеницы, который появился в результате дупликации гена VRN1,присутствует не во всех образцах пшеницы; его активностьснижает потребность в яровизации, что может быть использовано селекционерами для модуляции реакции яровизации (73).
С о с т о я н и е ф и з и о л о г и ч е с к о г о п о к о я п о ч е к. Для сельского хозяйства важно получение плодовых деревьев, адаптированных к местным климатическим условиям по срокам выхода из состояния покоя. Индукция покоя почек за счет зимних холодов представляет собой адаптивную особенность многолетних растений умеренного климата, которая обеспечивает оптимальную защиту вегетативных и репродуктивных меристем от низких температур (74).
Подобно регуляции цветения с помощью яровизации, выход почек древесных растений из состояния покоя требует воздействия определенного периода низких температур, и MADS-box гены DORMANCY-ASSOCIATED MADS-BOX(DAM, ортологи SVP и AGL24)относятся к ключевым регуляторам этого процесса (74, 75).
В геноме персика (Prunus persica) присутствует кластер из шести тандемно расположенных генов PpDAM1-PpDAM6, которые считаются одним из наиболее важных генетических элементов, лежащих в основе ответа на яровизацию (74, 75). У яблони (Malus × domestica) и груши (Pyrus communis) основные QTL, ассоциированные с ответом на холод и состоянием покоя почек, также связаны с локусами генов DAM (75).
Таким образом, считается, что гены DAM сыграли ключевую роль в точной настройке времени цветения и адаптации к различным климатическим зонам у культивируемых растений. Также показано, что, помимо генов DAM, сверхэкспрессия MADS-box гена BpMADS4 (подсемейство FUL) березы (Betula pendula) в растениях тополя (Populus tremula) приводит к задержке зимнего перехода почек в состояние покоя (76).
С т р о е н и е с о ц в е т и я. Важным целевым признаком для повышения урожайности при доместикации растений была структура соцветия (9).
Ярким примером служит кочан цветной капусты и брокколи, состоящий из плотной массы соцветий с задержкой развития соответственно на очень ранней и более поздней стадии, а также множество сортов с промежуточным фенотипом (77, 78). Фенотип цветной капусты у мутантных растений Arabidopsis объясняется двойной мутацией ap1 cal (47). Подобно этому, у сортов цветной капусты и брокколи нарушены строение и функция MADS-box генов BoCAL и BoAP1 (79). Это свидетельствует об отборе определенных аллелей BoCAL и BoAP1, в результате которого были получены растения с модифицированными соцветиями (78). При этом существование в геноме B. oleracea нескольких паралогов AP1 может определять различия между неодинаковыми фенотипами по соцветиям (78, 79).
Другой пример — разветвленные соцветия у томата (Solanum lyco-persicum), формирование которых находится под контролем MADS-box генов подсемейства SEPALLATA1 (SEP1): JOINTLESS2 (J2), ENHANCER OF JOINTLESS2 (EJ2) и LONG INFLORESCENCE (LIN) (80). Ветвление соцветий томата обычно сопровождается высоким процентом пустоцветов, а комбинации разных мутантных аллелей J2, EJ2 и LIN в зависимости от дозы гена способны уменьшить ветвление и одновременно увеличить скорость плодоношения, что повышает урожайность (80).
Еще один важный целевой признак — ветвление побегов (кущение) (81). Показано, что MADS-box ген OsMADS57 (подсемейство AGL17) влияет на кущение растений риса O. sativa. Мутация фактора транскрипции OsMADS57, связанная с отсутствием C-концевой области, значительно увеличивает кущение растения риса и, таким образом, повышает урожай зерна (82).
Ген OsMADS1 (подсемейство SEP1), сверхэкспрессия которого приводит к карликовости растений риса (83), может быть еще одной мишенью для изменения архитектуры соцветия.
С т р о е н и е ц в е т к а. С т е р и л ь н о с т ь. Согласно модели ABCDE, закладка цветковых органов определяется комбинаторным взаимодействием генов пяти различных классов активности: идентичность чашелистиков определяется генами классов A и Е, лепестков — A, B и Е, тычинок — B, C и Е, плодолистиков — C и Е, а семязачатков — С, Е и D (35). Почти все гены ABCDE кодируют содержащие MADS-домены транскрипционные факторы MIKC-типа. У Arabidopsis это AP1 (класс A); AP3 и PISTILLATA (PI) (B); AG, SHP1 и SHP2 (C), SEEDSTICK (STK) (D); SEP1, SEP2, SEP3 и SEP4 (E) (35). Потеря функции этих генов приводит к гомеотическим превращениям цветка. Например, мутация ag вызывает замену тычинок лепестками, а плодолистиков — новыми цветками с той же моделью развития (41). Такие фенотипы привлекательны с точки зрения селекции декоративных растений. Например, мутации ag, в том числе в цис-регуляторных областях гена (изменяющие профиль его экспрессии), служат причиной формирования махровых цветков у декоративных разновидностей японской вишни Prunus lannesiana и розы Rosas pp. (84, 85). У яблони подавление активности ортологов AG (MdMADS15 и MdMADS22) приводит к появлению декоративно привлекательных цветущих деревьев и уменьшению количества семян за счет мужской стерильности (86).
Бессемянность и мужская стерильность плодов яблони наблюдаются также в случае нокаута ортолога PI, MdPI, когда цветки формируют чашелистики вместо лепестков и плодолистики вместо тычинок (87).
Мужская стерильность и предотвращение ауткроссинга желательны для многих культур, так как эти признаки позволяют избежать потока генов между возделываемыми растениями и их родственными дикими видами. Способом сдерживания генов при одновременном сохранении мужской фертильности служит предотвращение раскрытия цветка (клейстогамия). Это показано на примере риса, цветки которого раскрываются под воздействием лодикул — органов, гомологичных лепесткам. Мутантные аллели ортолога AP3 риса SUPERWOMAN1 (SPW1, или OsMADS16), в зависимости от силы аллеля, могут вызывать ряд фенотипических изменений, включая мужскую стерильность и клейстогамию (88).
В ряде исследований определена ключевая роль содержащих MADS-домены белков типа I в регуляции размножения растений (в частности, в определении развития женского гаметофита, эмбрионов и эндосперма) и их решающее значение для установления репродуктивных границ между видами растений (89).
К а ч е с т в о п л о д о в и с е м я н. Качество плодов — один из основных признаков доместикации растений, включая изменение числа и размера плодов, количества семян, способности к растрескиванию, скорости созревания, сроков хранения и годности, а также внешней привлекательности и вкусовых характеристик плода. Как показывают результаты многочисленных исследований, ключевая регуляторная роль в формировании этих признаков и здесь принадлежит MADS-box генам. Так, у Arabidopsis гены SHP1, SHP2 и STK избыточно определяют идентичность семяпочек; тройная мутация stk shp1 shp2 приводит к абортированию или отсутствию семян (46).
Бессемянность относится к желательным признакам при селекционном улучшении сочных плодов, когда она способствует продлению сроков их хранения, а также использования для производства соков. Причина бессемянности у сортов винограда (Vitis vinifera) заключается в пониженном уровне экспрессии ортолога STK — VviAGL11 за счет изменений в его промоторе, где длина повторов обратно коррелирует с развитием семян (90). Генетическая характеристика локуса VviAGL11 позволяет виноделам и селекционерам оценивать растения на количество семян в плодах до достижения репродуктивной стадии (91). Уровни экспрессии ортолога STK у томата — SlyAGL11 положительно коррелируют со степенью развития семян; нокаут SlyAGL11 приводит к появлению плодов без семян (92). Подавление экспрессии ортологов STKу петунии (Petunia× hybrida) — FBP7 и FBP11 приводило к полной замене семязачатков на структуры, подобные плодолистикам (93). Таким образом, ортологи STK у различных видов растений могут быть использованы в селекции с целью сокращения числа семян.
Растрескивание плодов приводит к проблемам при сборе урожая зерновых и масличных культур. Известно, что растрескивание стручка у Arabidopsis находится под избыточным контролем генов SHP1 и SHP2; в случае двойной мутации shp1 shp2 спелый стручок остается закрытым (46). Возможно, гены SHP оказались мишенью при отборе зерновых на признак неосыпаемости зерна. Знания о функции этих генов могут быть использованы для повышения урожайности за счет уменьшения осыпания семян. К примеру, подавление ортолога SHP1 — BnSHP1 в растениях масличного рапса приводит к повышению устойчивости стручков к растрескиванию, тем самым увеличивая урожайность культуры (94).
В развитии плодов также участвует содержащий MADS-домен транскрипционный фактор FUL, который регулирует дифференцировку клеток плода в процессе развития и служит негативным регулятором экспрессии SHP1 и SHP2 (95). Сверхэкспрессия ортолога FUL горчицы — MADSB в растениях B. napus снижает растрескивание стручков (96). Интересно, что в случае сочного плода томата подавление активности двух ортологов FUL — FUL1 и FUL2 вызывает сильную задержку созревания плода, предположительно за счет снижения синтеза этилена и каротиноидов (97).
Еще один важный признак, учитываемый при селекции томата, — отсутствие суставной области на плодоножке, которая облегчает опадание плода (98). В спецификации зоны опадания участвует несколько MADS-box генов (98). Среди них наиболее подходящим для селекции растений считается J2; мутация j2 присутствует во многих линиях томата без суставной зоны (80, 98). Мутация в другом MADS-box гене — JOINTLESS1 (J1, или JOINTLESS), гомологичном гену времени цветения SVP, также приводит к бессуставному фенотипу (99). Однако его ценность для селекции сомнительна, поскольку соцветия j1 склонны к реверсии к вегетативному развитию после формирования нескольких цветков (80).
Показано, что ортологи SEP участвуют в созревании сочных плодов. Подавление активности гомологов SEP банана и яблони — соответственно MaMADS1/MaMADS2 и MdMADS8/MdMADS9 ингибирует созревание климактерических плодов и увеличивает срок их хранения (100, 101). Ортологи SEP участвуют и в развитии неклимактерических (нечувствительных к этилену) плодов, таких как клубника (102). Это указывает на ортологи SEP как на универсальную мишень при оптимизации созревания плодов.
Ключевым регулятором созревания сочного плода томата, а также важным геном, вовлеченным в доместикацию, считается MADS-box ген LeMADS-RIN (подсемейство SEP), мутация в котором приводит к несозреванию плода (103). Для зеленых и твердых плодов rinхарактерны отсутствие увеличения синтеза этилена и накопления пигментов и ароматических соединений (103). В гетерозиготном состоянии мутация rin широко используется в селекции сортов томата, поскольку она продлевает срок хранения плодов (104). Однако при этом нарушается питательная и вкусовая ценность плодов (вследствие низкого содержания ликопина и других соединений) (103). С помощью подхода CRISPR/Cas9 был создан ряд линий томата с различными SNP (single nucleotide polymorphisms) и короткими инделями в кодирующей последовательности гена RIN, приводящими к разной степени выраженности фенотипа несозревания плода (105). Эти линии рассматриваются в качестве кандидатов для использования в селекции сортов с увеличенным сроком хранения плодов (105).
Стоит упомянуть и MADS-box гены, участвующие в инициации путей биосинтеза метаболитов сочного плода. Сочные плоды растений (например, томат и перец) содержат два важных типа вторичных метаболитов — антоцианов и каротиноидов, которые не только окрашивают плод, но и выступают в качестве антиоксидантов (106).
Ген LeMADS-RIN относится к ключевым факторам регуляции биосинтеза каротиноидов в плодах томата (107). Мишенью продукта LeMADS-RIN при этом служат гены ключевых ферментов биосинтеза каротиноидов — фитоинсинтазы 1 (PSY1) и фитоиндесатуразы (PDS), тогда как гомологи AG — TOMATO AGAMOUS (TAG1) и TAG-LIKE1 (TAGL1) регулируют биосинтез каротиноидов, взаимодействуя с промоторами генов ликопин-β-циклазы (CYC-β) и каротиноид-изомеразы (CRTISO) (108, 109). В индукции экспрессии генов PSY1 и PDSи подавлении транскрипции генов ликопинциклаз (CYCB, LCYB и LCYE) также участвует MADS-box ген SlCMB1 (подсемейство SEP) (110).
Обнаружено немало свидетельств влияния MADS-box генов на биосинтез антоцианов в сочных плодах. Так, экспрессия MrMADS01 (подсемейство SEP) в ягодах восковницы красной (Myrica rubra) значительно увеличивается на последней стадии созревания, что позволило авторам предположить участие этого гена в биосинтезе антоцианов (111). Замалчивание гена PaMADS7 у черешни (Prunus avium) ингибировало созревание плодов и влияло, в числе прочего, на содержание антоцианов (112). В плодах красной груши (Pyrus) гены PbrMADS11 и PbrMADS12 участвуют в активации экспрессии структурных генов антоцианового пути, а также в регуляции реакции синтеза антоцианов в ответ на световые и температДоказательства того, что MADS-box гены оказались мишенью отбора во время доместикации, также были получены в исследованиях, выполненных на кукурузе (114). Например, ген ZEA AGAMOUS-LIKE1 (ZAGL1), который является гомологом гена времени цветения SOC1, на этапе одомашнивания, по-видимому, не только влиял на сроки цветения, но также способствовал росту числа рядов зерен в початках кукурузы, тем самым увеличивая размер плодов и урожайность (114, 115).
О т в е т р а с т е н и й н а с т р е с с ы. К признакам доместикации растений относятся механизмы устойчивости и адаптации к неблагоприятным факторам окружающей среды. На участие MADS-box генов в регуляции устойчивости растений к различным стрессам, таким как обезвоживание, засоление, низкие и высокие температуры, а также оксислительный и биотический стрессы, исследователи обратили внимание относительно недавно (39).
Так, у риса ген OsMADS26 (ортолог AGL12) известен как регулятор реакций, связанных с ответом на засуху и болезни, вызванные патогенами (116). Другой ген — OsMADS57 (клада AGL17) функционирует как стимулятор устойчивости к холодовому стрессу; кроме холода, экспрессия гена индуцируется воздействием засоления, засухи и абсцизовой кислоты (117).
MADS-box ген SVP Arabidopsis вызывает модификации некоторых процессов развития и функции газообмена в ответ на обезвоживание: растения с мутацией svp демонстрируют повышенную потерю влаги и поддерживают значительную скорость фотосинтетической ассимиляции CO2 в течение всего засушливого периода (118).
У томата солевой стресс, обезвоживание и поранение индуцируют экспрессию гена SlMBP11 (ортолог AGL15) (119). В то же время близкий гомолог SIMBP11 — ген SIMBP8 оказывает противоположный эффект на устойчивость растений томата к засолению (120). Ген TOMATO APETALA3 (TAP3) индуцируется в условиях холодового стресса (121), в то время как экспрессия генов TAP3,TOMATOMADSBOXGENE6 (TM6) и LePISTILLATA (LePI) подавляется в пыльниках в условиях повышенной температуры (122).
В ответ на холод, засуху и солевой стресс в растениях перца (Capsicum annuum) индуцируется экспрессия гена CaMADS (клада SEP1) (123), а у Ginkgo biloba — GbMADS9 (клада B-sister) (124). Различными стрессами (включая осмотический стресс) и фитогормонами индуцируется транскрипция AGAMOUS LIKE21 (AGL21) у Arabidopsis, что предполагает участие гена в регуляции пластичности корневой системы (ее способности изменять строение под влиянием факторов окружающей среды) и прорастания семян (125).
Еще один важный стресс для растений — обеднение почв минеральными элементами, такими как фосфор. Показано, что у пшеницы (T. aestivum) при P-депривации дифференциально регулируются девять MADS-box генов (126). Функциональный анализ одного из них — TaMADS51 показал, что его сверхэкспрессия в условиях дефицита фосфора улучшает рост растений, а также увеличивает биомассу, накопление фосфора и повышает антиоксидантную ферментативную активность (126). Еще одним примером служит ген ARABIDOPSIS NITRATE REGULATED 1 (ANR1, или AGL44) — хорошо известный позитивный регулятор развития корневой системы в ответ на доступность нитратов (127).
Плейотропия и избыточность MADS-box генов. Приведенные выше примеры показывают, что MADS-box гены, функции которых плейотропны и часто избыточны, были вовлечены в процессы доместикации растений. Важно отметить, что во многих случаях генные сети не были нарушены полностью — были введены более тонкие вариации, которые позволили точно настроить фенотип (128). Примером служат Brassica и томат, где вариации в FLC- и SEP-подобных генах приводят к модуляции соответственно времени цветения и архитектуры соцветий (62, 80). Во многих случаях определенные признаки избыточно регулируются несколькими паралогами MADS-box генов, что подчеркивает их потенциал для тонкой настройки фенотипа. Например, в геноме B. rapa присутствуют несколько FLC-паралогов (58-62), и комбинирование различных аллельных вариантов этих генов может позволить адаптировать время цветения к широкому спектру климатических условий. Точно так же комбинирование разных аллелей SEP-подобных генов дает возможность настроить структуру соцветия томата (80). Однако плейотропные эффекты многих MADS-box генов могут и создавать проблемы: в то время как мутации в SEP-генах J2 и EJ2 томата по отдельности оказывают благотворное влияние, двойная мутация из-за избыточности функции этих генов оказывается вредной: хотя растения j2 ej2 демонстрируют усиленное ветвление соцветий, одновременно увеличивается число пустоцветов (80). Однако во многих случаях этот и аналогичные эффекты можно смягчить посредством тщательного подбора комбинаций аллелей, которые влияют на один, но не на другой признак (80). Кроме того, следует учитывать дозозависимое действие многих аллелей MADS-box генов (129), а также паралогов MADS-box генов, например генов SEP у Arabidopsis (130), и это добавляет возможностей для точной настройки фенотипического результата.
Согласно модели «квартета», факторы транскрипции семейства MADS осуществляют свои функции в составе тетрамеров и могут иметь множество перекрывающихся ДНК-мишеней, часть из которых регулируется противоположным образом с помощью белковых комплексов разного состава (35, 38, 131). Поэтому при отсутствии функциональной избыточности введение мутаций в кодирующую последовательность MADS-box генов способно изменить конкретные взаимодействия белок-белок или белок-ДНК и, как следствие, разделить плейотропные функции одного гена (131).
В заключение отметим, что высокий функциональный консерватизм MADS-box генов и детальная характеристика их гомологов у модельных и культурных растений делает эти гены идеальными кандидатами для предсказуемого манипулирования фенотипами (80). Это может, в частности, достигаться посредством изменения цис-регуляторных элементов MADS-box генов и, как следствие, уровня их пространственно-временной экспрессии (во время конкретной фазы развития, в конкретной ткани), в том числе в ответ на различные сигналы (128). Модификации в кодирующей области также могут использоваться для тонкой настройки фенотипа, так как функция белков семейства MADS в значительной степени определяется взаимодействиями белок-белок и белок-ДНК, и, меняя партнеров и мишени, один и тот же белок может участвовать в разных путях развития (определение идентичности органов и других параметров роста и онтогенеза растения; ответ на стрессы; формирование различных хозяйственно ценных признаков) (131).
Таким образом, у растений MADS-box гены считаются одними из ключевых мишеней, которые были вовлечены в формирование признаков доместикации, повлиявших на продуктивность, адаптивность и воспроизводство — свойства, которые остаются хозяйственно значимыми и у современных возделываемых культур. Вариабельность гомологов MADS-box генов FLC, SOC1, SVP и VRN определяет различия во времени инициации цветения, в том числе в ответ на воздействие низких температур. Изменения в процессе физиологического покоя почек связаны с гомологами SVP, AGL24 и FUL. Морфологическая диверсификация соцветия и цветка ассоциированы с гомологами AP1/CAL, SEP, AP3, PI, AG и AGL17, тогда как стерильность и число плодов и семян — с AG, SEP, FUL и SVP. Гомологи MADS-box генов SVP, SEP, AP3, AGL12, AGL15, AGL17, AGL21 и AGL44 связывают с различиями в ответе растений на стрессы. Учитывая объем накопленных качественных и количественных данных, предсказание специфических фенотипических последствий изменений у MADS-box генов гораздо реальнее, нежели у подавляющего большинства генов других семейств. Продолжение фундаментальных и прикладных исследований MADS-box генов у самых разных видов не только приведет к более глубокому пониманию развития и эволюции растений, но также внесет большой вклад в улучшение сельскохозяйственных культур.
Число MADS-box генов в геномах различных видов растений, включая сельскохозяйственные культуры |
|||
Таксономическая группа |
Вид |
Число генов |
Ссылка |
Мхи |
Physcomitrella patens |
23 |
(43) |
Ликофиты |
Selaginella moellendorffii |
40 |
(132) |
Голосеменные |
Picea abies |
278 |
(33) |
Pinus taeda |
367 |
(133) |
|
Gnetum gnemon |
41 |
(133) |
|
Покрытосеменные однодольные |
Модельный вид для зерновых культур Brachypodium distachyon |
75 |
(33) |
Рис Oryza sativa |
75 |
(33) |
|
Пшеница Triticum aestivum |
180 |
(134) |
|
Покрытосеменные двудольные |
Модельный вид Arabidopsis thaliana |
107 |
(40) |
Базальная группа цветковых Amborella trichopoda |
33 |
(132) |
|
Капустные Brassica rapa |
160 |
(135) |
|
Соя Glycine max |
106 |
(136) |
|
Яблоня Malus domestica |
146 |
(137) |
|
Арбуз обыкновенный Citrullus lanatus |
39 |
(138) |
|
Латук посевной Lactuca sativa |
82 |
(139) |
|
Виноград Vitis vinifera |
90 |
(140) |
|
Картофель Solanum tuberosum |
167 |
(33) |
|
Томат Solanum lycopersicum |
131 |
(33) |
|
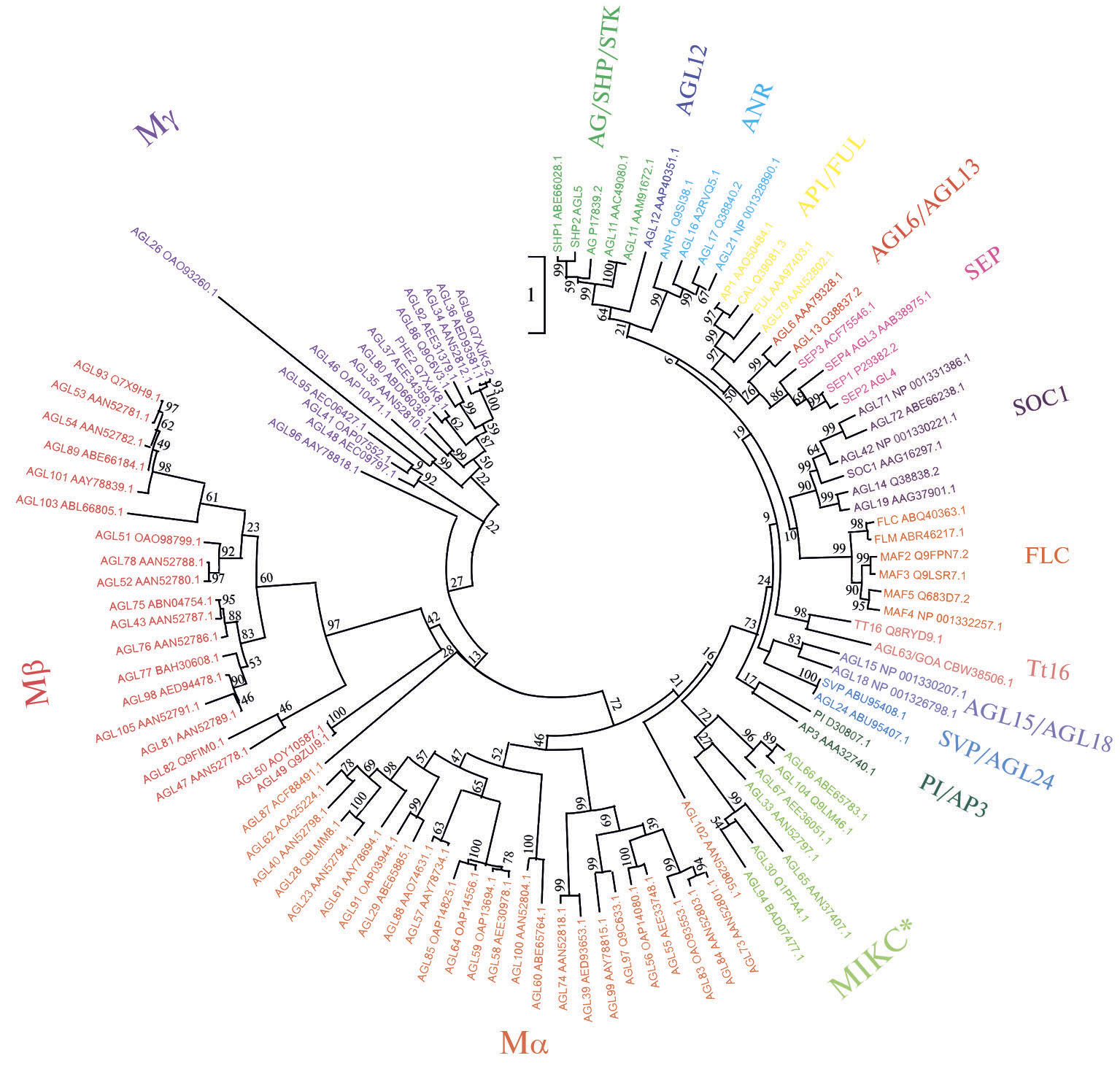
Филогения факторов регуляции транскрипции с доменом MADS у Arabidopsis thaliana. Подсемейства выделены разным цветом. Подсемейства Ma, Mb и Mγ включают MADS-белки типа I, остальные подсемейства — MADS-белки MIKC-типа II. Рядом с названием каждого белка указан его идентификационный номер в базе данных NCBI (https://www.ncbi.nlm.nih.gov/). Значения бутстрепа указаны в основании ветвей. Дендрограмма построена с помощью программы MEGA 7.0 (https://www.megasoftware.net/) с использованием метода максимального правдоподобия.
ЛИТЕРАТУРА
- Purugganan M.D., Fuller D.Q. The nature of selection during plant domestication. Nature, 2009, 457(7231): 843-848 (doi: 10.1038/nature07895).
- Meyer R.S., DuVal A.E., Jensen H.R. Patterns and processes in crop domestication: an historical review and quantitative analysis of 203 global food crops. New Phytologist, 2012, 196(1): 29-48 (doi: 10.1111/j.1469-8137.2012.04253.x).
- Fuller D.Q., Denham T., Arroyo-Kalin M., Lucas L., Stevens C.J., Qin L., Allaby R.G., Purugganan M.D. Convergent evolution and parallelism in plant domestication revealed by an expanding archaeological record. Proceedings of the National. Academy of Sciences of the USA, 2014, 111(17): 6147-6152 (doi: 10.1073/pnas.1308937110).
- Larson G., Piperno D.R., Allaby R.G., Purugganan M.D., Andersson L., Arroyo-Kalin M., Barton L., Vigueira C.C., Denham T., Dobney K., Doust A.N., Gepts P., Gilbert M.T.P., Gremillion K.J., Lucas L., Lukens L., Marshall F.B., Olsen K.M., Pires J.C., Richerson P.J., de Casas R.R., Sanjur O.I., Thomas M.G., Fuller D.Q. Current perspectives and the future of domestication studies. Proceedings of the National Academy of Sciences of the USA, 2014, 111(17): 6139-6146 (doi: 10.1073/pnas.1323964111 ).
- Martínez-Ainsworth N.E., Tenaillon M.I. Superheroes and masterminds of plant domestication. Comptes Rendus Biologies, 2016, 339(7-8): 268-273 (doi: 10.1016/j.crvi.2016.05.005).
- Stetter M.G., Gates D.J., Mei W., Ross-Ibarra J. How to make a domesticate. Current Biology, 2017, 27(17): R896-R900 (doi: 10.1016/j.cub.2017.06.048).
- Milla R., Bastida J.M., Turcotte M.M., Jones G., Violle C., Osborne C.P., Chacon-Labella J., Sosinski E.E., Kattge J., Laughlin D.C., Forey E., Minden V., Cornelissen J.H.C., Amiaud B., Kramer K., Boenisch G., He T., Pillar V.D., Byun C. Phylogenetic patterns and phenotypic profiles of the species of plants and mammals farmed for food. Nature Ecology and Evolution, 2018, 2(11): 1808-1817 (doi: 10.1038/s41559-018-0690-4).
- Manning K., Pelling R., Higham T., Schwenniger J.-L., Fuller D.Q. 4500-year old domesticated pearl millet (Pennisetum glaucum) from the Tilemsi Valley, Mali: new insights into an alternative cereal domestication pathway. Journal of Archaeological Science, 2011, 38(2): 312-322 (doi: 10.1016/j.jas.2010.09.007).
- Meyer R., Purugganan M.D. Evolution of crop species: genetics of domestication and diversification. Nature Reviews Genetics, 2013, 14(12): 840-852 (doi: 10.1038/nrg3605).
- Purugganan M.D. Evolutionary insights into the nature of plant domestication. Current Biology, 2019, 29(14): R705-R714 (doi: 10.1016/j.cub.2019.05.053).
- Purugganan M.D., Fuller D.Q. Archaeological data reveal slow rates of evolution during plant domestication. Evolution, 2011, 65(1): 171-183 (doi: 10.1111/j.1558-5646.2010.01093.x).
- Wang L., Beissinger T.M., Lorant A., Ross-Ibarra C., Ross-Ibarra J., Hufford M.B. The interplay of demography and selection during maize domestication and expansion. Genome Biology, 2017, 18(1): 215 (doi: 10.1186/s13059-017-1346-4).
- Ramos-Madrigal J., Smith B.D., Moreno-Mayar J.V., Gopalakrishnan S., Ross-Ibarra J., Gilbert M.T.P., Wales N. Genome sequence of a 5,310-year-old maize cob provides insights into the early stages of maize domestication. Current Biology, 2016, 26(23): 3195-3201 (doi: 10.1016/j.cub.2016.09.036).
- Smith O., Nicholson W., Kistler L., Mace E., Clapham A., Rose P., Stevens C., Ware R., Samavedam S., Barker G., Jordan D., Fuller D.Q., Allaby R.G. A domestication history of dynamic adaptation and genomic deterioration in Sorghum. Nature Plants, 2019, 5(4): 369-379 (doi: 10.1038/s41477-019-0397-9).
- Luo M., Yang Z.-L., You F.M., Kawahara T., Waines J.G., Dvorak J. The structure of wild and domesticated emmer wheat populations, gene flow between them, and the site of emmer domestication. Theoretical and Applied Genetics, 2007, 114(6): 947-959 (doi: 10.1007/s00122-006-0474-0).
- Allaby R.G. Integrating the processes in the evolutionary system of domestication. Journal of Experimental Botany, 2010, 61(4): 935-944 (doi: 10.1093/jxb/erp382).
- Zohary D. Unconscious selection and the evolution of domesticated plants. Economic Botany, 2004, 58: 5-10 (doi: 10.1663/0013-0001(2004)058[0005:USATEO]2.0.CO;2).
- Arnold M.L. Natural hybridization and the evolution of domesticated, pest and disease organisms. Molecular Ecology, 2014, 13(5): 97-1007 (doi: 10.1111/j.1365-294X.2004.02145.x).
- Janzen G.M., Wang L., Hufford M.B. The extent of adaptive wild introgression in crops. New Phytologist, 2018, 221(3): 1279-1288 (doi: 10.1111/nph.15457).
- Heslop-Harrison J.S., Schwarzacher T. Domestication, genomics, and the future for banana. Annals of Botany, 2007, 100(5): 1073-1084 (doi: 10.1093/aob/mcm191).
- Marcussen T., Sandve S.R., Heier L., Spannagl M., Pfeifer M., Internation Wheat Genome Sequencing Consortium, Jakobsen K.S., Wulff B.B., Steuernagel B., Mayer K.F., Olsen O.A. Ancient hybridizations among the ancestral genomes of bread wheat. Science, 2014, 345(6194): 1250092 (doi: 10.1126/science.1250092).
- Choi J.Y., Purugganan M.D. Multiple origin but single domestication led to Oryza sativa. G3 (Bethesda), 2018, 8(3): 797-803 (doi: 10.1534/g3.117.300334).
- Heerwaarden J., Doebley J., Briggs W.H., Glaubitz J.C., Goodman M.M., Sanchez Gonzalez J., Ross-Ibarra J. Genetic signals of origin, spread, and introgression in a large sample of maize landraces. Proceedings of the National. Academy of Sciences of the USA, 2011, 108(3): 1088-1092 (doi: 10.1073/pnas.1013011108).
- Mascher M., Schuenemann V.J., Davidovich U., Marom N., Himmelbach A., Hubner S., Korol A., David M., Reiter E., Reihl S., Schreiber M., Vohr S.H., Green R.E., Dawson I.K., Russell J., Kilian B., Muehlbauer G.J., Waugh R., Fahima T., Krause J., Weiss E., Stein N. Genomic analysis of 6,000-year-old cultivated grain illuminates the domestication history of barley. Nature Genetics, 2016, 48(9): 1089-1093 (doi: 10.1038/ng.3611).
- Cornille A., Giraud T., Smulders M.J., Roldán-Ruiz I., Gladieux P. The domestication and evolutionary ecology of apples. Trends in Genetics, 2014, 30(2): 57-65 (doi: 10.1016/j.tig.2013.10.002).
- Miller A.J., Gross B.L. From forest to field: perennial fruit crop domestication. American Journal of Botany, 2011, 98(9): 1389-1414 (doi: 10.3732/ajb.1000522).
- Gaut B.S., Seymour D.K., Liu Q., Zhou Y. Demography and its effects on genomic variation in crop domestication. Nature Plants, 2018, 4(8): 512-520 (doi: 10.1038/s41477-018-0210-1).
- Lemmon Z.H., Reem N.T., Dalrymple J., Soyk S., Swartwood K.E., Rodriguez-Leal D., Van Eck J., Lippman Z.B. Rapid improvement of domestication traits in an orphan crop by genome editing. Nature Plants, 2018, 4(10): 766-770 (doi: 10.1038/s41477-018-0259-x).
- Zsögön A., Cermak T., Naves E.R., Notini M.M., Edel K.H., Weinl S., Freschi L., Voytas V.F., Kudla J., Peres L.E.P. De novo domestication of wild tomato using genome editing. Nature Biotechnology, 2018, 36: 1211-1216 (doi: 10.1038/nbt.4272).
- Li T., Yang X., Yu Y., Si X., Zhai X., Zhang H., Dong W., Gao C., Xu C. Domestication of wild tomato is accelerated by genome editing. Nature Biotechnology, 2018, 36: 1160-1163 (doi: 10.1038/nbt.4273).
- Lenser T., Theißen G. Molecular mechanisms involved in convergent crop domestication. Trends in Plant Science, 2013, 18(12): 704-714 (doi: 10.1016/j.tplants.2013.08.007).
- Kantar M.B., Nashoba A.R., Anderson J.E., Blackman B.K., Rieseberg L.H. The genetics and genomics of plant domestication. BioScience, 2017, 67(11): 971-982 (doi: 10.1093/biosci/bix114).
- Schilling S., Pan S., Kennedy A., Melzer R. MADS-box genes and crop domestication: the jack of all traits. Journal of Experimental Botany, 2018, 69(7): 1447-1469 (doi: 10.1093/jxb/erx479).
- Theißen G., Rümpler F., Gramzow L. Array of MADS-box genes: facilitator for rapid adaptation? Trends in Plant Sciences, 2018, 23(7): 563-576 (doi: 10.1016/j.tplants.2018.04.008).
- Theißen G., Melzer R., Rümpler F. MADS-domain transcription factors and the floral quartet model of flower development: linking plant development and evolution. Development, 2016, 143(18): 3259-3271 (doi: 10.1242/dev.134080).
- Jiao Y., Paterson A.H. Polyploidy-associated genome modifications during land plant evolution. Philosophical Transactions of the Royal Society B: Biological Sciences, 2014, 369(1648): 20130355 (doi: 10.1098/rstb.2013.0355).
- Kim S., Park J., Yeom S.I., Kim Y.M., Seo E., Kim K.T., Kim M.S., Lee J.M., Cheong K., Shin H.S., Kim S.B., Han K., Lee J., Park M., Lee H.A., Lee H.Y., Lee Y., Oh S., Lee J.H., Choi E., Choi E., Lee S.E., Jeon J., Kim H., Choi G., Song H., Lee J., Lee S.C., Kwon J.K., Lee H.Y., Koo N., Hong Y., Kim R.W., Kang W.H., Huh J.H., Kang B.C., Yang T.J., Lee Y.H., Bennetzen J.L., Choi D. New reference genome sequences of hot pepper reveal the massive evolution of plant disease-resistance genes by retroduplication. Genome Biology, 2017, 18(1): 210 (doi: 10.1186/s13059-017-1341-9).
- Smaczniak C., Immink R.G, Angenent G.C., Kaufmann K. Developmental and evolutionary diversity of plant MADS-domain factors: insights from recent studies. Development, 2012, 139(17): 3081-3098 (doi: 10.1242/dev.074674).
- Castelán-Muñoz N., Herrera J., Cajero-Sánchez W., Arrizubieta M., Trejo C., García-Ponce B., Sánchez M.P., Álvarez-Buylla E.R., Garay-Arroyo A. MADS-box genes are key components of genetic regulatory networks involved in abiotic stress and plastic developmental responses in plants. Frontiers in Plant Science, 2019, 10: 853 (doi: 10.3389/fpls.2019.00853).
- Parenicovа L., de Folter S., Kieffer M., Horner D.S., Favalli C., Busscher J., Cook H.E., Ingram R.M., Kater M.M., Davies B., Angenent G.C., Colombo L. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis: new openings to the MADS world. The Plant Cell, 2003, 15(7): 1538-1551 (doi: 10.1105/tpc.011544).
- Bowman J.L., Smyth D.R., Meyerowitz E.M. Genes directing flower development in Arabidopsis. The Plant Cell, 1989, 1(1): 37-52 (doi: 10.1105/tpc.1.1.37).
- Sommer H., Beltrán J.P., Huijser P., Pape H., Lönnig W.E., Saedler H., Schwarz-Sommer Z. Deficiens, a homeotic gene involved in the control of flower morphogenesis in Antirrhinum majus: the protein shows homology to transcription factors. The EMBO Journal, 1990, 9(3): 605-61.
- Gramzow L., Theißen G. Phylogenomics reveals surprising sets of essential and dispensable clades of MIKCc-group MADS-box genes in flowering plants. Journal of Experimental Zoology. Part B, Molecular and Developmental Evolution, 2015, 324(4): 353-362 (doi: 10.1002/jez.b.22598).
- Lee J., Lee I. Regulation and function of SOC1, a flowering pathway integrator. Journal of Experimental Botany, 2010, 61(9): 2247-2254 (doi: 10.1093/jxb/erq098).
- Airoldi C.A., Davies B. Gene duplication and the evolution of plant MADS-box transcription factors. Journal of Genetics and Genomics, 2012, 39(4): 157-165 (doi: 10.1016/j.jgg.2012.02.008).
- Pinyopich A., Ditta G.S., Savidge B., Liljegren S.J., Baumann E., Wisman E., Yanofsky M.F. Assessing the redundancy of MADS-box genes during carpel and ovule development. Nature, 2003, 424(6944): 85-88 (doi: 10.1038/nature01741).
- Kempin S.A., Savidge B., Yanofsky M.F. Molecular basis of the cauliflower phenotype in Arabidopsis. Science, 1995, 267(5197): 522-525 (doi: 10.1126/science.7824951).
- Mandel M.A., Gustafson-Brown C., Savidge B., Yanofsky M.F. Molecular characterization of the Arabidopsis floral homeotic gene APETALA1. Nature, 1992, 360(6401): 273-277 (doi: 10.1038/360273a0).
- Alvarez-Buylla E.R., García-Ponce B., Garay-Arroyo A. Unique and redundant functional domains of APETALA1 and CAULIFLOWER, two recently duplicated Arabidopsis thaliana floral MADS-box genes. Journal of Experimental Botany, 2006, 57(12): 3099-3107 (doi: 10.1093/jxb/erl081).
- Cho L.H., Yoon J., An G. The control of flowering time by environmental factors. The Plant Journal, 2017, 90(4): 708-719 (doi: 10.1111/tpj.13461).
- Luo X., Chen T., Zeng X., He D., He Y. Feedback regulation of FLC by FLOWERING LOCUS T (FT) and FD through a 5' FLC promoter region in Arabidopsis. Molecular Plant, 2019, 12(3): 285-288 (doi: 10.1016/j.molp.2019.01.013).
- Preston J., Sandve S. Adaptation to seasonality and the winter freeze. Frontiers in Plant Science, 2013, 4: 167 (doi: 10.3389/fpls.2013.00167).
- Andrés F., Coupland G. The genetic basis of flowering responses to seasonal cues. Nature Reviews Genetics, 2012, 13(9): 627-639 (doi: 10.1038/nrg3291).
- Sharma N., Geuten K., Giri B.S., Varma A. The molecular mechanism of vernalization in Arabidopsis and cereals: role of Flowering Locus C and its homologs. Physiologia Plantarum, 2020, 170(3): 373-383 (doi: 10.1111/ppl.13163).
- Amasino R. Vernalization, competence, and the epigenetic memory of winter. The Plant Cell, 2004, 16(10): 2553-2559 (doi: 10.1105/tpc.104.161070).
- Hou J., Long Y., Raman H., Zou X., Wang J., Dai S., Xiao Q., Li C., Fan L., Liu B., Meng J. A Tourist-like MITE insertion in the upstream region of the BnFLC.A10 gene is associated with vernalization requirement in rapeseed (Brassica napus L.). BMC Plant Biology, 2012, 12: 238 (doi: 10.1186/1471-2229-12-238).
- Calderwood A., Lloyd A., Hepworth J., Tudor E.H., Jones D.M., Woodhouse S., Bilham L., Chinoy C., Williams K., Corke F., Doonan J.H., Ostergaard L., Irwin J.A., Wells R., Morris R.J. Total FLC transcript dynamics from divergent paralogue expression explains flowering diversity in Brassica napus. New Phytologist, 2020, 229(6): 3534-3548 (doi: 10.1111/nph.17131).
- Kakizaki T., Kato T., Fukino N., Ishida M., Hatakeyama K., Matsumoto S. Identification of quantitative trait loci controlling late bolting in Chinese cabbage (Brassica rapa L.) parental line Nou 6 gou. Breeding Science, 2011, 61: 151-159 (doi: 10.1270/jsbbs.61.151).
- Yuan Y.X., Wu J., Sun R.F., Zhang X.W., Xu D.H., Bonnema G., Wang X.W. A naturally occurring splicing site mutation in the Brassica rapa FLC1 gene is associated with variation in flowering time. Journal of Experimental Botany, 2009, 60(4): 1299-1308 (doi: 10.1093/jxb/erp010).
- Xiao D., Zhao J.J., Hou X.L., Basnet R.K., Carpio D.P., Zhang N.W., Bucher J., Lin K., Cheng F., Wang X.W., Bonnema G. The Brassica rapa FLC homologue FLC2 is a key regulator of flowering time, identified through transcriptional co-expression networks. Journal of Experimental Botany, 2013, 64(14): 4503-4516 (doi: 10.1093/jxb/ert264).
- Wu J., Wei K., Cheng F., Li S., Wang Q., Zhao J., Bonnema G., Wang X. A naturally occurring InDel variation in BraA.FLC.b (BrFLC2) associated with flowering time variation in Brassica rapa. BMC Plant Biology, 2012, 12: 151 (doi: 10.1186/1471-2229-12-151).
- Irwin J.A., Soumpourou E., Lister C., Ligthart J.D., Kennedy S., Dean C. Nucleotide polymorphism affecting FLC expression underpins heading date variation in horticultural brassicas. The Plant Journal, 2016, 87(6): 597-605 (doi: 10.1111/tpj.13221).
- Kennedy A., Geuten K. The role of FLOWERING LOCUS C relatives in cereals. Frontiers in Plant Science, 2020, 11: 617340 (doi: 10.3389/fpls.2020.617340).
- Sharma N., Ruelens P., D’hauw M., Maggen T., Dochy N., Torfs S., Kaufmann K., Rohde A., Geuten K. A flowering locus C homolog is a vernalization-regulated repressor in Brachypodium and is cold regulated in wheat. Plant Physiology, 2017, 173(2): 1301-1315 (doi: 10.1104/pp.16.01161).
- Bloomer R.H., Dean C. Fine-tuning timing: natural variation informs the mechanistic basis of the switch to flowering in Arabidopsis thaliana. Journal of Experimental Botany, 2017, 68(20): 5439-5452 (doi: 10.1093/jxb/erx270).
- Ratcliffe O.J., Kumimoto R.W., Wong B.J., Riechmann J.L. Analysis of the Arabidopsis MADS AFFECTING FLOWERING gene family: MAF2 prevents vernalization by short periods of cold. The Plant Cell, 2003, 15(5): 1159-1169 (doi: 10.1105/tpc.009506).
- Verhage L., Severing E.I., Bucher J., Lammers M., Busscher-Lange J., Bonnema G., Rodenburg N., Proveniers M.C., Angenent G.C., Immink R.G. Splicing-related genes are alternatively spliced upon changes in ambient temperatures in plants. PLoS ONE, 2017, 12(3): e0172950 (doi: 10.1371/journal.pone.0172950).
- Airoldi C.A., McKay M., Davies B. MAF2 is regulated by temperature-dependent splicing and represses flowering at low temperatures in parallel with FLM. PLoS ONE, 2015, 10(5): e0126516 (doi: 10.1371/journal.pone.0126516).
- Rosloski S.M., Jali S.S., Balasubramanian S., Weigel D., Grbic V. Natural diversity in flowering responses of Arabidopsis thaliana caused by variation in a tandem gene array. Genetics, 2010, 186(1): 263-276 (doi: 10.1534/genetics.110.116392).
- Dondup D., Dong G., Xu D., Zhang L., Zha S., Yuan X., Tashi N., Zhang J., Guo G. Allelic variation and geographic distribution of vernalization genes HvVRN1 and HvVRN2 in Chinese barley germplasm. Molecular Breeding, 2016, 36: 11 (doi: 10.1007/s11032-016-0434-6).
- Asp T., Byrne S., Gundlach H., Bruggmann R., Mayer K.F., Andersen J.R., Xu M., Greve M., Lenk I., Lübberstedt T. Comparative sequence analysis of VRN1 alleles of Lolium perenne with the co-linear regions in barley, wheat, and rice. Molecular Genetics and Genomics, 2011, 286(5-6): 433-447 (doi: 10.1007/s00438-011-0654-8).
- Fu D., Szucs P., Yan L., Helguera M., Skinner J.S., von Zitzewitz J., Hayes P.M., Dubcovsky J. Large deletions within the first intron in VRN-1 are associated with spring growth habit in barley and wheat. Molecular Genetics and Genomics, 2005, 273(1): 54-65 (doi: 10.1007/s00438-004-1095-4).
- Kippes N., Debernardi J.M., Vasquez-Gross H.A., Akpinar B.A., Budak H., Kato K., Chao S., Akhunov E., Dubcovsky J. Identification of the VERNALIZATION 4 gene reveals the origin of spring growth habit in ancient wheats from South Asia. Proceedings of the National Academy of Sciences of the USA, 2015, 112(39): E5401-E5410 (doi: 10.1073/pnas.1514883112).
- Bielenberg D.G., Wang Y., Li Z., Zhebentyayeva T., Fan S., Reighard G.L., Scorza R., Abbott A.G. Sequencing and annotation of the evergrowing locus in peach [Prunus persica (L.) Batsch] reveals a cluster of six MADS-box transcription factors as candidate genes for regulation of terminal bud formation. Tree Genetics and Genomes, 2008, 4: 495-507 (doi: 10.1007/s11295-007-0126-9).
- Falavigna V.D.S., Guitton B., Costes E., Andrés F. I want to (bud) break free: the potential role of DAM and SVP-like genes in regulating dormancy cycle in temperate fruit trees. Frontiers in Plant Sciences, 2019, 9: 1990 (doi: 10.3389/fpls.2018.01990).
- Hoenicka H., Nowitzki O., Hanelt D., Fladung M. Heterologous overexpression of the birch FRUITFULL-like MADS-box gene BpMADS4 prevents normal senescence and winter dormancy in Populus tremula L. Planta, 2008, 227(5): 1001-1011 (doi: 10.1007/s00425-007-0674-0).
- Carr S.M., Irish V.F. Floral homeotic gene expression defines developmental arrest stages in Brassica oleracea L. vars. botrytis and italica. Planta, 1997, 201(2): 179-188 (doi: 10.1007/BF01007702).
- Duclos D.V., Björkman T. Meristem identity gene expression during curd proliferation and flower initiation in Brassica oleracea. Journal of Experimental Botany, 2008, 59(2): 421-433 (doi: 10.1093/jxb/erm327).
- Purugganan M.D., Boyles A.L., Suddith J.I. Variation and selection at the CAULIFLOWER floral homeotic gene accompanying the evolution of domesticated Brassica oleracea. Genetics, 2000, 155(2): 855-862 (doi: 10.1093/genetics/155.2.855).
- Soyk S., Lemmon Z.H., Oved M., Fisher J., Liberatore K.L., Park S.J., Goren A., Jiang K., Ramos A., van der Knaap E., Van Eck J., Zamir D., Eshed Y., Lippman Z.B. Bypassing negative epistasis on yield in tomato imposed by a domestication gene. Cell, 2017, 169(6): 1142-1155 (doi: 10.1016/j.cell.2017.04.032).
- Sreenivasulu N., Schnurbusch T. A genetic playground for enhancing grain number in cereals. Trends in Plant Science, 2012, 17(2): 91-101 (doi: 10.1016/j.tplants.2011.11.003).
- Guo S., Xu Y., Liu H., Mao Z., Zhang C., Ma Y., Zhang Q., Meng Z., Chong K. The interaction between OsMADS57 and OsTB1 modulates rice tillering via DWARF14. Nature Communications, 2013, 4: 1566 (doi: 10.1038/ncomms2542).
- Jeon J.S., Lee S., Jung K.H., Yang W.S., Yi G.H., Oh B.G., An G.H. Production of transgenic rice plants showing reduced heading date and plant height by ectopic expression of rice MADS-box genes. Molecular Breeding, 2000, 6: 581-592 (doi: 10.1023/A:1011388620872).
- Dubois A., Raymond O., Maene M., Baudino S., Langlade N.B., Boltz V., Vergne P., Bendahmane M. Tinkering with the C-function: a molecular frame for the selection of double flowers in cultivated roses. PLoS ONE, 2010, 5(2): e9288 (doi: 10.1371/journal.pone.0009288).
- Liu Z., Zhang D., Liu D., Li F., Lu H. Exon skipping of AGAMOUS homolog PrseAG in developing double flowers of Prunus lannesiana (Rosaceae). Plant Cell Reports, 2013, 32(2): 227-237 (doi: 10.1007/s00299-012-1357-2).
- Klocko A.L., Borejsza-Wysocka E., Brunner A.M., Shevchenko O., Aldwinckle H., Strauss S.H. Transgenic suppression of AGAMOUS genes in apple reduces fertility and increases floral attractiveness. PLoS ONE, 2016, 11(8): e0159421 (doi: 10.1371/journal.pone.0159421).
- Yao J.L., Dong Y.H., Morris B.A.M. Parthenocarpic apple fruit production conferred by transposon insertion mutations in a MADS-box transcription factor. Proceedings of the National Academy of Sciences of the USA, 2001, 98(3): 1306-1311 (doi: 10.1073/pnas.031502498).
- Lombardo F., Kuroki M., Yao S.G., Shimizu H., Ikegaya T., Kimizu M., Ohmori S., Akiyama T., Hayashi T., Yamaguchi T., Koike S., Yatou O., Yoshida H. The superwoman1-cleistogamy2 mutant is a novel resource for gene containment in rice. Plant Biotechnology Journal, 2017, 15(1): 97-106 (doi: 10.1111/pbi.12594).
- Masiero S., Colombo L., Grini P.E., Schnittger A., Kater M.M. The emerging importance of type I MADS box transcription factors for plant reproduction. The Plant Cell, 2011, 23(3): 865-872 (doi: 10.1105/tpc.110.081737).
- Mejía N., Soto B., Guerrero M., Casanueva X., Houel C., Miccono M., Ramos R., Le Cunff L., Boursiquot J.M., Hinrichsen P., Adam-Blondon A.F. Molecular, genetic and transcriptional evidence for a role of VvAGL11 in stenospermocarpic seedlessness in grapevine. BMC Plant Biology, 2011, 11: 57 (doi: 10.1186/1471-2229-11-57).
- Bergamini C., Cardone M.F., Anaclerio A., Perniola R., Pichierri A., Genghi R., Alba V., Forleo L.R., Caputo A.R., Montemurro C., Blanco A., Antonacci D. Validation assay of p3_VvAGL11 marker in a wide range of genetic background for early selection of stenospermocarpy in Vitis vinifera L. Molecular Biotechnology, 2013, 54(3): 1021-1030 (doi: 10.1007/s12033-013-9654-8).
- Ocarez N., Mejía N. Suppression of the D-class MADS-box AGL11 gene triggers seedlessness in fleshy fruits. Plant Cell Reports, 2016, 35(1): 239-254 (doi: 10.1007/s00299-015-1882-x).
- Angenent G.C., Franken J., Busscher M., van Dijken A., van Went J.L., Dons H.J., van Tunen A.J. A novel class of MADS box genes is involved in ovule development in petunia. The Plant Cell, 1995, 7(10): 1569-1582 (doi: 10.1105/tpc.7.10.1569).
- Kord H., Shakib A.M., Daneshvar M.H., Azadi P., Bayat V., Mashayekhi M., Zarea M., Seifi A., Ahmad-Raji M. RNAi-mediated down-regulation of SHATTERPROOF gene in transgenic oilseed rape. 3 Biotech., 2015, 5(3): 271-277 (doi: 10.1007/s13205-014-0226-9).
- Ferrándiz C., Liljegren S.J., Yanofsky M.F. Negative regulation of the SHATTERPROOF genes by FRUITFULL during Arabidopsis fruit development. Science, 2000, 289(5478): 436-438 (doi: 10.1126/science.289.5478.436).
- Chandler J., Corbesier L., Spielmann P., Dettendorfer J., Stahl D., Apel K., Melzer S. Modulating flowering time and prevention of pod shatter in oilseed rape. Molecular Breeding, 2005, 15: 87-94 (doi: 10.1007/s11032-004-2735-4).
- Wang S., Lu G., Hou Z., Luo Z., Wang T., Li H., Zhang J., Ye Z. Members of the tomato FRUITFULL MADS-box family regulate style abscission and fruit ripening. Journal of Experimental Botany, 2014, 65(12): 3005-3014 (doi: 10.1093/jxb/eru137).
- Liu D., Wang D., Qin Z., Zhang D., Yin L., Wu L., Colasanti J., Li A., Mao L. The SEPALLATA MADS-box protein SLMBP21 forms protein complexes with JOINTLESS and MACROCALYX as a transcription activator for development of the tomato flower abscission zone. The Plant Journal, 2014, 77(2): 284-296 (doi: 10.1111/tpj.12387).
- Hileman L.C., Sundstrom J.F., Litt A., Chen M., Shumba T., Irish V.F. Molecular and phylogenetic analyses of the MADS-box gene family in tomato. Molecular Biology and Evolution, 2006, 23(11): 2245-2258 (doi: 10.1093/molbev/msl095).
- Ireland H.S., Yao J.L., Tomes S., Sutherland P.W., Nieuwenhuizen N., Gunaseelan K., Winz R.A., David K.M., Schaffer R.J. Apple SEPALLATA1/2-like genes control fruit flesh development and ripening. The Plant Journal, 2013, 73(6): 1044-1056 (doi: 10.1111/tpj.12094).
- Elitzur T., Yakir E., Quansah L., Zhangjun F., Vrebalov J., Khayat E., Giovannoni J.J., Friedman H. Banana MaMADS transcription factors are necessary for fruit ripening and molecular tools to promote shelf-life and food security. Plant Physiology, 2016, 171(1): 380-391 (doi: 10.1104/pp.15.01866).
- Seymour G.B., Ryder C.D., Cevik V., Hammond J.P., Popovich A., King G.J., Vrebalov J., Giovannoni J.J., Manning K. A SEPALLATA gene is involved in the development and ripening of strawberry (Fragaria × ananassa Duch.) fruit, a non-climacteric tissue. Journal of Experimental Botany, 2011, 62(3): 1179-1188 (doi: 10.1093/jxb/erq360).
- Vrebalov J., Ruezinsky D., Padmanabhan V., White R., Medrano D., Drake R., Schuch W., Giovannoni J. A MADS-box gene necessary for fruit ripening at the tomato ripening-inhibitor (rin) locus. Science, 2002, 296(5566): 343-346 (doi: 10.1126/science.1068181).
- Bai Y., Lindhout P. Domestication and breeding of tomatoes: what have we gained and what can we gain in the future? Annals of Botany, 2007, 100(5): 1085-1094 (doi: 10.1093/aob/mcm150).
- Ito Y., Nishizawa-Yokoi A., Endo M., Mikami M., Toki S. CRISPR/Cas9-mediated mutgenesis of the RIN locus that regulates tomato fruit ripening. Biochemical and Biophysical Research Communications, 2015, 467(1): 76-82 (doi: 10.1016/j.bbrc.2015.09.117).
- Dhar M.K., Sharma R., Koul A., Kaul S. Development of fruit color in Solanaceae: a story of two biosynthetic pathways. Briefings in Functional Genomics, 2015, 14(3): 199-212 (doi: 10.1093/bfgp/elu018).
- Martel C., Vrebalov J., Tafelmeyer P., Giovannoni J.J. The tomato MADS-box transcription factor RIPENING INHIBITOR interacts with promoters involved in numerous ripening processes in a COLORLESS NONRIPENING-dependent manner. Plant Physiology, 2011, 157(3): 1568-1579 (doi: 10.1104/pp.111.181107).
- Pan I.L., McQuinn R., Giovannoni J.J., Irish V.F. Functional diversification of AGAMOUS lineage genes in regulating tomato flower and fruit development. Journal of Experimental Botany, 2010, 61(6): 1795-1806 (doi: 10.1093/jxb/erq046).
- Li S., Xu H., Ju Z., Cao D., Zhu H., Fu D., Grierson D., Qin G., Luo Y., Zhu B. The RIN-MC Fusion of MADS-box transcription factors has transcriptional activity and modulates expression of many ripening genes. Plant Physiologist, 2018, 176(1): 891-909 (doi: 10.1104/pp.17.01449).
- Zhang J., Hu Z., Yao Q., Guo X., Nguyen V., Li F., Chen G. A tomato MADS-box protein, SlCMB1, regulates ethylene biosynthesis and carotenoid accumulation during fruit ripening. Scientific Reports, 2018, 8(1): 3413 (doi: 10.1038/s41598-018-21672-8).
- Zhao H.B., Jia H.M., Wang Y., Wang G.Y., Zhou C.C., Jia H.J., Gao Z.S.Dr. Genome-wide identification and analysis of the MADS-box gene family and its potential role in fruit development and ripening in red bayberry (Morella rubra). Gene, 2019, 717: 144045 (doi: 10.1016/j.gene.2019.144045).
- Qi X., Liu C., Song L., Li M. PaMADS7, a MADS-box transcription factor, regulates sweet cherry fruit ripening and softening. Plant Science, 2020, 301: 110634 (doi: 10.1016/j.plantsci.2020.110634).
- Wang R., Ming M., Li J., Shi D., Qiao X., Li L., Zhang S., Wu J. Genome-wide identification of the MADS-box transcription factor family in pear (Pyrus bretschneideri) reveals evolution and functional divergence. Peer Journal, 2017, 5: e3776 (doi: 10.7717/peerj.3776).
- Zhao Q., Weber A.L., McMullen M.D., Guill K., Doebley J. MADS-box genes of maize: frequent targets of selection during domestication. Genetics Research, 2011, 93(1): 65-75 (doi: 10.1017/S0016672310000509).
- Wills D.M., Fang Z., York A.M., Holland J.B., Doebley J.F. Defining the role of the MADS-box gene, Zea agamous-like1, a target of selection during maize domestication. Journal of Heredity, 2018, 109(3): 333-338 (doi: 10.1093/jhered/esx073).
- Khong G.N., Pati P.K., Richaud F., Parizot B., Bidzinski P., Mai C.D., Bès M., Bourrié I., Meynard D., Beeckman T., Selvaraj M.G., Manabu I., Genga A.M., Brugidou C., Nang Do V., Guiderdoni E., Morel J.B., Gantet P. OsMADS26 negatively regulates resistance to pathogens and drought tolerance in rice. Plant Physiology, 2015, 169(4): 2935-2949 (doi: 10.1104/pp.15.01192).
- Chen L., Zhao Y., Xu S., Zhang Z., Xu Y., Zhang J., Chong K. OsMADS57 together with OsTB1 coordinates transcription of its target OsWRKY94 and D14 to switch its organogenesis to defense for cold adaptation in rice. New Phytologist, 2018, 218(1): 219-231 (doi: 10.1111/nph.14977).
- Wang Z., Wang F., Hong Y., Yao J., Ren Z., Shi H., Zhu J.-K. The flowering repressor SVP confers drought resistance in arabidopsis by regulating abscisic acid catabolism. Molecular Plant, 2018, 11(9): 1184-1197 (doi: 10.1016/j.molp.2018.06.009).
- Guo X., Chen G., Cui B., Gao Q., Guo J.-E., Li A., Zhang L., Hu Z. Solanum lycopersicum agamous-like MADS-box protein AGL15-like gene, SlMBP11, confers salt stress tolerance. Molecular Breeding, 2016, 36: 125 (doi: 10.1007/s11032-016-0544-1).
- Yin W., Hu Z., Cui B., Guo X., Hu J., Zhu Z., Chen G. Suppression of the MADS-box gene SlMBP8 accelerates fruit ripening of tomato (Solanum lycopersicum). Plant Physiology and Biochemistry, 2017, 118: 235-244 (doi: 10.1016/j.plaphy.2017.06.019).
- Lozano R., Angosto T., Gómez P., Payán C., Capel J., Huijser P., Salinas J., Martinez-Zapater J.M. Tomato flower abnormalities induced by low temperatures are associated with changes of expression of MADS-Box genes. Plant Physiology, 1998, 117(1): 91-100 (doi: 10.1104/pp.117.1.91).
- Müller F., Xu J., Kristensen L., Wolters-Arts M., de Groot P. F. M., Jansma S. Y., Mariani C., Park S., Rieu I. High-temperature-induced defects in tomato (Solanum lycopersicum) anther and pollen development are associated with reduced expression of B-class floral patterning genes. PLoS ONE, 2016, 11(12): e0167614 (doi: 10.1371/journal.pone.0167614).
- Chen R., Ma J., Luo D., Hou X., Ma F., Zhang Y., Meng Y., Zhang H., Guo W. CaMADS, a MADS-box transcription factor from pepper, plays an important role in the response to cold, salt, and osmotic stress. Plant Science, 2019, 280: 164-174 (doi: 10.1016/j.plantsci.2018.11.020).
- Yang F., Xu F., Wang X., Liao Y., Chen Q., Meng X. Characterization and functional analysis of a MADS-box transcription factor gene (GbMADS9) from Ginkgo biloba. Scientia Horticulture, 2016, 212: 104-114 (doi: 10.1016/j.scienta.2016.09.042).
- Yu L.H., Wu J., Zhang Z.S., Miao Z.Q., Zhao P.X., Wang Z., Xiang C.B. Arabidopsis MADS-box transcription factor AGL21 acts as environmental surveillance of seed germination by regulating ABI5 expression. Molecular Plant, 2017, 10(6): 834-845 (doi: 10.1016/j.molp.2017.04.004).
- Shi S.-Y., Zhang F.-F., Gao S., Xiao K. Expression pattern and function analyses of the MADS thranscription factor genes in wheat (Triticum aestivum L.) under phosphorus-starvation condition. Journal of Integrative Agriculture, 2016, 15(8): 1703-1715 (doi: 10.1016/S2095-3119(15)61167-4).
- Zhang H., Forde B.G. An Arabidopsis MADS box gene that controls nutrient-induced changes in root architecture. Science, 1998, 279(5349): 407-409 (doi: 10.1126/science.279.5349.407).
- Swinnen G., Goossens A., Pauwels L. Lessons from domestication: targeting cis-regulatory elements for crop improvement. Trends in Plant Science, 2016, 21(6): 506-515 (doi: 10.1016/j.tplants.2016.01.014).
- Singh R., Low E.T., Ooi L.C., Ong-Abdullah M., Ting N.C., Nagappan J., Nookiah R., Amiruddin M.D., Rosli R., Manaf M.A., Chan K.L., Halim M.A., Azizi N., Lakey N., Smith S.W., Budiman M.A., Hogan M., Bacher B., Van Brunt A., Wang C., Ordway J.M., Sambanthamurthi R., Martienssen R.A. The oil palm SHELL gene controls oil yield and encodes a homologue of SEEDSTICK. Nature, 2013, 500(7462): 340-344 (doi: 10.1038/nature12356).
- Ditta G., Pinyopich A., Robles P., Pelaz S., Yanofsky M.F. The SEP4 gene of Arabidopsis thaliana functions in floral organ and meristem identity. Current Biology, 2014, 14(21): 1935-1940 (doi: 10.1016/j.cub.2004.10.028).
- Bartlett M.E. Changing MADS-box transcription factor protein—protein interactions as a mechanism for generating floral morphological diversity. Integrative and Comparative Biology, 2017, 57(6): 1312-1321 (doi: 10.1093/icb/icx067).
- He C., Si C., Teixeira da Silva J.A., Li M., Duan J. Genome-wide identification and classification of MIKC-type MADS-box genes in Streptophyte lineages and expression analyses to reveal their role in seed germination of orchid. BMC Plant Biology, 2019, 19(1): 223 (doi: 10.1186/s12870-019-1836-5).
- Gramzow L., Weilandt L., Theißen G. MADS goes genomic in conifers: towards determining the ancestral set of MADS-box genes in seed plants. Annals of Botany, 2014, 114(7): 1407-1429 (doi: 10.1093/aob/mcu066).
- Ma J., Yang Y., Luo W., Yang C., Ding P., Liu Y., Qiao L., Chang Z., Geng H., Wang P., Jiang Q., Wang J., Chen G., Wei Y., Zheng Y., Lan X. Genome-wide identification and analysis of the MADS-box gene family in bread wheat (Triticum aestivum L.). PLoS ONE, 2017, 12(7): e0181443 (doi: 10.1371/journal.pone.0181443).
- Duan W., Song X., Liu T., Huang Z., Ren J., Hou X., Li Y. Genome-wide analysis of the MADS-box gene family in Brassica rapa (Chinese cabbage). Molecular Genetics and Genomics, 2015, 290(1): 239-255 (doi: 10.1007/s00438-014-0912-7).
- Shu Y., Yu D., Wang D., Guo D., Guo C. Genome-wide survey and expression analysis of the MADS-box gene family in soybean. Molecular Biology Reports, 2013, 40(6): 3901-3911 (doi: 10.1007/s11033-012-2438-6).
- Tian Y., Dong Q., Ji Z., Chi F., Cong P., Zhou Z. Genome-wide identification and analysis of the MADS-box gene family in apple. Gene, 2015, 555(2): 277-290 (doi: 10.1016/j.gene.2014.11.018).
- Wang P., Wang S., Chen Y., Xu X., Guang X., Zhang Y. Genome-wide Analysis of the MADS-Box gene family in watermelon. Computational Biology and Chemistry, 2019, 80: 341-350 (doi: 10.1016/j.compbiolchem.2019.04.013).
- Ning K., Han Y., Chen Z., Luo C., Wang S., Zhang W., Li L., Zhang X., Fan S., Wang Q. Genome-wide analysis of MADS-box family genes during flower development in lettuce. Plant, Cell & Environment, 2019, 42(6): 1868-1881 (doi: 10.1111/pce.13523).
- Grimplet J., Martínez-Zapater J.M., Carmona M.J. Structural and functional annotation of the MADS-box transcription factor family in grapevine. BMC Genomics, 2016, 17: 80 (doi: 10.1186/s12864-016-2398-7).