doi: 10.15389/agrobiology.2020.4.816rus
УДК 579.62:579.852.11:615.33:575.113
Исследование выполнено при поддержке гранта РФФИ № 19-316-90041 «Полногеномное секвенирование штаммов бацилл, выделенных из рубцового содержимого различных жвачных животных».
ГЕНОМНЫЙ И ФЕНОТИПИЧЕСКИЙ ПОТЕНЦИАЛ АНТИМИКРОБНОЙ АКТИВНОСТИ ШТАММА БАКТЕРИИ Bacillus megaterium В-4801
Г.Ю. ЛАПТЕВ1, Е.А. ЙЫЛДЫРЫМ2, Т.П. ДУНЯШЕВ1, Л.А. ИЛЬИНА2,
Д.Г. ТЮРИНА2, В.А. ФИЛИППОВА2, Е.А. БРАЖНИК2, Н.В. ТАРЛАВИН2,
А.В. ДУБРОВИН2, Н.И. НОВИКОВА2, В.Х. МЕЛИКИДИ2, С.Н. БИКОНЯ2
Генетические детерминанты штаммов бактерий Bacillus sp., определяющие возможность биосинтеза разнообразных антимикробных соединений, представляют особый научный интерес, поскольку благодаря им эти микроорганизмы нашли широкое применение в качестве основы пробиотиков. Важный этап системного анализа механизмов пробиотического действия, в частности антимикробной активности микроорганизмов, — реконструкция его метаболической карты, то есть сбор и визуализация всех потенциально возможных процессов в клетке. В представленной работе впервые описаны потенциально заложенные генетические механизмы синтеза ряда биологически активных веществ у выделенного нами ранее штамма бактерии Bacillus megaterium В-4801, в частности возможность синтезировать каносамин — бактериоцин, относящийся к группе аминогликозидов, который может выполнять важную роль в реализации пробиотических свойств благодаря выраженной антимикробной активности. Нашей целью было изучение антимикробной активности штамма Bacillus megaterium В-4801 в отношении патогенных и условно-патогенных бактерий, а также поиск генов, связанных с антимикробной активностью, на основе полногеномного секвенирования. Штамм B. megaterium В-4801, депонированный в коллекции ООО «БИОТРОФ+», обладает выраженной пробиотической активностью. Антимикробную активность в отношении Staphylococcus aureus, Candida tropicalis, Clostridium sp., Escherichia coli оценивали методом отсроченного антагонизма (метод «колодцев»). Библиотеку ДНК для полногеномного секвенирования готовили с помощью набора Nextera XT («Illumina, Inc.», США). Нуклеотидные последовательности определяли с использованием прибора MiSeq («Illumina, Inc.», США) и комплекта реактивов MiSeq Reagent Kit v3 (300-cycle) («Illumina, Inc.», США). Недостоверные последовательности и адаптеры удаляли с помощью программы Trimmomatic-0.38. Отфильтрованные по длине не менее от 50 до 150 п.н. парноконцевые последовательности собирали de novo с использованием геномного сборщика SPAdes-3.11.1. Функциональную аннотацию генома проводили в программах PROKKA 1.12 и RAST 2.0. Оценку пула генов, связанных с антимикробной активностью, и построение метаболической карты выполняли с помощью базы данных KEGG Pathway (http://www.genome.jp/kegg/). Культуральными методами была выявлена антагонистическая активность B. megaterium В-4801 в отношении патогенных и условно-патогенных микроорганизмов. Зоны задержки роста тест-штаммов составляли от 2±0,15 до 25±1,4 мм. Геном штамма B. megaterium В-4801 был представлен одной кольцевой хромосомой размером 6 113 972 п.н., содержащей 37,5 % ГЦ-пар. Показано, что более 45 % генов B. megaterium В-4801 вовлечены в функции транспорта и метаболизма аминокислот, транскрипции, трансляции, транспорта и метаболизма углеводов, белков. Определены ключевые генетические локусы, детерминирующие синтез антимикробных метаболитов. В составе генома секвенированного штамма локализованы гены (FabD, FabF, FabG, FabZ, FabI и др.), связанные с продукцией белков, участвующих в синтезе алифатических ненасыщенных карбоновых кислот с числом углеродных атомов от 3 до 18, в частности масляной, капроновой, каприловой, каприновой, лауриновой, миристиновой, пальмитиновой, стеариновой, олеиновой. Согласно имеющимся сведениям, все эти вещества обладают выраженными антимикробными свойствами. Кроме того, нами обнаружен целый кластер генов (Asm22-24, Asm43-45, Asm47), связанных с биосинтезом бактериоцина каносамина, который относится к группе аминогликозидов, а также поликетидных ансамициновых антибиотиков из группы макролидов. Установленный пробиотический потенциал свидетельствует о роли исследованного штамма как потенциального кандидата в качестве основы для пробиотиков, в том числе для использования в животноводстве. Проведенный геномный анализ выявил новые системы оперонов, контролирующих метаболические пути синтеза антимикробных веществ, ранее не описанные для B. megaterium.
Ключевые слова: полногеномное секвенирование, Bacillusmegaterium, биосинтез жирных кислот, бактериоцины, антимикробная активность, каносамин, ансамициновые антибиотики, пробиотики.
В настоящее время в животноводстве и птицеводстве возрос интерес к натуральным биопрепаратам в связи с вступлением в силу с 1 января 2020 года Федерального закона № 280-ФЗ «Об органической продукции и о внесении изменений в отдельные законодательные акты Российской Федерации». Согласно закону, при производстве органической продукции запрещается использование антибиотиков, за исключением тех, которые разрешены к применению действующими в России национальными, межгосударственными и международными стандартами в сфере производства органической продукции.
Генетические детерминантыштаммов бактерий Bacillus sp., определяющие возможность биосинтеза разнообразных антимикробных соединений, представляют особый научный интерес, поскольку благодаря им эти микроорганизмы нашли широкое применение в качестве биопестицидов в растениеводческой практике и как основа терапевтических агентов и пробиотиков в животноводстве. По мнению некоторых исследователей (1), штаммы рода Bacillus более перспективны для создания пробиотических препаратов, нежели традиционно используемые молочнокислые бактерии, поскольку образуют в цикле развития эндоспоры и, следовательно, более устойчивы к агрессивным факторам в пищеварительной системе. В то время как Bacillus sp. — типичные комменсальные бактерии рубца и кишечника сельскохозяйственных животных (2), увеличение обилия популяций лактобактерий в рубце жвачных связывают с возникновением лактатного ацидоза, снижением рН и сокращением численности бактерий, синтезирующих целлюлазы (2).
Антимикробные бактериальные метаболиты Bacillus sp. в среднем включают 87 % органических кислот, спиртов, кетонов, алканов, альдегидов, алкенов от суммарной доли общего пула антимикробных компонентов и 13 % других веществ — рибосомных пептидов (бактериоцинов и ферментов), поликетидов, нерибосомных пептидов (3). На примере одного из штаммов B. subtilis показано, что по меньшей мере 4-5 % его генома приходится на долю оперонов, связанных с синтезом антимикробных соединений (4).
Интерес к изучению способности штаммов Bacillus sp. синтезировать бактериоцины, а также пептидные и липопептидные антибиотики в настоящее время возрос (5, 6), появляются сведения о неизвестных ранее веществах (7-9). Многие из бактериоцинов Bacillus sp. относят к лантибиотикам категории посттрансляционно модифицированных пептидов, широко распространенных среди различных бактериальных таксонов.
Представители рода Bacillus также продуцируют множество немодифицированных бактериоцинов, часть из которых сходны с педиоциноподобными бактериоцинами молочнокислых бактерий, в то время как другие имеют совершенно новые пептидные последовательности (10). Бактериоцины Bacillus sp. представляют практический интерес в связи с возможностью ингибировать разнообразные патогенные формы, включая грамотрицательные и грамположительные бактерии, дрожжи и микромицеты (7-9).
Значительно меньше работ посвящено изучению синтеза микроорганизмами рода Bacillus органических кислот. Есть указания на возможность продуцирования некоторыми представителями Bacillus sp. молочной кислоты (11), известной своей антимикробной активностью в отношении Escherichia coli, Pseudomonas aeruginosa, Salmonella enterica и др. (12). Штамм Bacillusmegaterium ELI 24, выделенный из почвы в Мексике, секретировал значительные количества янтарной кислоты, которая была идентифицирована спектроскопическими методами (13). С использованием хроматографического анализа было показано, что штамм Bacillus megaterium P1, изолированный из почвы, в зависимости от состава среды для культивирования синтезировал летучие (муравьиная, уксусная, пропионовая, масляная) и нелетучие (щавелевая, яблочная, янтарная, винная и лимонная) органические кислоты в различных комбинациях (14).
Полногеномное секвенирование в настоящий момент признано наиболее эффективной технологией подробной генетической характеристики штаммов микроорганизмов, их свойств и метаболических процессов (15). Важным этапом системного анализа механизмов пробиотического действия, в частности антимикробной активности микроорганизмов, может быть реконструкция его метаболической карты, то есть сбор и визуализация всех процессов, потенциально происходящих в клетке. Экспериментальные данные о степени антимикробной активности в отношении патогенных форм в совокупности с результатами полногеномного секвенирования позволяют реконструировать модели, максимально приближенные к реальности. Их построение дает возможность прогнозировать взаимоотношения микроорганизма с различными патогенными и условно-патоген-ными формами в среде пищеварительной системы животных с целью создания эффективных пробиотических биопрепаратов.
В настоящее время достаточно активно ведется работа по изучению геномов штаммов Bacillus sp., перспективных для создания биопрепаратов. P. Li с соавт. (15) применяли метод полногеномного секвенирования для оценки пробиотического потенциала штамма Bacillus sp. DU-106 — активного продуцента L-молочной кислоты. Другие исследователи (16) применяли эту методику для изучения генома Bacillus clausii ENTPro — основы для производства пробиотика Enterogermina («Sanofi Synthelabo Pvt. Ltd.», Индия). Секвенированием полных геномов у B. megaterium занимались M. Eppinger с соавт. (17), проанализировавшие штаммы QM B1551 и DSM319. J.I. Vílchez с соавт. (18) изучили последовательность штамма TG1-E1, перспективного для создания землеудобрительных препаратов. L. Liu с соавт. (19) исследовали геном штамма WSH-002, перспективного для создания рекомбинантных белков и витаминов.
В представленной работе у штамма бактерии Bacillus megaterium впервые описаны потенциально заложенные генетические механизмы синтеза ряда биологически активных веществ, в частности способность синтезировать каносамин — бактериоцин, относящийся к группе аминогликозидов, который может выполнять важную роль в реализации пробиотических свойств благодаря выраженной антимикробной активности.
Нашей целью было изучение антимикробной активности штамма Bacillus megaterium В-4801 в отношении патогенных и условно-патогенных бактерий, а также поиск генов, связанных с антимикробной активностью, на основе полногеномного секвенирования.
Методика. Материалом для исследования служил штамм B. megaterium В-4801 из коллекции ООО «БИОТРОФ+», обладающий выраженной пробиотической активностью. Тест-культуры, которые использовались в качестве объектов для исследования антагонистических свойств, были получены из Всероссийской коллекции непатогенных микроорганизмов сельскохозяйственного назначения Всероссийского НИИ сельскохозяйственной микробиологии (г. Санкт-Петербург—Пушкин).
Антимикробную активность в отношении патогенных и условно-патогенных микроорганизмов (Staphylococcus aureus, Candida tropicalis, Clostridium sp., Escherichia coli) оценивали in vitro методом отсроченного антагонизма методом (метод «колодцев»). Для этого 100 мкл взвеси культуры B. megaterium В-4801 (107 КОЕ/мл) помещали в лунку в центре чашки Петри с подсушенной в течение 24-48 ч агаризованной средой ГРМ (ФБУН ГНЦ прикладной микробиологии и биотехнологии, Россия) с добавлением глюкозы (7 г/л), содержащей культуры условно-патогенных тест-штаммов. Через 48 ч инкубации при 37±1 °С учитывали величину зоны угнетения роста тест-штаммов.
Выделение ДНК проводили по стандартным методикам с использованием набора Genomic DNA Purification Kit («Fermentas, Inc.», Литва) согласно прилагаемой инструкции (2). Анализ основан на селективном детергентно-опосредованном осаждении ДНК из субстрата с применением растворов для лизиса клеточных стенок и осаждения ДНК, раствора 1,2 М хлорида натрия, хлороформа.
Библиотеку ДНК для полногеномного секвенирования готовили с помощью набора Nextera XT («Illumina, Inc.», США). Нуклеотидные последовательности определяли с использованием прибора MiSeq («Illumina, Inc.», США) совместно с комплектом реактивов MiSeq Reagent Kit v3 (300-cycle) («Illumina, Inc.», США). Недостоверные последовательности и адаптеры удаляли в программе Trimmomatic-0.38 (https://www.osc.edu/book/ex-port/html/4385) (20). Отфильтрованные по дли-не не менее чем от 50 до 150 п.н. парноконцевые последовательности собирали de novo с использованием геномного сборщика SPAdes-3.11.1 (http://cab.spbu.ru/software/spades/) (21). Функциональную аннотацию генома проводили в программах PROKKA 1.12 (https://github.com/kbase-apps/ProkkaAnnotation) (22) и RAST 2.0 (https://rast.nmpdr.org) (23). Для оценки пула генов, связанных с антимикробной активностью, и построения метаболической карты использовали базу данных KEGG Pathway (http://www.genome.jp/kegg/) (24, 25).
Математическую и статистическую обработку результатов проводили с помощью программных пакетов Microsoft Office Excel 2003, R-Studio (Version 1.1.453) (https://rstudio.com). Оценивали среднее значение для каждой выборки (M) и среднеквадратическое отклонение (±SD).
Результаты. Штамм B. megaterium В-4801 обладал выраженным антагонистическим действием в отношении тест-культур Staphylococcus aureus и Candida tropicalis: размер зон задержки роста составлял соответственно 25±1,4 и 10±0,7 мм (рис. 1). Clostridium sp. оказался наиболее устойчивым к воздействию B. megaterium В-4801 (2±0,15 мм). Для Escherichia coli величина зоны задержки роста составляла 5±0,3 мм. Это позволяет предположить, что в составе культуральной жидкости B. megaterium В-4801 присутствуют антимикробные вещества, диффундирующие в агар.
Полученные в этом тесте данные имеют важное практическое значение, поскольку S. aureus связан с возникновением заболеваний крупного рогатого скота, прежде всего мастита (26). Ранее нами была доказана связь между увеличением обилия Staphylococcus sp.в рубце и возрастанием количества соматических клеток в молоке дойных коров (2). C. tropicalis также патогенен для крупного рогатого скота, в частности может распространяться через кровоток к периферическим органам, обнаружена его связь с абортированием (27, 28). Эти факты позволяют сделать вывод, что штамм B. megaterium В-4801 перспективен при разработке биоконтролирующих средств для подавления патогенной микробиоты, в частности посредством его интродукции в пищеварительную систему животных.
Геном штамма B. megaterium В-4801 был аннотирован с помощью набора инструментов RAST с присвоением уникального геномного идентификатора 1404.252. Геном былпредставлен одной кольцевой хромосомой размером 6 113 972 п.н., содержащей 37,5 % ГЦ-пар. В состав хромосомы входили 6324 открытые рамки считывания, способные определять синтез полипептидов, 129 — для синтеза тРНК и 6 — для рРНК. Плазмидная часть включала 78379 п.н. и содержала 23,5 % ГЦ-пар, что было на 14 % меньше, чем в хромосоме.
Сравнение контигов исследуемого штамма с базой данных нуклеотидных последовательностей NCBI (https://www.ncbi.nlm.nih.gov/genome/mic-robes/) с помощью базы данных PATRIC (https://www.patricbrc.org) показало высокую степень его сходства с геномом B. megaterium QM B1551 545693.3 (рис. 2). Оба этих штамма были аналогичны двум другим штаммам B. megaterium. Интересно, чтоупомянутые четыре штамма B. megaterium оказались достаточно близко связаны с кластером Clostridium sp. Ранее S. Porwal с соавт. (29) показали, что штамм B. megaterium (с содержанием ГЦ-пар от 38 до 39 %) лишь отдаленно связан с видами B. cereus и B. subtilis, что противоречило традиционным представлениям.
Согласно результатам анализа метаболических подсистем, то есть групп белков, которые совместно реализуют определенный биологический процесс, более 45 % генов B. megaterium В-4801 были вовлечены в функции транспорта и метаболизма аминокислот, транскрипции, трансляции, транспорта и метаболизма углеводов, белков (рис. 3). Исследуемый штамм обладал продуктами для функционирования полного набора метаболических путей, включая гликолиз, цикл трикарбоновых кислот и пентозофосфатный путь.
Факт того, что у B. megaterium В-4801 значительное количество генов (17,5 %) было связано с метаболизмом углеводов, вполне закономерен, поскольку доказано, что Bacillus sp. продуцируют множество антибиотикоподобных соединений нерибосомной природы и органических кислот, для синтеза которых требуется активный углеводный обмен (4, 13). Среди множества синтезируемых белков важную роль играют ферменты, определяющие поступление в клетку сахаров и этапы их окисления (пермеазы, гидролазы). В результате этих процессов образуются, в частности, пируват, 2-оксо-глутарат, оксалоацетат и ацетил-КоА, которые служат предшественниками синтеза жирных кислот, аминокислот, поликетидов и целого ряда других жизненно необходимых метаболитов.
Важно присутствие в геноме заметного количества (более 20 %) генов, реализующих взаимодействия с окружающей средой, в частности связанных с формированием клеточной оболочки и капсулированием, моторикой и хемотаксисом, клеточным сигналингом, реакцией на стресс, что указывает на высокий потенциал пробиотической активности. Вероятно, этот набор генов может способствовать выживанию штамма в условиях агрессивной среды желудочно-кишечного тракта, а также адгезии к клеткам эпителия хозяина. Значительная доля генома аннотировалась как связанная с синтезом витаминов, в частности В1, В2, В9 и биотина, играющих важную роль во многих обменных процессах в макроорганизмах (32). Ранее L. Liu с соавт. (19) при проведении полногеномного секвенирования выявили потенциальную способность штамма B. megaterium к синтезу витаминов.
Мы определили у B. megaterium В-4801 ключевые генетические локусы, детерминирующие синтез ряда антимикробных метаболитов — жирных кислот, каносамина, относящегося к группе аминогликозидов, а также поликетидных ансамициновых бактериоцинов из группы макролидов. Идентификацию ключевых продуктов, вовлеченных у штамма B. megaterium В-4801 в пути синтеза антимикробных метаболитов проводили с использованием базы данных Kegg Pathway (рис. 4).
В геноме B. megaterium В-4801 были локализованы гены (FabD, FabF, FabG, FabZ, FabI и др.), связанные с продукцией белков, которые участвуют в синтезе алифатических ненасыщенных карбоновых кислот с числом углеродных атомов от 3 до 18, в частности масляной, капроновой, каприловой, каприновой, лауриновой, миристиновой, пальмитиновой, стеариновой, олеиновой. Метаболические карты синтеза некоторых карбоновых кислот у штамма B. megaterium В-4801 представлены на рисунке 4 в качестве иллюстрации.
Согласно базе данных KEGG Pathway (см. рис. 4), предшественники для биосинтеза жирных кислот у B. megaterium В-4801 образовывались из пула ацетил-КоА. На первом этапе происходил перенос ацетильной группы от ацетил-КоА на молекулу ацилпереносящего белка (АПБ). Ацетил-АПБ выполняет функцию затравки, к которой присоединяется С2-фрагмент. Донором С2-фрагмента служила молекула малонил-АПБ, синтезирующаяся также из ацетил-КоА. Эту реакцию катализировал фермент FabD (малониловый СоА-ациловый белок-носитель трансацилаза, ЕС 2.3.1.39), что ранее (33) уже было показано на примере Streptomycescoelicolor, но не описано для Bacillussp.
Дальнейшие превращения у штамма B. megaterium В-4801 имели некоторые различия для жирных кислот и будут описаны нами на примере масляной кислоты. В результате присоединения С2-фрагмента к ацетил-АПБ образовывался ацетоацетил-АПБ. Судя по данным базы KEGG Pathway, белок FabF катализировал конденсацию ацетил-КоА с малонил-АПБ для получения ацетоацетил-АПБ. Подобный путь был ранее описан для Escherichia coli (34). По мнению авторов, синтетический путь, приводящий к образованию жирных кислот у E. coli, требует двух специализированных белков — FabA и FabB. Однако гены, связанные с синтезом FabA и FabB, встречаются только у грамотрицательных представителей Proteobacteria. В то же время показано (35), что у грамположительной бактерии Enterococcus faecalis имеется белок, аннотированный как FabZ1, который функционально заменяет белок E. coli FabA. Вероятно, FabF у B. megaterium В-4801аналогичен по функциям FabА и FabB у E. coli.
На следующем этапе у штамма B. megaterium В-4801с помощью серии ферментативных реакций с участием белков FabG, FabZ, FabI (FabK, FabL) происходило восстановление окисленных углеродных атомов ацетоацетил-АПБ, приводящее через стадии метаболитов-предшественников к образованию бутирила-АПБ — радикала масляной кислоты. Завершающую стадию в биосинтезе жирных кислот катализировал фермент FabI (синонимы FabK, FabL), инициирующий гидратацию 2,3-двойной связи в производных эноил-АПБ, что было ранее показано для E. coli (36).
Таким образом, при секвенировании генома штамма B. megaterium В-4801 нами были выявлены практически все основные ферменты, ответственные за образование жирных кислот (С3-С18). Ранее у штамма Bacillus sp. DU-106 (15) были обнаружены ферменты, участвующие в синтезе органических кислот, в частности в образовании лактата, включая L-лактатдегидрогеназу, D-лактатдегидрогеназу, лактальдегиддегидрогеназу и малат/лактатдегидрогеназу. Отметим, что подобные ферменты не выявляли у B. cereus (37). Тем не менее в более позднем исследовании (15) у Bacilluscereus DU-106 в составе генома были выявлены гены, связанные с синтезом лактата.
Данные, полученные на геномном уровне, стали дополнительным подтверждением того, что штамм B. megaterium В-4801обладает потенциальными пробиотическими свойствами, поскольку антимикробная активность указанных кислот подтверждена исследованиями. Так, широко известна выраженная антимикробная активность масляной кислоты в отношении Escherichia coli, Salmonella enterica serovar Typhimurium, Campylobacter jejuni, Clostridium perfringens, Streptococcus pneumoniae, Str. suis (38). Исследования показали, что ряд жирных кислот, в частности капроновая, каприловая, каприновая, лауриновая и миристиновая, проявляли антагонизм в отношении Streptococcusmutans, Str. gordonii, Str. sanguis, Candida albicans, Fusobacterium nucleatum и Porphyromonas gingivalis (39). V. Prabhadevi с соавт. (40) отмечали антимикробную активность стеариновой кислоты, а M.M. Rahman с соавт. (41) — пальмитиновой.
Интересно отметить, что у B. megaterium В-4801 был обнаружен целый кластер генов (Asm22-24, Asm43-45, Asm47), связанных с биосинтезом бактериоцина каносамина, относящегося к группе аминогликозидов, а также поликетидных ансамициновых антибиотиков из группы макролидов. Так, гены Asm43-45 ассоциированы с биосинтезом антимикробного вещества каносамина (3-амино-3-дезокси-D-глюкозы) через промежуточное звено — УДП-a-Д-каносамин. В работе S. Umezawa с соавт. (42) продемонстрирован подобный путь биосинтеза каносамина у B. pumilus (ранее известного как B. aminoglucosidicus), хотя вовлеченные в процесс гены не были идентифицированы. Способность к синтезу каносамина обнаружена и у Bacillus cereus (43). Позже у B. cereus был описан ряд генов, участвующих в биосинтезе каносамина, хотя сам путь не был определен (44). N.D. Vetter с соавт. (45) обнаружили ряд генов, в частности NtdA, NtdB и NtdC у Bacillus subtilis 168, связанных с биосинтезом этого антимикробного вещества. Возможность синтеза каносамина у штаммов видаBacillusmegateriumвыявлена нами впервые.
Каносамин и УДП-a-Д-каносамин служат также промежуточными звеньями в биосинтезе 3-амино-5-гидроксибензойной кислоты 46). 3-Амино-5-гидроксибензойная кислота используется в клетках микроорганизмов в качестве стартового блока для сборки углеродного каркаса прекурсоров ансамициновых антибиотиков при помощи модулярной поликетидной синтазы I. Ансамицины — это класс бактериальных макроциклических поликетидов, продуцируемых главным образом представителями филума Actinobacteria и рода Bacillus (47, 48). Доказано (49), что ансамицины проявляют широкий спектр антимикробной активности. Для реализации синтеза 3-амино-5-гидроксибензойной кислоты у штамма бактерии B. megaterium В-4801 нами обнаружен фермент транскетолаза, а также ряд генов, в частности Asm47, Asm23 и Asm24. Так, хорошо известно (50), что Asm23 кодирует дегидратазу дегидрохината II типа, катализирующую дегидратацию 5-дезокси-5-амино-3-дегидрохината — предшественника 3-амино-5-гидроксибензойной кислоты. Ген Asm24 предположительно катализирует дегидратацию 5-дезокси-5-амино-3-дегидроксима-та до 3-амино-5-гидроксибензойной кислоты. Ранее у Actinosynnema pretiosum ssp. auranticum ATCC 31565 был обнаружен набор генов Asm22-24, Asm43-45 и Asm47, которые были связаны с синтезом 3-амино-5-гидрок-сибензойной кислоты (50).
Возможность синтеза бактериоцинов штаммом B. megaterium В-4801 закономерна, поскольку бактерии рода Bacillus — известные продуценты подобных веществ (8). Например, галлидермин, ген которого идентифицирован в составе генома B. clausii, эффективно предотвращает образование биопленки у патогенов S. aureus и S. epidermidis (51). Лактицин 3147 A2 и лейкоциклин Q, найденные у B. amyloliquefaciens, — бактериоцины широкого спектра действия. Лактицин был эффективно использован в лечении бактериальных маститов, стафилококковых и энтерококковых инфекций, включая патологии, вызванные ванкомицин-резистентными энтерококками (9). Лихеницидин VK21A2, обнаруженный у B. paralicheniformis, проявлял антимикробную активность в отношении нескольких патогенных штаммов — Listeria monocytogenes, метициллин-резистентного S. aureus и ванкомицин-резистентного энтерококка (7). У штамма B. mega-terium KC246043.1 обнаружено биологически активное соединение, сходное с бацитрацином (52). I.A. Malanicheva с соавт. (53) выявили у B. me-gaterium возможность продуцировать антибактериальные антибиотики, различающиеся по спектру действия. Три из них относились к пептидным антибиотикам, еще три — к ранее не описанным соединениям. Все вещества были активны против метициллинрезистентного штамма Staphylococcus aureus INA 00761, а также против Pseudomonas aeruginosa ATCC 27853 и Escherichia coli ATCC 25922.
Обобщая сказанное, следует отметить, что ранее в нескольких исследованиях (19, 18) были получены полногеномные последовательности различных штаммов B. megaterium, но описанные нами генетические механизмы синтеза ряда жирных кислот и бактериоцинов у этих микроорганизмов обнаружены не были и приводятся нами впервые.
Итак, у штамма Bacillus megaterium В-4801 нами выявлена антагонистическая активность в отношении патогенных и условно-патогенных микроорганизмов. Геном штамма B. megaterium В-4801 представлен одной кольцевой хромосомой размером 6 113 972 п.н., содержащей 37,5 % ГЦ-пар. Более 45 % генов B. megaterium В-4801 вовлечены в функции транспорта и метаболизма аминокислот, транскрипции, трансляции, транспорта и метаболизма углеводов, белков. Определены ключевые генетические локусы, детерминирующие синтез антимикробных метаболитов. В составе генома секвенированного штамма локализованы гены (FabD, FabF, FabG, FabZ, FabI и др.), связанные с продукцией белков, которые участвуют в синтезе алифатических ненасыщенных карбоновых кислот с числом углеродных атомов от 3 до 18, в частности масляной, капроновой, каприловой, каприновой, лауриновой, миристиновой, пальмитиновой, стеариновой, олеиновой. Известно, что все эти вещества обладают выраженными антимикробными свойствами. У B. megaterium В-4801 мы обнаружили кластер генов (Asm22-24, Asm43-45, Asm47), связанных с биосинтезом бактериоцина каносамина, относящегося к группе аминогликозидов, а также поликетидных ансамициновых антибиотиков из группы макролидов. Созданный нами генетический паспорт штамма B. megaterium В-4801 представляет фундаментальный научный интерес, а также содержит ценную коммерческого информацию. Выявленный пробиотический потенциал B. megaterium В-4801 свидетельствует о роли этого штамма как потенциального кандидата в качестве основы для пробиотиков, в том числе для животноводства. Продолжением представленного исследования может быть экспериментальное изучение жирных кислот и бактериоцинов с помощью аналитических методов для более глубокого понимания механизма их пробиотического действия. Кроме того, наличие в геноме бактерии оперонов, которые кодируют продукты, потенциально вовлеченные в различные метаболические пути, не означает, что эти пути функциональны. Представляет интерес подтверждение их функциональности посредством анализа продукции соответствующих белков или метаболитов при помощи протеомных и метаболомных подходов, а также адресного анализа конкретных соединений.
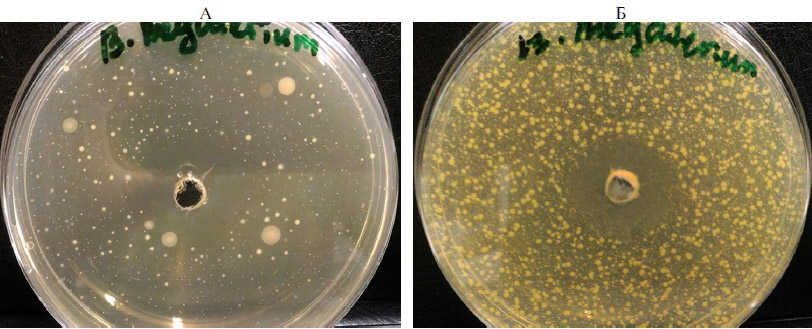
Рис. 1. Антагонистическое действие Bfcillus megaterium В-4801 в отношении тест-культур Staphylococcus aureus (А) и Candida tropicalis (Б). Чашки Петри засеяны газоном тест-орга-низмов, в центре визуализируется зона подавления тест-культуры патогена.

Рис. 2. Дендрограмма, отражающая филогенетическое родство исследуемого штамма Bacillus megaterium В-4801 с наиболее близкими репрезентативными геномами бактерий, представленными в базе данных NCBI (https://www.ncbi.nlm.nih.gov/genome/microbes/). Расстояние между ними оценено с применением технологии Mash/MinHash (30) и построено средствами PATRIC (31).
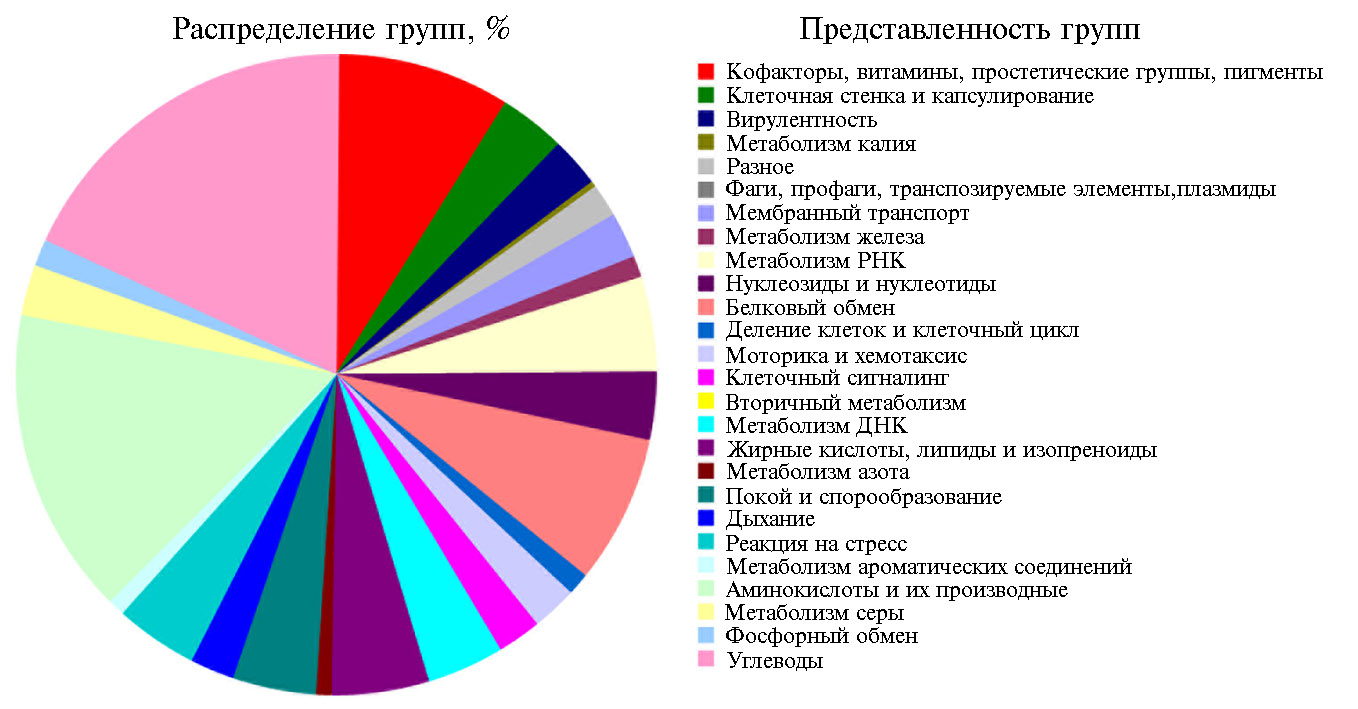
Рис. 3. Метаболические системы штамма Bacillus megaterium В-4801 на основе результатов функциональной аннотации согласно базе данных RAST (https://rast.nmpdr.org).
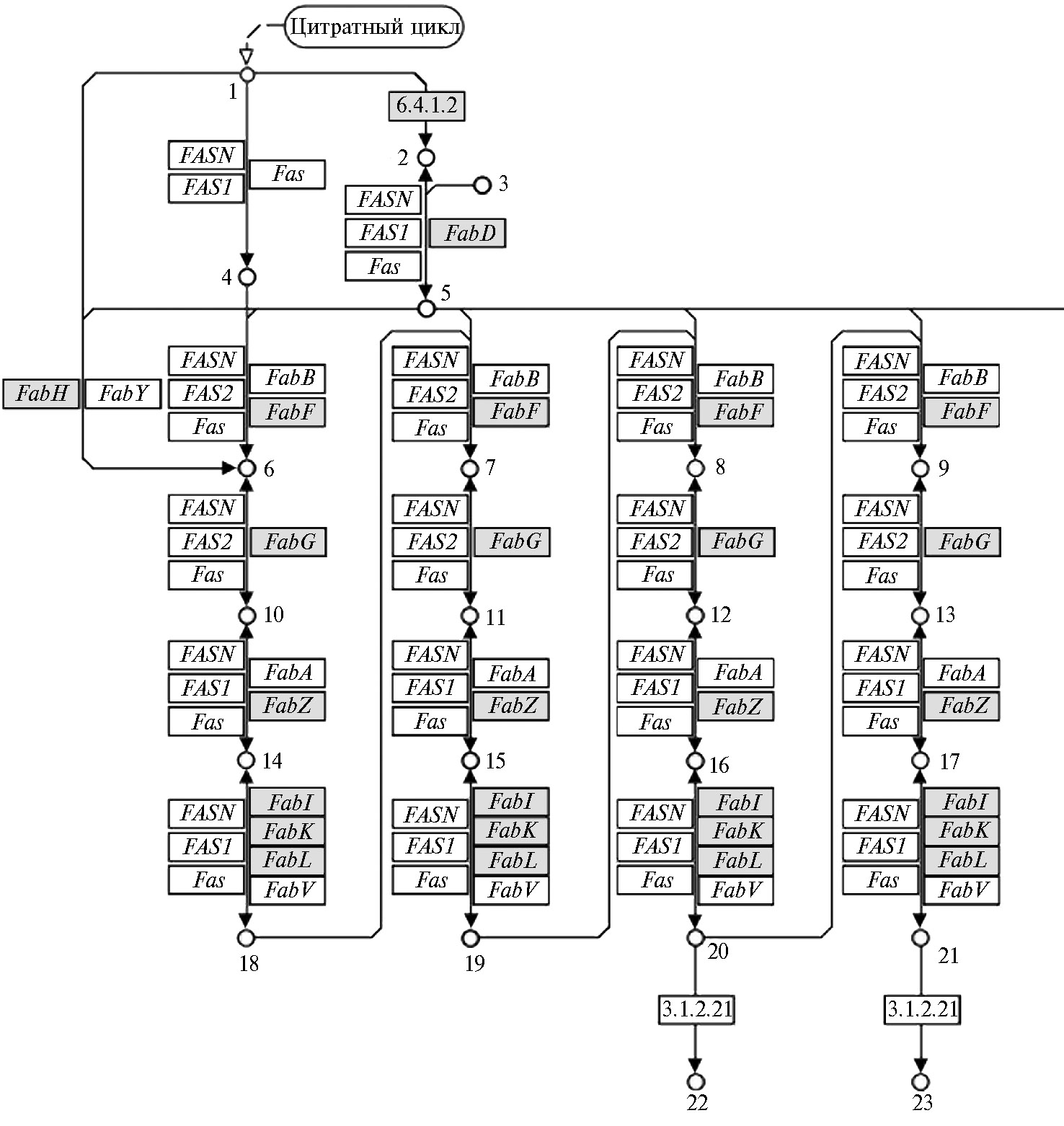
Рис. 4. Метаболические пути, приводящие к образованию жирных кислот и их предшественников у штамма бактерии Bacillus megaterium В-4801 (схема построена с использованием базы данных KEGG Pathway, http://www.genome.jp/kegg/): 1 — ацетил-КоА, 2 — малонил-КоА, 3 — ацилпереносящий белок (АПБ), 4 — ацетил-[АПБ], 5 — малонил-[АПБ], 6 — ацетоацетил-[АПБ], 7 — 3-оксогексаноил-[АПБ], 8 — 3-оксооктаноил-[АПБ], 9 — 3-оксодеканоил-[АПБ], 10 — (R)-3-гидроксибутаноил-[АПБ], 11 — (R)-3-гидроксигексаноил-[АПБ], 12 — (R)-3-гидроксиоктаноил-[АПБ], 13 — (R)-3-гидроксидеканоил-[АПБ], 14 — бут-2-эноил-[АПБ], 15 — транс-гекс-2-эноил-[АПБ], 16 — транс-окт-2-эноил-[АПБ], 17 — транс-дек-2-эноил-[АПБ], 18 — бутирил-[АПБ], 19 — гексаноил-[АПБ], 20 — октаноил-[АПБ], 21 — деканоил-[АПБ], 22 — каприловая кислота, 23 — каприновая кислота. Цифрами в прямоугольниках обозначены коды ферментов в номенклатуре IUPAC; серым цветом отмечены гены, присутствующие в геноме; стрелочки, направленные на кружки, обозначают направление шагов в реакции.
ЛИТЕРАТУРА
- Hong H.A., Duc L.H., Cutting S.M. The use of bacterial spore formers as probiotics. FEMS Microbiology Reviews, 2010, 29(4): 813-835 (doi: 10.1016/j.femsre.2004.12.001).
- Ильина Л.А. Изучение микрофлоры рубца крупного рогатого скота на основе молекулярно-биологического метода T-RFLP с целью разработки способов ее оптимизации. Канд. дис. СПб, 2012.
- Caulier S., Nannan C., Gillis A., Licciardi F., Bragard C., Mahillon J. Overview of the antimicrobial compounds produced by members of the Bacillus subtilis. Frontiers in Microbiology, 2019, 10: 302 (doi: 10.3389/fmicb.2019.00302).
- Stein T. Bacillus subtilis antibiotics: structures, syntheses and specific functions. Molecular Microbiology, 2005, 56(4): 845-857 (doi: 10.1111/j.1365-2958.2005.04587.x).
- Cotter P.D., Hill C., Ross R.P. What's in a name? Class distinction for bacteriocins. Nature Reviews Microbiology, 2006, 4(2): 160 (doi: 10.1038/nrmicro1273-c2).
- Gabrielsen C., Brede D.A., Nes I.F., Diep D.B. Circular bacteriocins: biosynthesis and mode of action. Applied and Environmental Microbiology, 2014, 80(22): 6854-6862 (doi: 10.1128/AEM.02284-14).
- Begley M., Cotter P.D., Hill C., Ross R.P. Identification of a novel two-peptide lantibiotic, lichenicidin, following rational genome mining for LanM proteins. Applied and Environmental Microbiology, 2009, 75(17): 5451-5460 (doi: 10.1128/AEM.00730-09).
- Saising J., Dube L., Ziebandt A.-K., Voravuthikunchai S.P., Nega M., Götz F. Activity of gallidermin on Staphylococcus aureus and Staphylococcus epidermidis biofilms. Antimicrobial Agents and Chemotherapy, 2012, 56(11): 5804-5810 (doi: 10.1128/AAC.01296-12).
- Field D., Cotter P.D., Hill C., Ross R.P. Bioengineering lantibiotics for therapeutic success. Frontiers in Microbiology, 2015, 6: 1363 (doi: 10.3389/fmicb.2015.01363).
- Abriouel H., Franz C.M., Ben Omar N., Gálvez A. Diversity and applications of Bacillus bacteriocins. FEMS Microbiology Reviews, 2011, 35(1): 201‐232 (doi: 10.1111/j.1574-6976.2010.00244.x).
- Poudel P., Tashiro Y., Miyamoto H., Miyamoto H., Okugawa Y., Sakai K. Direct starch fermentation to L-lactic acid by a newly isolated thermophilic strain, Bacillus sp. Journal of Industrial Microbiology and Biotechnology, 2015, 42: 143-149 (doi: 10.1007/s10295-014-1534-0).
- Alakomi H.L., Skyttä E., Saarela M., Mattila-Sandholm T., Latva-Kala K., Helander I.M. Lactic acid permeabilizes gram-negative bacteria by disrupting the outer membrane. Applied and Environmental Microbiology, 2000, 66(5): 2001‐2005 (doi: 10.1128/aem.66.5.2001-2005.2000).
- Ortiz A., Sansinenea E. Succinic acid production as secondary metabolite from Bacillus megaterium ELI24. The Natural Products Journal, 2020, 10(2): 154-157 (doi: 10.2174/2210315509666190410153811).
- Chuanqing Z., Jiang A.U., Huang A., Qi W. Cao X. Guangxiang studies on the acid-production characteristics of Bacillus megaterium strain P17. AIP Conference Proceedings, 2017, 1839(1): 020054 (doi: 10.1063/1.4982419).
- Li P., Tian W., Jiang Z., Liang Z., Wu X., Du B. Genomic characterization and probiotic potency of Bacillus sp. DU-106, a highly effective producer of L-lactic acid isolated from fermented yogurt. Frontiers in Microbiology, 2018, 9: 2216 (doi: 10.3389/fmicb.2018.02216).
- Khatri I., Sharma G., Subramanian S. Composite genome sequence of Bacillus clausii, a probiotic commercially available as Enterogermina®, and insights into its probiotic properties. BMC Microbiology, 2019, 19(1): 307 (doi: 10.1186/s12866-019-1680-7).
- Eppinger M., Boyke B., Mitrick A.J., Janaka N.E., Kirthi K.K., Koenig S.S.K., Creasy H.H., Rosovitz M.J., Riley D.R., Daugherty S., Martin M., Elbourne L.D.H., Paulsen I., Biedendieck R., Braun C., Grayburn S., Dhingra S., Lukyanchuk V., Ball B., Ul-Qamar R., Seibel J., Bremer E., Jahn D., Ravel J., Vary P.S. Genome sequences of the biotechnologically important Bacillus megaterium strains QM B1551 and DSM319. Journal of Bacteriology, 2011, 193(16): 4199-4213 (doi: 10.1128/JB.00449-11).
- Vílchez J.I., Tang Q., Kaushal R., Wang W., Suhui L., Danxia H., Zhaoqing C., Heng Z., Renyi L., Huiming Z. Complete genome sequence of Bacillus megaterium strain TG1-E1, a plant drought tolerance-enhancing bacterium. Microbiology Resource Announcements, 2018, 7(12): e00842-18 (doi: ).
- Liu L., Li Y., Zhang J., Zou W., Zhou Z., Liu J., Li X., Wang L., Chen J. Complete genome sequence of the industrial strain Bacillus megaterium WSH-002. Journal of Bacteriology, 2011, 193(22): 6389-6390 (doi: 10.1128/JB.06066-11).
- Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114‐2120 (doi: 10.1093/bioinformatics/btu170).
- Nurk S., Bankevich A., Antipov D., Gurevich A., Korobeynikov A., Lapidus A., Prjibelsky A., Pyshkin A., Sirotkin A., Sirotkin Y., Stepanauskas R., McLean J., Lasken R., Clingenpeel S.R., Woyke T., Tesler G., Alekseyev M.A., Pevzner P.A. Assembling genomes and mini-metagenomes from highly chimeric reads. In: Research in Computational Molecular Biology. RECOMB 2013. Lecture Notes in Computer Science, vol. 7821. Springer, Berlin, Heidelberg, 2013: 158-170 (doi: 10.1007/978-3-642-37195-0_13).
- Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics, 2014, 30(14): 2068‐2069 (doi: 10.1093/bioinformatics/btu153).
- Aziz R.K. The RAST server: rapid annotations using subsystems technology. BMC Genomics, 2008, 9(75) (doi: 10.1186/1471-2164-9-75).
- Kanehisa M., Goto S. KEGG: Kyoto encyclopedia of genes and genomes. Nucleic Acids Research, 2000, 28(1): 27-30 (doi: 10.1093/nar/28.1.27).
- Kanehisa M., Goto S., Sato Y., Furumichi, M. Tanabe M. KEGG for integration and interpretation of large-scale molecular datasets. Nucleic Acids Research, 2012, 40(D1): D109-D114 (doi: 10.1093/nar/gkr988).
- Boss R., Cosandey A., Luini M., Artursson K., Bardiau M., Breitenwieser F., Hehenberger E., Lam Th., Mansfeld M., Michel A., Mösslacher G., Naskova J., Nelson S., Podpečan O., Raemy A., Ryan E., Salat O., Zangerl P., Steiner A., Graber H.U. Bovine Staphylococcus aureus: Subtyping, evolution, and zoonotic transfer. Journal of Dairy Science, 2016, 99(1): 515‐528 (doi: 10.3168/jds.2015-9589).
- Loken K.I., Thompson E.S., Hoyt H.H., Ball R.A. Infection of the bovine udder with Candida tropicalis. Journal of the American Veterinary Medical Association, 1959, 134(9): 401‐403.
- Wohlgemuth K., Knudtson W. Bovine abortion associated with Candida tropicalis. Journal of the American Veterinary Medical Association, 1973, 162(6): 460‐461.
- Porwal S., Lal S., Cheema S., Kalia V.C. Phylogeny in aid of the present and novel microbial lineages: diversity in Bacillus. PLoS ONE, 2009, 4(2): e4438 (doi: 10.1371/journal.pone.0004438).
- Ondov B.D., Treangen T.J., Melsted P., Mallonee A.B., Bergman N.H., Koren S., Phillippy A.M. Mash: fast genome and metagenome distance estimation using MinHash. Genome Biology, 2016, 17: 132 (doi: 10.1186/s13059-016-0997-x).
- Wattam A.R., Davis J.J., Assaf R., Boisvert S., Brettin T., Bun C., Conrad N., Dietrich E.M., Disz T., Gabbard J.L., Gerdes S., Henry C.S., Kenyon R.W., Machi D., Mao C., Nordberg E.K., Olsen G.J., Murphy-Olson D.E., Olson R., Overbeek R., Parrello B., Pusch G.D., Shukla M., Vonstein V., Warren A., Xia F., Yoo H., Stevens R.L. Improvements to PATRIC, the all-bacterial bioinformatics database and analysis resource center. Nucleic Acids Research, 2017, 45(D1): D535-D542 (doi: 10.1093/nar/gkw1017).
- Frye T.M., Williams S.N., Graham T.W. Vitamin deficiencies in cattle. Veterinary Clinics of North America: Food Animal Practice, 1991, 7(1): 217‐275 (doi: 10.1016/s0749-0720(15)30817-3).
- Szafranska A.E., Hitchman T.S., Cox R.J., Crosby J., Simpson T.J. Kinetic and mechanistic analysis of the malonyl CoA:ACP transacylase from Streptomyces coelicolor indicates a single catalytically competent serine nucleophile at the active site. Biochemistry, 2002, 41(5): 1421-1427 (doi: 10.1021/bi012001p).
- Cronan J.E., Thomas J. Bacterial fatty acid synthesis and its relationships with polyketide synthetic pathways. Methods in Enzymology, 2009, 459: 395‐433 (doi: 10.1016/S0076-6879(09)04617-5).
- Wang H., Cronan J.E. Functional replacement of the FabA and FabB proteins of Escherichia coli fatty acid synthesis by Enterococcus faecalis FabZ and FabF homologues. Journal of Biological Chemistry, 2004, 279(33): 34489‐34495 (doi: 10.1074/jbc.M403874200).
- Yu X., Liu T., Zhu F., Khosla C. In vitro reconstitution and steady-state analysis of the fatty acid synthase from Escherichia coli. Proceedings of the National Academy of Sciences, 2011, 108(46): 18643-18648 (doi: 10.1073/pnas.1110852108).
- Johnson S.L., Daligault H.E., Davenport K.W., Jaissle J., Frey K.G., Ladner J. T., Broomall S.M., Bishop-Lilly K.A., Bruce D.C., Gibbons H.S., Coyne S.R., Lo C.-C., Meincke L., Munk A.C., Koroleva G.I., Rosenzweig C.N., Palacios G.F., Redden C.L., Minogue T.D., Chain. P.S. Complete genome sequences for 35 biothreat assay-relevant Bacillus species. Genome Announcements, 2015, 3(2): e00151-15 (doi: 10.1128/genomeA.00151-15).
- Kovanda L., Zhang W., Wei X., Luo J., Wu X., Atwill E.R., Vaessen S., Li X., Liu Y. In vitro antimicrobial activities of organic acids and their derivatives on several species of gram-negative and gram-positive bacteria. Molecules, 2019, 24(20): 3770 (doi: 10.3390/molecules24203770).
- Huang C.B., Alimova Y., Myers T.M., Ebersole J.L. Short- and medium-chain fatty acids exhibit antimicrobial activity for oral microorganisms. Archives of Oral Biology, 2011, 56(7): 650‐654 (doi: 10.1016/j.archoralbio.2011.01.011).
- Prabhadevi V., Sahaya S.S., Johnson M., Venkatramani B., Janakiraman N. Phytochemical studies on Allamanda cathartica L. using GC-MS. Asian Pacific Journal of Tropical Biomedicine, 2012, 2(2): S550-S554 (doi: 10.1016/S2221-1691(12)60272-X).
- Rahman M.M., Ahmad S.H., Mohamed M.T., Ab Rahman M.Z. Antimicrobial compounds from leaf extracts of Jatropha curcas, Psidium guajava, and Andrographis paniculata. Scientific World Journal, 2014, 2014: 635240 (doi: 10.1155/2014/635240).
- Umezawa S., Shibahara S., Omoto S., Takeuchi T., Umezawa H. Studies on the biosynthesis of 3-amino-3-deoxy-D-glucose. The Journal of Antibiotics, 1968, 21(8): 485-491 (doi: 10.7164/antibiotics.21.485).
- Milner J.L., Silo-Suh L., Lee J.C., He H., Clardy J., Handelsman J. Production of kanosamine by Bacillus cereus UW85. Applied and Environmental Microbiology, 1996, 62(8): 3061-3065.
- Kevany B.M., Rasko D.A., Thomas M.G. Characterization of the complete zwittermicin A biosynthesis gene cluster from Bacillus cereus. Applied and Environmental Microbiology, 2009, 75(4): 1144-1155 (doi: 10.1128/AEM.02518-08).
- Vetter N.D., Langill D.M., Anjum S., Boisvert-Martel J., Jagdhane R.C., Omene E., Zheng H., van Straaten K.E., Asiamah I., Krol E.S., Sanders D.A., Palmer D.R. A previously unrecognized kanosamine biosynthesis pathway in Bacillus subtilis. Journal of the American Chemical Society, 2013, 135(16): 5970-5973 (doi: 10.1021/ja4010255).
- Arakawa K., Müller R., Mahmud T., Yu T.W., Floss H.G. Characterization of the early stage aminoshikimate pathway in the formation of 3-amino-5-hydroxybenzoic acid: the RifN protein specifically converts kanosamine into kanosamine 6-phosphate. Journal of the American Chemical Society, 2002, 124(36): 10644-10645 (doi: 10.1021/ja0206339).
- Kim W.-G., Song N.-K., Yoo I.-D. Trienomycin G, a new inhibitor of nitric oxide production in microglia cells, from Streptomyces sp. 91614. The Journal of Antibiotics, 2002, 55(2): 204-207 (doi: 10.7164/antibiotics.55.204).
- Kang Q., Shen Y., Bai L. Biosynthesis of 3,5-AHBA-derived natural products. Natural Product Reports, 2012, 29: 243-263 (doi: 10.1039/C2NP00019A).
- Wrona I.E., Agouridas V., Panek J.S. Design and synthesis of ansamycin antibiotics. Comptes Rendus Chimie, 2008, 11: 1483-1522 (doi: 10.1016/j.crci.2008.07.003).
- Yu T.W., Bai L., Clade D., Hoffmann D., Toelzer S., Trinh K.Q., Xu J., Moss S.J., Leistner E., Floss H.G. The biosynthetic gene cluster of the maytansinoid antitumor agent ansamitocin from Actinosynnema pretiosum. Proceedings of the National Academy of Sciences, 2002, 99(12): 7968‐7973 (doi: 10.1073/pnas.092697199).
- Khatri I., Sharma G., Subramanian S. Composite genome sequence of Bacillus clausii, a probiotic commercially available as Enterogermina®, and insights into its probiotic properties. BMC Microbiology, 2019, 19(1): 307 (doi: 10.1186/s12866-019-1680-7).
- Al-Thubiani A.S.A., Maher Y.A., Fathi A., Abourehab M.A.S., Alarjah M., Khan M.S.A., Al-Ghamdi S.B. Identification and characterization of a novel antimicrobial peptide compound produced by Bacillus megaterium strain isolated from oral microflora. Saudi Pharmaceutical Journal, 2018, 26(8): 1089‐1097 (doi: 10.1016/j.jsps.2018.05.019).
- Malanicheva I.A., Kozlov D.G., Sumarukova I.G., Efremenkova O.V., Zenkova V.A., Katrukha G.S., Reznikova M.I., Tarasova O.D., Sineokiĭ S.P., Él'-Registan G.I. Antimicrobial activity of Bacillus megaterium strains. Microbiology, 2012, 81: 178-185 (doi: 10.1134/S0026261712020063).