doi: 10.15389/agrobiology.2019.4.705rus
УДК 636.4.033:636.082.12:575.113
При выполнении исследований использовалось оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ВИЖ им. академика Л.К. Эрнста. Исследования выполнены при поддержке Минобрнауки России, уникальный номер проекта RFMEFI60417X0182.
ИЗУЧЕНИЕ ГЕНЕТИЧЕСКОЙ АРХИТЕКТУРЫ КОНВЕРСИИ КОРМА У ХРЯКОВ (Sus scrofa) ПОРОДЫ ДЮРОК НА ОСНОВЕ ПОЛНОГЕНОМНОГО АНАЛИЗА SNP
А.А. БЕЛОУС1, А.А. СЕРМЯГИН1, О.В. КОСТЮНИНА1, G. BREM2, Н.А. ЗИНОВЬЕВА1
Конверсия корма (feed conversion ratio — FCR, кг/кг), которую рассчитывают как отношение количества потребленного корма к приросту живой массы, — важнейший показатель, определяющий экономическую эффективность производства свинины. Разработка автоматизированных кормовых станций сделала возможным проведение точного индивидуального учета потребления корма при групповом содержании свиней, что стало основой для интеграции показателя FCR в программы селекционно-племенной работы. Развитие методов высокопроизводительного генотипирования по десяткам тысяч однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP) сделало возможным выявление генетических факторов, ассоциированных с хозяйственно полезными признаками животных, на уровне геномов. Проведенное ранее изучение различных популяций показало наличие в геноме свиней множественных QTLs для признака FCR, при этом участки генома, идентифицированные в разных исследованиях, лишь частично перекрывались. В настоящем сообщении мы представляем результаты полногеномного анализа в одной из российских популяций хряков породы дюрок, который показал наличие 30 SNPs, достоверно ассоциированных с показателем конверсии корма, а также позиционных и функциональных генов-кандидатов, продукты которых участвуют в регуляции пролиферации и дифференцировки различных типов клеток, в гематопоэзе и метаболизме липидов. Целью работы было выявление генетических факторов, влияющих на эффективность использования корма у хрячков породы дюрок, фенотипированных индивидуально по показателю конверсии корма и генотипированных по ~ 70 тыс. однонуклеотидных полиморфизмов на уровне генома. Исследования проводили на 715 хрячках породы дюрок, меченных электронными чипами. Потребление корма учитывали индивидуально с использованием автоматических кормовых станций MLPII («Schauer Agrotronic AG», Швейцария) и GENSTAR («Cooperl Arc Atlantique», Франция). Генотипирование выполняли с ДНК-чипом высокой плотности Porcine GGP HD (платформа GeneSeek Genomic Profiler, «Neogene», США), содержащим ~ 70 тыс. SNPs. После контроля качества для полногеномных ассоциативных исследований (GWAS) отобрано 44810 SNP. Среднесуточные приросты живой массы (ADG) в исследуемой выборке свиней составили 962,0±5,1 г/сут, конверсия корма (FCR) — 2,53±0,02 кг/кг. По результатам GWAS-анализа выявили 30 достоверно значимых SNP, локализованных на SSC2, SSC3, SSC4, SSC6, SSC7, SSC12 и SSC15 (p < 0,00001), в том числе 3 SNP — с уровнем достоверности, превышающим порог для полногеномных исследований, включая H3GA0010441 (p < 4,14×10-7), ALGA0119936 (p < 1,03×10-6) на SSC3 и ASGA0028727 (p < 1,17×10-06) на SSC6. На SSC2, SSC6 и SSC15 обнаружены блоки, содержание соответственно 10 (в области 29,0-30,9 сМ, сборка генома Sscrofa10.2), 7 (79,1-80,3 сМ) и 3 SNPs (69,3-70,7 сМ). Аннотация генов-кандидатов, локализованных в непосредственной близости от достоверных SNPs, выявила гены, продукты которых принимают участие в гетерогенных биологических процессах (регуляция пролиферации и дифференцировки различных типов клеток, гематопоэз, метаболизм липидов). Необходимо продолжение исследований по валидации обнаруженных ассоциаций в других популяциях свиней. Идентификация дополнительных QTLs для FCR корма расширит понимание геномной архитектуры этого важнейшего селекционного признака.
Ключевые слова: полногеномное ассоциативное исследование, конверсия корма, среднесуточный прирост, толщина шпика, хряки породы дюрок.
Увеличение продуктивности при снижении затрат кормов относится к главным целям селекции сельскохозяйственных животных (1, 2). Конверсия корма (feed conversion ratio, FCR), рассчитываемая как отношение количества потребленного корма к приросту живой массы, — важнейший показатель, определяющий экономическую эффективность производства свинины, так как затраты на корма достигают 70 % в себестоимости свинины (3, 4). Разработка автоматизированных кормовых станций сделала возможным проведение точного индивидуального учета потребления корма при групповом содержании свиней, что стало основой для интеграции показателя FCR в программы селекционно-племенной работы (5-7). Детальный анализ наследуемости конверсии корма, проведенный на трех породах свиней (8), выявил умеренную степень генетической изменчивости между отдельными породами (h2 = 0,30-0,54). Индивидуальные различия свиней по показателю конверсии корма, основанные на генетических факторах, могут служить источником для выявления ДНК маркеров с целью прогнозирования этого показателя и его использования в селекции свиней (9-13).
На основании анализа генетического сцепления микросателлитных маркеров был идентифицирован ряд QTLs для показателя конверсии корма (14). Так, в кроссбредной популяции (мэйшан × крупная белая) были выявлены QTLs для FCR на SSC11 и SSC14 (15). В F2 ресурсной популяции (белый дюрок × эрхуальская порода) с использованием 183 микросателлитов обнаружены 3 QTLs для FCR на SSC2, SSC7 и SSC9 (16). Исследование ресурсной популяции крупная белая ½ пьетрен с использованием 118 микросателлитных маркеров показало наличие QTLs для FCR на SSC7 (17). Картирование, проведенное в кроссбредной популяции свиней с использованием 88 информативных микросателлитов, выявило наличие QTL для FCR на SSC2, SSC6 и SSC7 (для периода откорма от 90 до 120 кг) и на SSC2, SSC4 и SSC14 (для периода откорма от 60 до 140 кг) (18). Однако большинство QTLs, идентифицированных на основании анализа сцепления, характеризовалось относительно низкой точностью локализации (в интервале более 20 сМ).
Повышение точности картирования QTL и идентификации соответствующих ДНК маркеров стало возможным благодаря созданию ДНК-чипа, позволяющего проводить одновременный анализ десятков тысяч SNPs (Porcine60K BeadChip, «Illumina, Inc.», США). У свиней породы дюрок было установлено наличие регионов, достоверно ассоциированных с FCR, на SSC4, SSC7, SSC8 и SSC14 (19), на SSC4 и SSC15 (20), а также на SSC12 (21). Полногеномные ассоциативные исследования (genome wide association study, GWAS), проведенные в F2 ресурсной популяции, показали присутствие QTL для FCR на SSC7 (22). У терминальных хряков Максгро описаны 12 1-Mb регионов на SSC6, SSC7, SSC9, SSC11, SSC14 и SSC15, ответственных за более 0,5 % генетической изменчивости признака (23). Таким образом, проведенные ранее исследования показали наличие в геноме свиней множественных QTLs, связанных с конверсией корма, при этом участки генома, идентифицированные в разных работах, перекрывались лишь частично. Вовлечение в исследования новых популяций свиней расширяет понимание геномной архитектуры этого важного селекционного признака.
Нами впервые на популяции свиней российской репродукции изучена генетическая обусловленность наследования комплексного селекционно и экономически значимого показателя — конверсии корма с использованием технологии микроматриц. В настоящем сообщении мы представляем результаты полногеномного ассоциативного анализа в одной из российских популяций хряков породы дюрок, фенотипированных индивидуально по показателю конверсии корма и генотипированных по ~ 70 тыс. однонуклеотидных полиморфизмов на полногеномном уровне. В результате выявлены 30 SNPs, достоверно ассоциированные с показателем конверсии корма, а также позиционные и функциональные гены-кандидаты, продукты которых участвуют в регуляции пролиферации и дифференцировки различных типов клеток, в гематопоэзе и метаболизме липидов.
Целью нашей работы было изучение генетических факторов, влияющих на эффективность использования корма у хрячков породы дюрок.
Методика.Исследования проводили на базе ООО «СГЦ» (Воронежская область, п. Верхняя Хава) в период с октября 2017 по ноябрь 2018 года на 715 хряках породы дюрок, меченных электронными чипами. У всех животных отбирали пробы ткани (ушной выщип), которые консервировали 96 % спиртом и хранили при -20 °С. Средний возраст животных на начало и окончание эксперимента составил соответственно 77,6±0,3 и 156,2±0,4 сут. Хрячки содержались в станках с щелевыми полами группами по 15 гол. (площадь пола 1,30 м2/гол.) при температуре 18 °С. Животные имели неограниченный доступ к кормам и воде. Индивидуальный учет потребления корма осуществляли с использованием автоматических кормовых станций MLPII-RAP («Schauer Agrotronic AG», Швейцария) и GENSTAR («Cooperl Arc Atlantique», Франция).
Величину FCR рассчитывали для каждого животного как отношение потребленного корма к приросту живой массы за весь период выращивания. Значения по параметрам начальной и конечной живой массы хряков, среднесуточных приростов и продолжительности тестирования на станциях проверялись на соответствие нормальному распределению (M±3σ). Учитывая различия в продолжительности периода выращивания между группами, неоднородность формирования групп животных по живой массе (как при постановке, так и при снятии с откорма), а также интенсивность приростов, для получения сопоставимых значений конверсии корма использовали уравнение множественной линейной регрессии показателя FCR, рассчитанное в программе STATISTICA 10 («StatSoft, Inc.», США):
FCR(r) = 4,6738 - 0,0158x1 - 0,0170x2 + 0,0183x3 - 0,0024x4,
где FCR(r) — регрессионное значение конверсии корма; x1 — продолжительность периода откорма на станции; x2 — живая масса в начале откорма; x3 — живая масса в конце откорма; x4 — величина среднесуточного прироста живой массы за период; 4,6738 — свободный член уравнения, константная величина.
Для выделения ДНК из проб ткани свиней использовали набор ДНК-Экстран 2 (ООО «НПФ Синтол», Россия) в соответствии с рекомендациями производителя. Концентрацию двухцепочечной ДНК определяли с помощью флуориметра Qubit 2.0 («Invitrogen/Life Technologies», США). Для определения качества ДНК измеряли соотношение OD260/OD280 (cпектрофотометр NanoDrop8000, «ThermoFisher Scientific», США). Для анализа использовали ДНК с OD260/OD280 = 1,6-1,8. Кроме того, качество ДНК оценивали посредством гель-электрофореза в 1 % агарозном геле.
Полногеномное генотипирование проводили с ДНК-чипом высокой плотности Porcine GGP HD (платформа GeneSeek Genomic Profiler, «Neogene», США), содержащим ~ 70 тыс. SNPs. Контроль качества и фильтрацию данных генотипирования для каждого SNP и каждого образца выполняли с программным пакетом PLINK 1.9 (http://zzz.bwh.har-vard.edu/plink/), применяя следующие фильтры (в скобках даны соответствующие команды в программе PLINK): качество генотипирования по всем исследуемым SNPs для индивидуума не ниже 90 % (--mind); качество генотипирования для каждого из исследованных SNPs по всем индивидуумам не ниже 90 % (--geno); частота встречаемости минорных аллелей (MAF) 0,03 (--maf); отклонение SNP генотипов от распределения Харди-Вайнберга в совокупности протестированных образцов с достоверностью p-value < 10-6 (--hwe). После контроля качества для GWAS-анализа отобрали 44810 SNPs.
Для выявления ассоциаций SNP маркеров с показателем FCR применяли регрессионный анализ в PLINK 1.90 (--assoc --adjust --qt-means). Для подтверждения достоверного влияния SNP и определения значимых регионов в геноме свиней использовали тест для проверки нулевых гипотез по Бонферрони при пороговом значении p < 1,12×10-6, 0,05/44810. Данные визуализировали в пакете qqman с помощью языка программирования R (24). Для поиска генов-кандидатов, локализованных в области идентифицированных SNP, использовали геномный ресурс Sscrofa10.2 (https://www.ncbi.nlm.nih.gov/assembly/GCF_000003025.5/, дата обращения 02.04.2019). Функциональные аннотации генов выполняли с привлечением базы данных GeneCards (http://www.genecards.org/, дата обращения 02.04.2019). Расчет средних арифметических (M) и стандартных ошибок средних (±SEM), коэффициента вариации (Cv, %) осуществляли в MS Excel 2013.
Результаты. Начальная и конечная живая масса хрячков, используемых в исследованиях, составила соответственно 35,3±0,2 и 110,5±0,5 кг при среднесуточных приростах за контрольный период 962,0±5,1 г, средняя конверсия корма — 2,53±0,02 кг/кг (табл. 1).
В результате проведенного GWAS-анализа мы идентифицировали 30 SNPs на SSC2, SSC3, SSC4, SSC6, SSC7, SSC12 и SSC15, достоверно ассоциированных с показателем FCR (p < 0,00001), при этом на SSC2, SSC6 и SSC15 были обнаружены блоки из 3-10 SNPs. Для трех SNPs (H3GA0010441, ALGA0119936 на SSC3 и ASGA0028727 на SSC6) достоверность превышала порог для полногеномных исследований (p < 1,2×10-6) (рис., табл. 2).
Наиболее интересные позиционные и функциональные гены-кан-дидаты, идентифицированные в непосредственной близости от значимых SNPs, включают гены ABTB2, CAPRIN1 (локализованы на SSC3 внутри блока из 10 SNP-кандидатов в области 29,0-30,9 сМ), RBAK (локализован на SSC3 в непосредственной близости (-10825) от полногеномного SNP ALGA0119936), PTPRU, MECR, MED18, PHACTR4 и RCC1 (локализованы на SSC6 внутри блока из 7 SNP-кандидатов в области 79,1-80,3 сМ). Продукт ABTB2 обладает активностью гетеродимеризации белков, продукт CAPRIN1 участвует в регуляции пролиферации и миграции различных типов клеток.Белок, кодируемый LMO2, играет центральную роль в развитии гематопоэза, кодируемый RBAK — взаимодействует с андрогеновым рецептором, влияющим на пролиферацию и дифференцировку клеток, а также фактором транскрипции E2F, играющим решающую роль в контроле клеточного цикла. PTPRU является сигнальной молекулой, регулирующей широкий спектр клеточных процессов, включая рост, дифференцировку, митотический цикл. Среди известных функций MECR — участие в метаболизме и биосинтезе жирных кислот. Продукт MED18 — один из компонентов медиаторного комплекса, вовлечен в регуляцию транскрипции, также установлено его участие в метаболизме липидов.Функции белка, кодируемого PHACTR4, включают участие в пролиферации клеток нервной системы, а также взаимодействие с a-актином скелетных мышц, вовлеченных в моторику у разных типов клеток. У человека установлена связь полиморфизмов в генах CAPRIN1, LMO2, MECR, PTPRU, PHACTR4 и RCC1 с систолическим давлением, в LMO2, PTPRU и ABTB2 — с концентрацией протеинов в крови, в CAPRIN1, LMO2, MED18, PHACTR4 иRBAK — с эритроцитарными характеристиками. Известно, что уровень систолического давления ассоциирован с интенсивностью роста в ювенальный период (25). В свою очередь, установлена связь гематологических параметров с интенсивностью роста свиней (26, 27), коррелирующей, как известно, с конверсией корма (5). Показана связь содержания протеинов (гликопротеина и гаптоглобина) в крови свиней с интенсивностью роста и эффективностью использования корма (28).
Таким образом, проведенные нами полногеномные исследования в популяции хряков породы дюрок выявили наличие 30 SNPs, достоверно ассоциированных с показателем конверсии корма, которые локализованы на SSC2, SSC3, SSC4, SSC6, SSC7, SSC12 и SSC15 (p < 0,00001), при этом на SSC2, SSC6 и SSC15 обнаружены блоки из 3-10 SNPs. Анализ геномных регионов в области локализации достоверных SNPs показал наличие нескольких позиционных и функциональных генов-кандидатов, продукты которых принимают участие в регуляции пролиферации и дифференцировки различных типов клеток, гематопоэзе, метаболизме липидов. Для валидации обнаруженных ассоциаций в других популяциях свиней необходимы дополнительные исследования. Идентификация QTLs по признаку конверсии корма расширяет наше понимание геномной архитектуры этого важнейшего селекционного признака.
1. Характеристика фенотипических показателей в исследованной выборке хряков (Sus scrofa) породы дюрок (n = 715, ООО «СГЦ», Воронежская обл., п. Верхняя Хава, октябрь 2017-ноябрь 2018 года) |
||||
Показатель |
M±SEM |
Min |
Max |
CV, % |
Начальная живая масса, кг |
35,3±0,2 |
19,9 |
53,0 |
15,7 |
Конечная живая масса, кг |
107,3±0,5 |
75,0 |
157,0 |
10,9 |
ADG, г/сут |
962,0±5,1 |
547,5 |
1507,7 |
14,0 |
ADFI, г/сут |
2411±15 |
1318 |
3762 |
16,4 |
FCR, кг/кг |
2,53±0,02 |
1,33 |
4,03 |
16,2 |
Примечание. ADG, г/сут — среднесуточный прирост; ADFI, г/сут — среднесуточное потребление корма; FCR, кг/кг — конверсия корма; M — среднее значение; SEM — ошибка среднего; Min — минимальное значение, Max — максимальное значение; CV, % – коэффициент вариации (отношение среднеквадратического отклонения σ случайной величины к ее ожидаемому значению). |
||||
2. Достоверно значимые (p < 0,00001) SNPs, ассоциированные с конверсией корма у хряков породы дюрок, и потенциальные гены-кандидаты (n = 715, ООО «СГЦ», Воронежская обл., п. Верхняя Хава, октябрь 2017-ноябрь 2018 года) |
|||||
SSC |
Число SNP |
SNP |
Позиция |
p |
Ген-кандидат (позиции) |
2 |
10 |
INRA0059415 |
28977392 |
2,57×10-6 |
ABTB2 28,946,563 ... 29,149,547; NAT10 29,153,541 ... 29,194,433; CAPRIN1 29,199,142 ... 29,242,779; LMO2 29,401,270 ... 29,422,801; |
1 |
ALGA0015465 |
124437922 |
3,73×10-6 |
CCDC112 124,337,488 ... 124,341,509; TICAM2 124,610,898 ... 124,613,028; TMED7 124,682,078 ... 124,691,898 |
|
3 |
1 |
H3GA0008543 |
7379529 |
2,24×10-6 |
LAMTOR4 7,350,308 ... 7,402,741 |
1 |
WU_10.2_3_8562889 |
8562889 |
4,56×10-6 |
COL26A1 8,559,750 ... 8,703,022; MYL10 8,726,761 ... 8,738,595 |
|
1 |
H3GA0010441а |
116920902 |
4,14×10-7 |
CLIP4 116,931,705 ... 117,021,159 |
|
2 |
WU_10.2_3_144118805 ALGA0119936а |
144118805 |
1,83×10-61,03×10-6 |
RBAK 144,137,154... 144,154,400 |
|
4 |
1 |
WU_10.2_4_140678150 |
140678150 |
4,40×10-6 |
LMO4-201 141,341,709… 141,354,291 |
6 |
7 |
WU_10.2_6_79124865 |
79124865 |
7,22×10-6 |
MED18 79,123,55…79,156,756; PHACTR4 79,256,482…79,343,715; RCC1 79,351,068…79,378,340; TRNAU1AP 79,381,689…79,406,923; RAB42 79,417,316…79,419,319; TAF12 79,432,412…79,441,136; YTHDF2 79,553,445…79,583,689; OPRD1 79,611,950…79,662,042; EPB41 79,809,404…79,916,611; TMEM200B 79,916,247…79,920,674; SRSF4 79,952,072…79,979,921; MECR 79,986,968…80,018,140; |
7 |
1 |
H3GA0022804 |
109049333 |
7,98×10-6 |
DIO2 109,299,323 … 109,308,576 |
12 |
1 |
WU_10.2_12_15902684 |
15902684 |
6,47×10-6 |
MARCH10 15,861,982…15,944,352 |
15 |
1 |
MARC0002947 |
35674169 |
3,69×10-6 |
PTPN18 35,772,241...35,792,658 |
1 |
DRGA0015116 |
50414290 |
2,54×10-6 |
TENM3 50,292,659...50,995,047 |
|
3 |
H3GA0044471 |
69276645 |
1,02×10-5 |
KCNJ3 69,329,216...69,481,280; NR4A2 70,671,034 ... 70,689,181; GPD2 70,841,147...70,946,575 |
|
Примечание. а — SNP с уровнем достоверности, превышающим порог для полногеномных ассоциаций. Указаны позиции в сборке генома Sscrofa10.2 (https://www.ncbi.nlm.nih.gov/assembly/GCF_000003025.5/). |
|||||
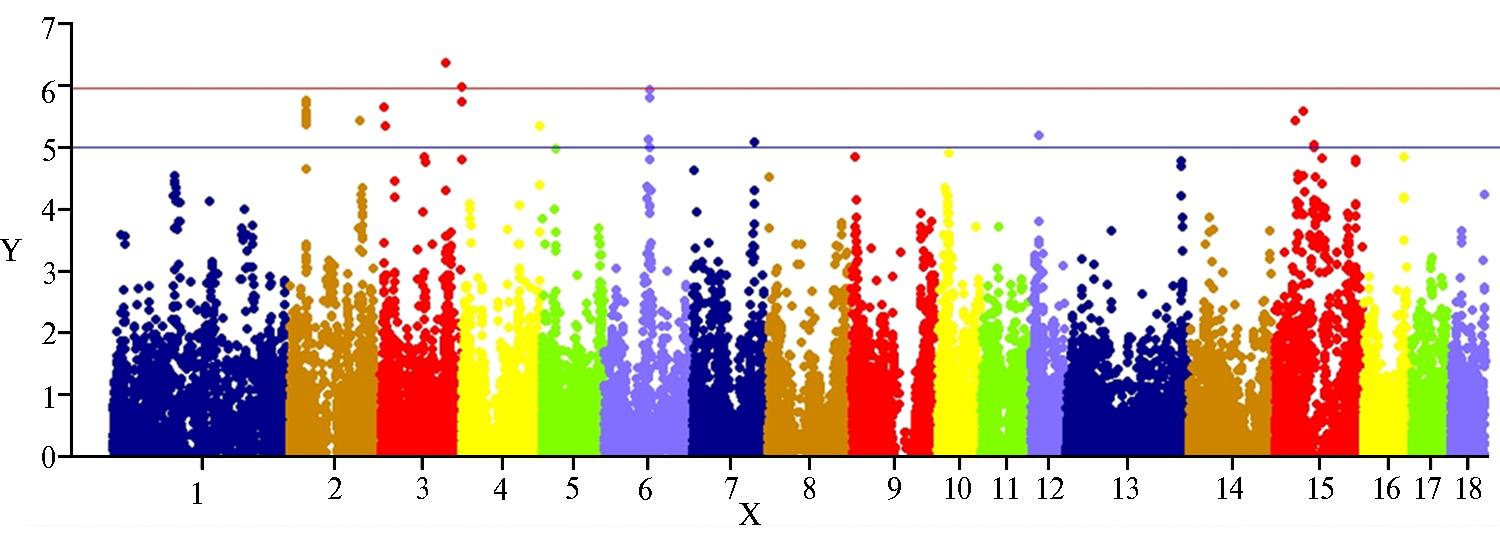
Результаты GWAS-анализа для показателя конверсии корма в исследованной выборке хряков (Sus scrofa) породы дюрок: ось Х — номер хромосомы свиней; ось Y – обращенный десятичный логарифм уровня достоверности -log10(p); верхняя горизонтальная линия — порог достоверности для полногеномных ассоциаций, -log10(p) = 1,2×10-6; нижняя горизонтальная линия — порог достоверности для суггестивных ассоциаций -log10(p) = 1,02×10-5 ) (n = 715, ООО «СГЦ», Воронежская обл., п. Верхняя Хава, октябрь 2017-ноябрь 2018 года).
ЛИТЕРАТУРА
- Kanis E., De Greef K.H., Hiemstra A., van Arendonk J.A. Breeding for societally important traits in pigs. J. Anim. Sci., 2005, 83(4): 948-957 (doi: 10.2527/2005.834948x).
- Ito T., Fukawa K., Kamikawa M., Nikaidou S., Taniguchi M., Arakawa A., Tanaka G., Mikawa S., Furukawa T., Hirose K. Effects of correcting missing daily feed intake values on the genetic parameters and estimated breeding values for feeding traits in pigs. Animal Science Journal, 2018, 89: 12-20 (doi: 10.1111/asj.12891).
- Hoque M.A., Suzuki K. Genetic parameters for production traits and measures of residual feed intake in Duroc and Landrace pigs. Animal Science Journal,2008, 79: 543-549 (doi: 10.1111/j.1740-0929.2008.00562.x).
- Hoque M.A., Suzuki K., Kadowaki H., Shibata T., Oikawa T. Genetic parameters for feed efficiency traits and their relationships with growth and carcass traits in Duroc pigs. Journal of Animal Breeding and Genetics, 2007,124(3): 108-116 (doi: 10.1111/j.1439-0388.2007.00650.x).
- Maselyne J., Saeys W., Van Nuffel A. Quantifying animal feeding behaviour with a focus on pigs. Physiol. Behav., 2015, 138: 37-51 (doi: 10.1016/j.physbeh.2014.09.012).
- Schulze V., Roehe R., Looft H., Kalm E. Effects of continuous and periodic feeding by electronic feeders on accuracy of measuring feed intake information and their genetic association with growth performances. Journal of Animal Breeding and Genetics, 2001, 118: 403-416 (doi: 10.1046/j.1439-0388.2001.00158.x).
- Lorenzo Bermejo J., Roehe R., Schulze V., Looft H., Kalm E. Genetic change of feed intake curves in growing pigs using non-linear two-stage genetic analysis and linear random regression models. Journal of Animal Breeding and Genetics, 2003, 120: 217-227 (doi: 10.1046/j.1439-0388.2003.00396.x).
- Do D.N., Strathe A.B., Jensen J., Mark T., Kadarmideen H.N. Genetic parameters for different measures of feed efficiency and related traits in boars of three pig breeds. J. Anim. Sci., 2013, 91(9): 4069-4079 (doi: 10.2527/jas.2012-6197).
- Brown-Brandl T., Rohrer G., Eigenberg R. Analysis of feeding behavior of group housed growing-finishing pigs. Computers and electronics in agriculture, 2013, 96(1): 246-252 (doi: 10.1016/j.compag.2013.06.002).
- Белоус А.А., Сермягин А.А., Костюнина О.В., Требунских Е.А., Зиновьева Н.А. Генетические и паратипические факторы, характеризующие эффективность использования корма у свиней породы дюрок. Сельскохозяйственная биология, 2018, 53(4): 712-722 (doi: 10.15389/agrobiology.2018.4.712rus).
- Chen Y., Piper E., Zhang Y., Tier B., Graser H.U., Luxford B.G., Moran C. A single nucleotide polymorphism in suppressor of cytokine signalling-2 is associated with growth and feed conversion efficiency in pigs. Animal Genetics, 2011, 42(2): 219-221 (doi: 10.1111/j.1365-2052.2010.02107.x).
- Duthie C., Simm G., Doeschl-Wilson A., Kalm E., Knap P.W., Roehe R. Quantitative trait loci for chemical body composition traits in pigs and their positional associations with body tissues, growth and feed intake. Animal Genetics, 2008, 39(2): 130-140 (doi: 10.1111/j.1365-2052.2007.01689.x).
- Oliveira Peixoto J., Facioni Guimaraes S.E., Savio Lopes P., Menck Soares M.A., Vieira Pires A., Gualberto Barbosa M.V., Almeida Torres R., Almeida e Silva M. Associations of leptin gene polymorphisms with production traits in pigs. Journal of Animal Breeding and Genetics, 2006, 123: 378-383 (doi: 10.1111/j.1439-0388.2006.00611.x).
- PigQTLDataBase. Интернет-ресурс: http://www.animalgenome.org/cgi-bin/QTLdb/SS/index. Дата обращения 11.03.2019.
- Houston R.D., Haley C.S., Archibald A.L., Rance K.A. A QTL affecting daily feed intake maps to Chromosome 2 in pigs. Mamm. Genome, 2005, 16: 464-470 (doi: 10.1007/s00335-004-4026-0).
- Zhang Z.Y., Ren J., Ren D.R., Ma J.W., Guo Y.M., Huang L.S. Mapping quantitative trait loci for feed consumption and feeding behaviors in a White Duroc × Chinese Erhualian resource population. J. Anim. Sci., 2009, 87(11): 3458-3463 (doi: 10.2527/jas.2008-1694).
- Gilbert H., Riquet J., Gruand J., Billon Y. Detecting QTL for feed intake traits and other performance traits in growing pigs in a Piétrain—Large White backcross. Animal, 2010, 4(8): 1308-1318 (doi: 10.1017/S1751731110000339).
- Shirali M., Duthie C.-A., Doeschl-Wilson A., Knap P.W., Kanis E., van Arendonk J.A., Roehe R. Novel insight into the genomic architecture of feed and nitrogen efficiency measured by residual energy intake and nitrogen excretion in growing pigs. BMC Genetics, 2013, 14: Article number 121 (doi: 10.1186/1471-2156-14-121).
- Sahana G., Kadlecová V., Hornshøj H., Nielsen B., Christensen O.F. A genome-wide association scan in pig identifies novel regions associated with feed efficiency trait. J. Anim. Sci., 2013, 91: 1041-1050 (doi: 10.2527/jas.2012-5643).
- Wang K., Liu D., Hernandez-Sanchez J., Chen J., Liu C., Wu Z., Fang M., Li N. Genome wide association analysis reveals new production trait genes in a male Duroc population. PLoS ONE, 2015, 10: e0139207 (doi: 10.1371/journal.pone.0139207).
- Ding R., Yang M., Wang X., Quan J., Zhuang Z., Zhou S., Li S., Xu Z., Zheng E., Cai G., Liu D., Huang W., Yang J., Wu Z. Genetic architecture of feeding behavior and feed efficiency in a Duroc pig population. Front. Genet., 2018, 9: 220 (doi: 10.3389/fgene.2018.00220).
- Guo Y.M., Zhang Z.Y., Ma J.W., Ai H.S., Ren J., Huang L.S. A genomewide association study of feed efficiency and feeding behaviors at two fattening stages in a White Duroc ½ Erhualian F population. J. Anim. Sci., 2015, 93(4): 1481-1489 (doi: 10.2527/jas.2014-8655).
- Reyer H., Shirali M., Ponsuksili S., Murani E., Varley P.F., Jensen J., Wimmers K. Exploring the genetics of feed efficiency and feeding behaviour traits in a pig line highly selected for performance characteristics. Molecular Genetics and Genomics: MGG, 2017, 292(5): 1001-1011 (doi: 10.1007/s00438-017-1325-1).
- Turner S.D. qqman: an R package for visualizing GWAS results using Q-Q and Manhattan plots. Journal of Open Source Software, 2018, 3(25): 731 (doi: 10.21105/joss.00731).
- Kagura J., Adair L.S., Munthali R.J., Pettifor J.M., Norris S.A. association between early life growth and blood pressure trajectories in Black South African children. Hypertension, 2016, 68(5): 1123-1131 (doi: 10.1161/HYPERTENSIONAHA.116.08046).
- Bhattarai S., Nielsen J.P. Association between hematological status at weaning and weight gain post-weaning in piglets. Livestock Science, 2015, 182: 64-68 (doi: 10.1016/j.livsci.2015.10.017).
- Serem J.K., Wahome R.G., Gakuya D.W., Kiama S.G., Gitao G.C., Onyango D.W. Growth performance, feed conversion efficiency and blood characteristics of growing pigs fed on different levels of Moringa oleifera leaf meal. Journal of Veterinary Medicine and Animal Health, 2017, 9(11): 327-333 (doi: 10.5897/JVMAH2017.0570).
- Clapperton M., Bishop S.C., Cameron N.D., Glass E.J. Associations of acute phase protein levels with growth performance and with selection for growth performance in Large White pigs. Animal Science, 2005, 81(2): 213-220 (doi: 10.1079/ASC50180213).