УДК 636.2+599.735.52]:575
ГЕНОМНОЕ РАСПРЕДЕЛЕНИЕ ISSR-МАРКЕРОВ (AG)9C И (GA)9C У ВИДОВ BOVINAE И CAPRINAE
В.И. ГЛАЗКО
Выполняли сравнительный анализ спектров продуктов амплификации участков геномной ДНК, фланкированных нуклеотидными последовательностями (AG)9C и (GA)9C, у представителей Bovinae (крупный рогатый скот, зубр, бизон) и Caprinae (породы домашней овцы, снежный баран). Получены данные о присутствии в геномах исследованных видов участков ДНК, высококонсервативных по длине не только в пределах видов одного подсемейства, но и между представителями Bovinae и Caprinae. Обсуждается возможная связь такой консервативности с принадлежностью микросателлитных локусов (AG)9C и (GA)9C к динуклеотидным пурин-пиримидиновым участкам геномной ДНК.
Ключевые слова: полиморфизм, инвертированные повторы ДНК, микросателлитные локусы, пурин-пиримидиновые последовательности.
Применение молекулярно-генетических методов для решения задач общей и частной популяционной генетики зависит от изученности размаха и особенностей полиморфизма молекулярно-генетических маркеров элементов геномов (нуклеотидных позиций в кодонах структурных генов, собственно структурных генов, некодирующих последовательностей в различных вариантах повторов), который, как известно, может существенно различаться. Пример — контрастные по уровню полиморфизма гены, кодирующие гистоны или иммуноглобулины, некоторые микросателлитные локусы с разницей по частоте мутирования более чем в 100 раз (1). Из этого следует, что популяционно-генетическая оценка близости или удаленности генетических структур исследуемых групп организмов в значительной степени зависит от особенностей полиморфизма элементов, используемых в качестве молекулярно-генетических маркеров.
В последние годы широкое распространение в подобных ис-следованиях получила оценка степени полиморфизма так называемых фрагментов «анонимной» ДНК — участков ДНК, фланкированных инвертированными повторами декануклеотидов (метод RAPD — random amplified polymorphic DNA) или микросателлитных локусов (метод ISSR — inter-simple sequence repeat) (2). Привлекательность этих маркеров связана с тем, что они дают возможность перейти от традиционного изучения полиморфизма единичных локусов (структурных генов, микросателлитных локусов) к анализу полилокусных спектров, отражающих полиморфизм множественных геномных участков, по сути — к геномным сравнениям. Применение микросателлитных локусов в качестве праймеров для выявления полиморфизма различных участков геномной ДНК основано на том, что метод ISSR, маркирующий такой полиморфизм, позволяет получать многолокусные и высокополиморфные спектры фрагментов генома. Удобство получения многолокусных спектров (несмотря на определенную зависимость числа выявляемых локусов от условий полимеразной цепной реакции) (3), а также невозможность отличить гетерозиготу от гомозиготы при наличии ампликона, маркирующего отдельный геномный локус, привело к широкому использованию этого метода в популяционной генетике. Относительно повышенная степень полиморфизма ISSR-маркеров может быть обусловлена высокой частотой мутирования микросателлитных локусов: принято считать, что по таким локусам она в среднем в 1000 раз выше, чем по структурным генам, а сами микросателлитные локусы достаточно равномерно распределены по геному (4). В то же время накапливаются свидетельства того, что скорость мутирования у этих локусов существенно варьирует и тесно связана с коровым мотивом, а также с локализацией по длине хромосомы (5). Специфика полилокусных спектров ISSR-маркеров и их полиморфизма может зависеть от нуклеотидных последовательностей флангов подобных анонимных последовательностей ДНК.
Для оценки такой возможности мы выполнили сравнительный анализ продуктов амплификации анонимных последовательностей ДНК, фланкированных инвертированными повторами с участками пурин-пиримиди-новых последовательностей (GA)9C и (AG)9C, у доместицированных и диких видов Bovinae (крупный рогатый скот, зубр, бизон) и Caprinae (породы домашней овцы, снежный баран). Пурин-пиримидиновые нуклеотидные мотивы привлекают особое внимание, поскольку показана их локализация в промоторах многих генов и экспериментально обоснована возможность участия в регуляции генной экспрессии (6). Кроме того, динуклеотиды AG формируют каноническую интрон-экзонную границу (7).
Методика. Исследовали образцы крови у животных серой украинской, якутской (пос. Черга, Алтайский край), белоголовой украинской (хозяйство «Антонины», Хмельницкая обл., Украина) пород крупного рогатого скота (КРС), американского бизона, воспроизводящегося в биосферном заповеднике «Аскания-Нова» (Украина) (образцы крови и органов из авторской коллекции Н.И. Ясинецкой), зубров (Беловежская Пуща, Белоруссия) (предоставлены Т.П. Сипко), а также у кулундинской овцы (пос. Черга, Алтайский край), асканийского многоплодного каракуля (пос. Маркеево, Херсонская обл., Украина) (по 10 гол.) и снежного барана (5 гол., предоставлены Т.П. Сипко).
При оценке полиморфизма участков ДНК, фланкированных инвертированными повторами микросателлитных локусов (2), в качестве праймеров использовали последовательности (GA)9C и (AG)9C. Геномную ДНК выделяли из лимфоцитов периферической крови по стандартной методике (2). Полимеразную цепную реакцию (ПЦР — PCR) проводили в следующих условиях: первоначальная денатурация — 2 мин при 95 °С; денатурация — 30 с при 95 °С, отжиг — 30 с при 55 °С, элонгация — 2 мин при 72 °С (37 циклов); завершающая элонгация — 7 мин при 72 °С. Продукты амплификации (ампликоны) разделяли в 2 % агарозном геле (маркер молекулярных масс — фрагменты ДНК GeneRuler™ 100 bp DNA Ladder Plus, «MBI Fermentas», США) и окрашивали бромистым этидием с визуализацией в УФ-свете.
Результаты. Анализ спектров ампликонов по участкам между инвертированными повторами микросателлитных локусов позволил выявить следующие межвидовые различия. На электрофореграммах суммарно идентифицировалось по 22 фрагмента ДНК для праймеров (GA)9C и (AG)9C (табл. 1, рис., А) длиной 0,4-2,5 т.п.н. (считали, что каждый ампликон соответствует отдельному локусу). В анализ включали только те ампликоны (условно обозначенные как мажорные), которые надежно воспроизводились и легко типировались в спектрах. У всех трех видов исследованных животных отмечались общие продукты амплификации размером 1,0; 1,4; 1,7 и 2,1 т.п.н. при использовании праймера (AG)9C. Видоспецифичные фрагменты для КРС и зубров в этом спектре отсутствовали, но наблюдались
1. Длина фрагментов ДНК (ампликонов) у крупного рогатого скота (КРС), зубра (З) и бизона (Б), полученных в ПЦР при использовании праймеров (AG)9C и (GA)9C |
||||||
Длина ампликона, т.п.н. |
Праймер (AG)9C |
Праймер (GA)9C |
||||
КРС |
З |
Б |
КРС |
З |
Б |
|
0,4 |
- |
- |
- |
- |
- |
- |
П р и м е ч а н и е. «+» и «-» — соответственно присутствие и отсутствие фрагмента ДНК определенной длины в спектре продуктов амплификации. |
||||||
Распределение спектров ампликонов в варианте с (GA)9C изменялось. Так, наиболее широким обладали представители КРС (8 фрагментов от 0,5 до 2,5 т.п.н.), причем ампликоны размером 0,7; 1,2 и 2,5 т.п.н. обнаружили только у этой группы животных, а фрагменты длиной около 0,5 и 1,1 т.п.н. были общими для особей всех изученных видов. У зубра и бизона не выявлялись видоспецифичные ампликоны. У КРС и зубра имелось два общих ампликона (0,6 и 1,6 т.п.н), у КРС и бизона — один (1,5 т.п.н.) (см. табл. 1).
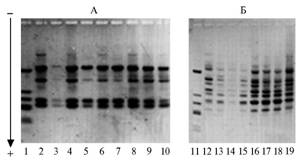 |
Электрофорeтические спектры продуктов амплификации ДНК у крупного рогатого скота (А) и домашней овцы (Б) при использовании соответственно праймера (GА)9C и праймера (AG)9C: дорожки 1 и 11 — маркер молекулярных масс; 2-4 — серая украинская порода; 5-7 — белоголовая украинская порода; 8-10 — якутская порода, 12-15 — асканийский многоплодный каракуль; 16-19 — кулундинская овца. |
То есть при оценке межвидовая дифференциация существенно варьи-ровала в зависимости от используемого праймера и длины ампликона. Если с (AG)9C видоспецифичные ампликоны обнаружили у бизона, то с (GA)9C — у КРС. Очевидно, что полилокусность маркеров ISSR-PCR удобна и, по-видимому, незаменима при межгеномных сравнениях.
При сопоставлении генетических структур, суммарно полученных при использовании праймеров (GA)9C и (AG)9C, по распределению продуктов амплификации у животных серой украинской породы, зубра и бизона (каждую зону в порядке увеличения молекулярной массы обозначали как один локус, отсутствие фрагмента учитывали как гомозиготу по рецессивному аллелю) наиболее близким к КРС оказался зубр.
У представителей пород домашней овцы, асканийского многоплодного каракуля, кулундинской овцы и снежного барана на электрофореграммах продуктов амплификации выявлялось по пять мажорных амликонов с размером продуктов 0,5-1,9 т.п.н. в зависимости от используемого праймера.
С последовательностью (AG)9C (табл. 2, см. рис., Б) спектры продуктов амплификации у двух пород овец полностью совпадали; общими между спектрами ампликонов домашней овцы и снежного барана были фрагменты ДНК 0,5; 0,6; 1,0 и 1,7 т.п.н. Только у особей дикого вида в спектре выявили ампликон длиной 1,3 т.п.н., в то же время фрагмент 1,1 т.п.н., характерный для видов домашней овцы, отсутствовал (см. табл. 2).
| 2. Длина фрагментов ДНК (ампликонов) у асканийского многоплодного каракуля (АМК), кулундинской овцы (КО) и снежного барана (СБ), полученных в ПЦР при использовании праймеров (AG)9C и (GA)9C | ||||||
Длина ампликона, т.п.н. |
Праймер (AG)9C |
Праймер (GA)9C |
||||
АМК |
КО |
СБ |
АМК |
КО |
СБ |
|
0,4 |
- |
- |
- |
- |
- |
- |
П р и м е ч а н и е. То же, что в таблице 1. |
||||||
При использовании последовательности (GA)9C в качестве праймера длина ампликонов составляла 0,5-1,9 т.п.н. У обоих видов исследованных животных наблюдались общие продукты амплификации размером 0,5; 1,5 и 1,9 т.п.н.; фрагменты 1,6 и 1,7 т.п.н. имелись только в спектрах у домашней овцы, фрагмент 1,4 т.п.н. — у снежного барана. Внутривидовых различий между породами овец при использовании указанного праймера не обнаружили. Иными словами, межвидовая генетическая дифференциация по ISSR-PCR маркерам у представителей семейства Caprinae также существенно варьировала в зависимости от используемого праймера.
Следует подчеркнуть высокую точность отжига в эксперименте: последовательность (AG)9C можно было бы рассматривать как последовательность (GA)8GC, (GA)9C — (AG)8AC. Но при очевидном сходстве праймеров (в том числе одинаковой принадлежности корового динуклеотида к пуринам) получаемые спектры продуктов амплификации существенно отличались друг от друга (см. табл. 1, 2). В то же время присутствие в спектрах мажорных ампликонов одинаковой длины, фланкированных инвертированными повторами (AG)9C и (GA)9C, у видов одного и того же подсемейства (Bovinae, Caprinae), а также у видов из разных подсемейств оставалось высококонсервативным. Так, при использовании праймера (AG)9C имелось два ампликона длиной 1,0 и 1,7 т.п.н., общих для представителей видов Bovinae и Caprinae,(GA)9C — один фрагмент ДНК длиной 0,5 т.п.н., общий для особей всех исследованных видов.
Выполненное сравнение спектров ампликонов с использованием в качестве праймеров фрагментов микросателлитных локусов (AG)9C и (GA)9C свидетельствует, что в ряде случаев длина фрагментов ДНК, фланкированных ими, высококонсервативна и сохраняется у видов не только одного, но даже разных подсемейств. Следовательно, несмотря на высокую вариабельность, определенное постоянство нуклеотидных последовательностей и распределения инвертированных повторов (AG)9C и (GA)9C сохраняется в геномах видов, принадлежащих к разным подсемействам. Такой консерватизм хорошо согласуется с представлениями А. Лима-де-Фариа о неслучайном распределении семейств тандемных повторов по длине хромосом и об участии некоторых из них в структурно-функциональной организации линейных хромосом эукариотов даже на таких небольших участках ДНК (2 т.п.н.), которые используют в популяционных исследованиях в качестве ISSR-маркеров геномного полиморфизма.
Неоднородность структуры эукариотической хромосомы известна достаточно давно. В основе формирования линейной хромосомы лежит образование двух основных морфологических структур — центромерного и теломерных участков. Теломерные районы у большинства видов животных, как известно, формируются с участием фермента теломеразы, обладающего активностью РНК-зависимой ДНК-полимеразы с РНК-затравкой для амплификации теломерных повторов (гексануклеотид TTAGGG). У некоторых видов насекомых, в частности у дрозофилы, теломерные повторы образуются за счет репликации ретротранспозонов (8).
Структура центромерного участка у многих видов включает семейства транспозонов и ретротранспозонов (9, 10). Кроме того, белки, участвующие в упаковке центромерной области и позволяющие кинетохору формироваться здесь, имеют выраженную гомологию с транспозазой ряда ДНК-транспозонов (11, 12). Накопленные данные дают основание считать, что эти две основные морфологические структуры хромосомы эукариотов возникли при участии транспозирующихся элементов, задающих определенные линейные координаты хромосомы. Предположение о наличии таких координат впервые было сформулировано в работах А. Лима-де-Фариа на основании наблюдений о неслучайности чередования гетерохроматиновых блоков по длине хромосом у ряда видов растений. Он высказал гипотезу о «хромосомных полях» — нуклеотидных последовательностях и скоплении различных семейств повторов, включая центромерные и теломерные, с которыми непосредственно связана морфология хромосом (13).
В последние годы накоплено достаточно подтверждений тесной связи между молекулярной структурой наследственного материала и морфологией хромосом — тем, что А. Лима-де-Фариа называл «хромосомным фенотипом». В частности, показано неслучайное распределение семейств ретротранспозонов по длине хромосом у Arabidopsis (14) и ряда видов грибов (15), а также семейств ретротранспозонов в центромерных участках хромосом у некоторых видов растений, например кукурузы (16), и локализация ретротранспозонов в теломерных районах. Подобная неоднородность позволяет ожидать, что полиморфизм разных геномных элементов, которые широко используются для изучения генетической структуры популяций у животных и растений, может зависеть от распределения таких элементов относительно линейных координат хромосом.
Таким образом, несмотря на определенное сходство между нукле-отидными мотивами (AG)9C и (GA)9C, при использовании их в качестве праймеров в полимеразной цепной реакции (ПЦР) с геномной ДНК одних и тех же животных образуются разные спектры продуктов амплификации ДНК. Это свидетельствует о высокой точности отжига и надежности идентификации геномных фрагментов, фланкированных указанными последовательностями. Существует определенная зависимость состава фрагментов в спектрах от условий ПЦР, однако в них выделяются мажорные фрагменты, которые надежно воспроизводятся в разных условиях. Длина большинства таких ампликонов консервативна не только у разных видов в пределах рода и подсемейства, но также у представителей разных подсемейств. Выявленный консерватизм свидетельствует о неслучайности геномного распределения инвертированных повторов (AG)9C и (GA)9C, что, по-видимому, связано с их принадлежностью к пурин-пиримидиновым динуклеотидным мотивам.
Л И Т Е Р А Т У Р А
1. T h u i l l e t A.-C., B r u D., D a v i d J. e.a. Direct estimation of mutation rate for 10 microsatellite loci in durum wheat, Triticum turgidum (L.) Thell. ssp. durum desf. Mol. Biol. Evol., 2002, l9(1): 122-125.
2. Z i e t k i e w i c z E., R a f a l s k i A., L a b u d a D. Genome fingerprinting by sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 1994, 20: 176-183.
3. W i e s n e r I., W i e s n e r o v a D. Insertion of a reamplification round into the ISSR-PCR protocol gives new flax fingerprinting patterns. Cell Mol. Biol. Letters, 2003, 8: 743-748.
4. T o t h G., G a s p a r i Z., J u r k a J. Microsatellites in different eukaryotic genomes: survey and analysis. Genome Res., 2000, 10: 417-432.
5. K a l i s h J.M., S e i d m a n M.M., W e k s D.L. e.a. Triplex-induced recombination and repair in the pyrimidine motif. Nucl. Acids Res., 2005, 33(11): 3492-3502.
6. Y a g i l G. The over-representation of binary DNA tracts in seven sequenced chromosomes. BMC Genomics, 2004, 5: 19-37.
7. S h e t h N., R o c a X., H a s t i n g s M.L. e.a. Comprehensive splice-site analysis using comparative genomics. Nucl. Acids Res., 2006, 34(14): 3955-3967.
8. W a l t e r M.F., B i e s s m a n n M.R., B e n i t e z C. e.a. Effects of telomere length in Drosophila melanogaster on life span, fecundity, and fertility. Chromosoma, 2007, 116: 41-51.
9. J i n W., L a m b J.C., V e g a J.M. e.a. Molecular and functional dissection of the maize B chromosome centromere. The Plant Cell, 2005, 17: 1412-1423.
10. N a s u d a S., H u d a k o v a S., S c h u b e r t I. e.a. Stable barley chromosomes without centromeric repeats. PNAS, 2005, 102: 9842-9847.
11. C a s o l a C., H u c k s D., F e s c h o t t e C. Convergent domestication of pogo-like transposases into centromere-binding proteins in fission yeast and mammals. Mol. Biol. Evol., 2008, 25(1): 29-41.
12. Z h a n g W., L e e H y e - R a n, K o o D a l - H o e e.a. Epigenetic modification of centromeric chromatin: hypomethylation of DNA sequences in the CENH3-associated chromatin in Arabidopsis thaliana and maize. The Plant Cell, 2008, 20: 25-34.
13. L i m a - de - F a r i a А. The chromosome field theory confirmed by DNA and hybridization. Riv. Biol., 1987, 80: 266-268.
14. K e n d a l W.S., S u o m e l a D.P. Large-scale genomic correlations in Arabidopsis thaliana relate to chromosomal structure. BMC Genomics, 2005, 6: 82-89.
15. T h o n M.R., P a n H., D i e n e r S. e.a. The role of transposable element clusters in genome evolution and loss of synteny in the rice blast fungus Magnaporthe oryzae. Genome Biol., 2006, 7(2): 1-9 (Article R16, http://genomebiology.com/2006/7/2/R16).
16. J i n W., L a m b J.C., V e g a J.M. e.a. Molecular and functional dissection of the maize B chromosome centromere. The Plant Cell, 2005, 17: 1412-1423.
GENOMIC DISTRIBUTION OF ISSR MARKERS OF (AG)9C AND (GA)9C IN BOVINAE AND CAPRINAE SPECIES
V.I. Glazko
The author made a comparative analysis of spectrums of amplification products of genomic DNA parts, flanking by nucleotide sequences of (AG)9C and (GA)9C, in Bovinae (cattle, auroch, bison) and Caprinae (domestic seep breeds, bighorn sheep) species. The data was obtained about the presence in genomes of investigated species of high conservative DNA region both within species of one subspecies and between individuals of Bovinae and Caprinae. The possible correlation of such conservatism was discussed in connection with a belonging of microsatellite loci of (AG)9C and (GA)9C to dinucleotide purine-pyrimidine regions of genomic DNA.
Key words: polymorphism, DNA inverted repeats, microsatellite locuses, purine/pyrimidine sequenses.
ФГОУ ВПО Российский государственный аграрный университет РГАУ—МСХА им. К.А. Тимирязева, |
Поступила в редакцию |













