doi: 10.15389/agrobiology.2019.3.469rus
УДК 635.127:577.152.31:577.151.64:577.21
QTL КАРТИРОВАНИЕ ИЗОФЕРМЕНТНЫХ ФОРМ ЭСТЕРАЗ ЗРЕЛЫХ СЕМЯН У Brassica rapa L. (обзор)
А.С. РУДАКОВА1, С.В. РУДАКОВ1, А.М. АРТЕМЬЕВА2, Д.А. ФАТЕЕВ2, Н.В. КОЧЕРИНА3, Ю.В. ЧЕСНОКОВ3
С 1960-х годов изоферменты хорошо известны как одни из наиболее распространенных биохимических маркеров. Установление общей изменчивости изоферментных систем и выявление их генетического контроля сохраняют актуальность, позволяя вскрывать тонкие механизмы взаимоотношения организма с окружающей средой и гомеостаза и разрабатывать эффективные биохимические маркеры для экспресс-оценки генетически и селекционно значимого материала. В представленной работе нами впервые идентифицированы и картированы локусы хромосом, отвечающие за проявление активности 13 различных изоформ эстераз зрелых семян у Brassica rapa L. В исследовании использовали линии удвоенных гаплоидов двух картирующих популяций — DH30 и DH38. Все выявленные изоформы эстераз по электрофоретической подвижности подразделялись на три группы. Группа изоформ А1-А3 обладала большой молекулярной массой и малой электрофоретической подвижностью, группа изоформ В1-В7 характеризовалась средней молекулярной массой и проявляла среднюю электрофоретическую подвижность, группу С1-С3 составляли изоформы с малой молекулярной массой и, соответственно, наибольшей электрофоретической подвижностью. Каждая из родительских форм (как и каждая из исследованный линий картирующих популяций) имела уникальный электрофоретический спектр изоформ эстераз. На основании полученных электрофоретических данных для обеих популяций был проведен QTL-анализ и установлены локусы хромосом, определяющие проявление каждой изоформы эстеразы, выявленной у линий картирующих популяций DH30 и DH38. Использованный подход множественного интервального QTL-картирования (composite interval mapping) в сочетании с тестом пермутации (1000 повторений) при уровне статистической значимости p < 0,05 позволил идентифицировать и локализовать на хромосомах локусы (QTL, quantitative trait loci), определяющие проявление всех изоформ эстераз, идентифицированных методом гель-электрофореза. Исключение составили изоформы А1 для популяции DH38 и изоформы В7 для популяции DH30, для которых мы не получили результатов QTL-анализа из-за малого объема исходных данных по этим изоформам у соответствующей картирующей популяции. Всего картировали 35 QTL изоформ эстераз в случае картирующей популяции DH30 и 39 QTL для популяции DH38. В результате проведенного QTL-анализа выявили молекулярные маркеры, генетически сцепленные с идентифицированными локусами, и рассчитали процент фенотипической изменчивости, определяемый каждым из выявленных QTL. По данным изоферментного анализа был проведен расчет гетерозиготности для обеих популяций в каждом локусе Hl и общей гетерозиготности Hобщ., а также дисперсии гетерозиготности Var(Hl) по одному локусу и дисперсии средней гетерозиготности внутри каждой из популяций Var(Hобщ.). Выявленная гетерозиготность рассматривалась как средняя порция локусов с двумя различными аллелями в одном локусе у одной особи и могла быть определена как наблюдаемая гетерозиготность, характеризующая часть генов, по которым изучаемая популяция гетерозиготна. Показано, что в изученных популяциях линий удвоенных гаплоидов QTL, детерминирующие комплекс изоферментов эстераз, находятся в основном во 2-й, 4-й, 6-й и 9-й группах сцепления и формируют блоки коадаптированных генов и геномные коадаптированные блоки генов, что подчеркивает важность вклада этих локусов в онтогенез и адаптивность растений B. rapa. Выполненное молекулярно-генетическое картирование в сочетании с биохимическим анализом спектров изоформ эстераз зрелых семян у B. rapa позволили обнаружить генетические детерминанты, определяющие проявление изученных признаков, а также распределение картированных QTL по геному, что в перспективе дает возможность проводить эффективный молекулярно-генетический скрининг образцов коллекции и селекционного материала у вида B. rapa по изученным биохимическим признакам при генетико-селекционных исследованиях.
Ключевые слова: Brassica rapa L., биохимический анализ, эстеразы, изоформы, зрелые семена, картирование, QTL, гетерозиготность популяций.
С 1960-х годов изоферменты хорошо известны как одни из наиболее распространенных биохимических маркеров, пригодных для установления генетической изменчивости как в естественных, так и искусственных селекционно значимых популяциях растений (1-3) и животных (4-6). Установление общей изменчивости изоферментных систем и выявление их генетического контроля остается актуальным, поскольку исследования проблемы адаптации на биохимическом уровне позволяют не только вскрывать тонкие механизмы взаимоотношения организма с окружающей средой и сохранения гомеостаза в процессе роста и развития как в нормальных, так и экстремальных условиях (7), но и разрабатывать эффективные биохимические маркеры для экспресс-оценки генетически и селекционно значимого материала (8).
Эстеразы (ЕС 3.1.1.) объединяют большое число различных карбоксилэфирных гидролаз. Некоторые из них имеют очень широкую субстратную специфичность и способны гидролизовать как эндогенные, так и экзогенные эфирные связи различной структуры (9). Тот факт, что ферменты, которые показывают эстеразную активность, могут также гидролизовать и неэфирные связи, поднимает вопрос об эволюционной физиологической и адаптивно-экологической роли этих эстераз. Так, возможность ингибирования α- и β-эстераз в растениях была описана для арилэстераз в молодых листьях маниоки и оценена как маркер патогенеза после инфицирования бактериями (10). Тесты по ингибированию показали, что органофосфаты (инсектициды) ингибируют одни и активируют другие растительные эстеразы в Aspidosperma polyneuron (11). Следовательно, растительные эстеразы могут служить биохимическими индикаторами, используемыми для обнаружения инсектицидных остатков и определения их токсичности при контроле загрязнения окружающей среды. В целом эстеразная система организмов характеризуется присутствием большого ряда изоферментов и значительной индивидуальной и популяционной вариабельностью. У животных большинство эстеразных ферментов неспецифичны и демонстрируют перекрывающуюся субстратную специфичность (3). В некоторых случаях эти изоферменты относительно специфичны, как, например, холинэстеразы и карбонильные ангидразы.
Функциональная характеристика растительных эстераз пока недостаточно исследована, хотя у растений эстеразы, по-видимому, представляют одну из наиболее изучаемых групп изоферментов. Еще в конце 1960-х—начале 1970-х годов проводились исследования эстераз у кукурузы (1, 12, 13), ячменя (2), картофеля (14), хлопка (15, 16), овса (17, 18), сахарной свеклы (19), пшеницы (20, 21) и многих других видов. Наиболее подробно генетический контроль эстераз описан у пшеницы (21-25). Кроме того, определена «чужеродно»-пшеничная гомеология хромосом (4, 20) и идентифицированы «чужеродно»-пшеничные гибриды (26, 27), а также выявлена изменчивость эстераз у гексаплоидных генотипов (28). Все эти работы были направлены на установление не столько физиологической, сколько генетической составляющей такой биохимической системы, как эстеразы высших растений. Отсутствие эпистатических взаимодействий и кодоминантный характер наследования изоформ эстераз делают их удобным инструментом для быстрого и эффективного изучения процессов биохимической адаптации к изменяющимся условиям окружающей среды. Такой тип биохимических маркеров удобен и для решения практических задач селекции в качестве средства, способного ускорить и упростить процесс отбора селекционно значимого материала.
В наших предшествующих исследованиях выявлен полиморфизм изоферментного профиля эстераз, выделенных из зрелых семян, у образцов сортового и линейного селекционного материала гексаплоидной мягкой пшеницы (Triticum aestivum L.), при этом средняя гетерозиготность (H) образцов по 10 выявленным локусам, кодирующим изоформы эстераз, составила 0,924 (29). Были выделены наиболее перспективные родительские формы, различающиеся по фенотипическим признакам селекционного интереса и по спектрам изоформ эстераз, — сорта Злата, Мера и гомозиготные линии АФИ91, АФИ177, ITMI7, ITMI44, ITMI83 и ITMI115 и установлена возможность определения полиморфизма эстераз в гибридных поколениях. В другом нашем исследовании (30) показан широкий спектр разнообразия электрофоретических профилей эстеразных изоферментов зрелых семян (восемь изоформ с молекулярными массами от 37,7 до 57,6 кД) у 25 образцов редиса (Raphanus sativus L.), которые были разделены на 13 электрофоретических зимотипов, отличающихся друг от друга наличием или отсутствием определенных зон. Наиболее распространенным был электрофоретический зимотип Гр. 1, который имели 24 % оцененных образцов. Зимотип Гр. 5 характеризовался максимальным числом зон — 8, зимотипы Гр. 3 и Гр. 12 — наименьшим (по 4). Мономорфными оказались седьмая и восьмая зоны эстераз (соответственно Мr = 39,7 кД и Мr = 37,7 кД), остальные шесть зон были полиморфными, при этом частота каждой зоны для различных зимотипов варьировала от 6,58 до 17,11 %. Полученные результаты позволили выделить перспективный исходный материал для селекции.
Однако до настоящего времени не было проведено ни одного исследования линий удвоенных гаплоидов по изоферментному спектру эстераз. Преимущество такого подхода заключается в том, что, например, молекулярно-генетические карты картирующих популяций Brassicarapa L. DH30 и DH38 насыщены SSR и AFLP молекулярными маркерами с интервалом картирования 2,4-2,6 сМ. Чем плотнее расположены маркеры, тем точнее карта. AFLP маркеры в настоящее время относительно редко, но все же используются в комплексе с маркерами других типов. В то же время в отсутствие других известных маркеров изоформ эстераз в перспективе для рутинного скрининга селекционного или иного генетического материала можно конвертировать AFLP маркеры, расположенные в пиках выявленных QTL, в SCAR или CAPS маркеры. Все это позволяет снять вопрос о надежности и воспроизводимости результатов при использовании маркеров, полученных с использованием определенного генетического материала, поскольку такой вопрос возникает очень часто, если не всегда (29-31). Различное положение локусов количественных признаков селекционного интереса в упомянутых популяциях DH30 и DH38 отмечено во многих исследованиях, что совершенно естественно, так как генетически популяции различны (31). Но тем это интереснее, поскольку для массового коллекционного или селекционного скрининга наиболее надежны маркеры локусов, находящихся в близких позициях в обеих популяциях.
В настоящей работе мы впервые провели изоферментный анализ у линий удвоенных гаплоидов Brassica rapa L., что позволило не только идентифицировать локализацию локусов, определяющих проявление анализируемых изоформ эстераз, в группах сцепления на хромосомах, но и найти статистические взаимосвязи между выявленными QTL (quantitative trait loci) этих изоферментов и наблюдаемой гетерозиготностью для каждого локуса.
Целью работы было картирование QTL, определяющих проявление различных изоформ эстераз зрелых семян у линий удвоенных гаплоидов Brassica rapa L.
Методика. Эстеразные профили семян анализировали у 80 линий картирующих популяций Brassica rapa L. (3 родительские формы, 50 линий потомства DH38 и 27 линий потомства DH30). Подробности получения и использования популяций DH30 и DH38 для картирования QTL морфологических и биохимических признаков у B. rapa описаны нами ранее (31).
Зрелые семена тщательно размалывали в фарфоровой ступке, по 100 мг полученной муки помещали в пробирки типа эппендорф, добавляли по 1 мл гексана, тщательно перемешивали и оставляли для обезжиривания на ночь в холодильнике при 4 °С, затем центрифугировали 10 мин при 15 тыс. об/мин, надосадочную жидкость сливали и оставляли пробы под тягой для высушивания на воздухе. Ферменты экстрагировали из обезжиренных и высушенных образцов 0,05 М Трис-HCl буфером при pH 8,3 в присутствии 2-меркаптоэтанола (2 мкл/мл) и глицерина (10 %). Соотношение мука:буфер составляло 1:4, продолжительность экстракции — 14-18 ч при температуре в интервале от 4 до 8 °С. Образцы центрифугировали 10 мин, отбирали надосадочную жидкость и замораживали пробы при -20 °С для предотвращения инактивации изоферментов при хранении. Перед внесением в камеру для электрофореза образцы размораживали при комнатной температуре. Изоферменты разделяли методом нативного вертикального электрофореза в PAAG (32), концентрация разделяющего и концентрирующего гелей — соответственно 11 и 5 %. Электрофорез осуществляли в камере Мini-PROTEAN Tetra Cell («Bio-Rad Laboratories, Inc.», США). В качестве маркеров молекулярных масс использовали Prestained Protein Ladder («Thermo Scientific», США). В каждый карман концентрирующего геля вносили по 20 мкг белка. Предварительно концентрацию белка в ферментных препаратах определяли методом связывания красителя по M.M. Bradford (33), в качестве стандарта использовали бычий сывороточный альбумин («Promega Corporation», США). Электрофорез проводили на холоде (10-15 °С), при 10 B/см в течение 2,5 ч. По окончании разделения гель обрабатывали реактивом на неспецифическую эстеразу (34). Для этого гель выдерживали в свежеприготовленном растворе красителя, состоящем из 100 мг α-нафтилацетата и 120 мг β-нафтилацетата («Sigma-Aldrich Chemie GmbH», Швейцария), растворенных в 10 мл 70 % этанола; 500 мг Fast Blue RR («Sigma-Aldrich Co.», США); 4 мл пропанола и 60 мл 0,1 М фосфатного буфера рН 6,0. После появления коричневато-фиолето-вых полос избыток красителя отмывали 10 % уксусной кислотой. Полученные зимограммы сканировали (Epson Expression 10000XL, «GE Healthcare», США). Оценку каждого образца (значение Rf всех полос в треке, расчет молекулярных масс по стандартам, расчет относительного количества каждой зоны в треке) проводили с использованием программы Phoretix 1D Advansed («TotalLab, Ltd», Великобритания).
Для картирования выявленных QTL использовали компьютерную программу MAPQTL 6.0 («Kyazma B.V.», Нидерланды) (35), с помощью которой установили присутствие и расположение QTL-кандидатов в группе сцепления (интервал картирования 5 сМ), значения LOD (logarithm of odds) (p = 0,05) и степень фенотипического проявления признаков (% Expl.), которые объясняются соответствующим QTL, для каждой изоформы эстеразы в каждой популяции. Значимость LOD устанавливали в тесте пермутации (1000 повторений) (36).
Коэффициент корреляции признак—маркер рассчитывали для уровня значимости р = 0,05 как статистически достоверное сцепление маркерного локуса с QTL, определяющим изучаемый признак, на основе эмпирически полученных варианс для каждой пары признак—маркер (37). Используя данных по картированию выявленных QTL, выполняли графическое построение молекулярно-генетических карт с помощью компьютерной программы MapChart 2.2 (https://www.wur.nl/en/Research-Results/Research-Insti-tutes/plant-research/biometris/Software-Service.htm) (38). Статистическую обработку данных проводили методами многофакторного анализа (35).
Определяли степень наблюдаемой гетерозиготности H для каждого локуса, а также ее среднее значение по нескольким локусам (общая гетерозиготность Hобщ.). Гетерозиготность популяции в каждом локусе Hl и общую гетерозиготность Hобщ. рассчитывали в соответствии с описанием (39, 40) по следующим формулам:
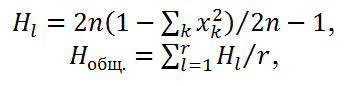
где l — порядковый номер локуса, n — размер популяции, xk — оцениваемая частота k-го аллеля l-го локуса, r — общее число локусов.
Находили дисперсии гетерозиготности Var(Hl) по одному локусу и дисперсию средней гетерозиготности внутри популяции Var(Hобщ.) (41):
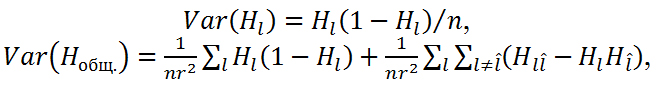
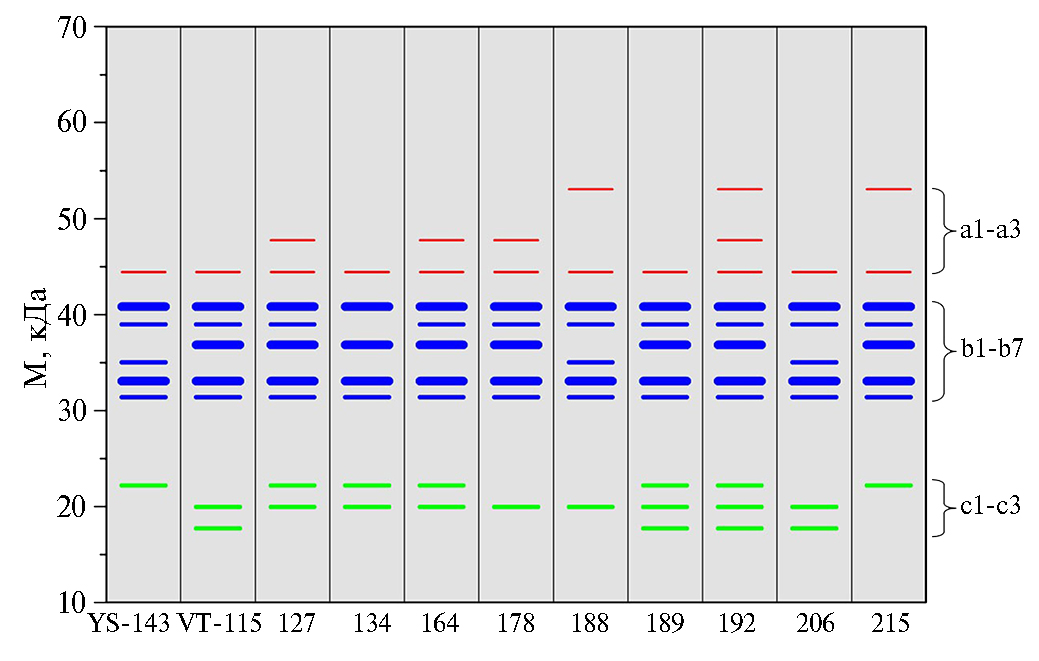
Результаты. Электрофоретический анализ изоферментов эстераз зрелых семян у линий удвоенных гаплоидов из двух картирующих популяций B. rapa и их трех родительских форм выявил 13 изоформ эстеразных ферментов, которые подразделялись на три группы по электрофоретической подвижности (рис. 1). Группа изоформ А1-А3 обладала большой молекулярной массой и малой электрофоретической подвижностью, группа В1-В7 проявляла среднюю молекулярную массу и среднюю подвижность, изоформы группы С1-С3 имели малую молекулярную массу и самую высокую электрофоретическую подвижность. Следует отметить, что интенсивность зон А1-А3 и С1-С3 варьировала относительно слабо, а В1-В7 — значительно. Кроме того, в отличие от групп А1-А3 и С1-С3, включавших по три изоформы, группа В1-В7 была представлена семью различными изоформами эстраз.
Каждая из родительских форм, как и каждая из исследованных линий картирующих популяций, обладала своим уникальным электрофоретическим спектром изоформ эстераз. Группа С1-С3 состояла из изоформ, присущих как отцовскому, так и каждому из материнских образцов. У этой группы эстераз проявлялся кодоминантный тип наследования. Группа В1-В7 у родительских форм состояла из шести электрофоретических зон, пять из которых были идентичны, а зона В3, отсутствовавшая у отцовских растений YS-143, присутствовала у обеих материнских форм — VT-115 и PC-175, проявляя доминантный тип наследования. В случае популяции DH30 зона В4 имелась у отцовской формы YS-143 и отсутствовала у материнской VT-115, также проявляя при этом доминантный тип наследования. У линии 97 картирующей популяции DH30, а также у линий 80, 123, 127 и 136 картирующей популяции DH38 обнаруживалась изоформа В7, отсутствующая у всех трех родительских форм. Появление этой изоформы у перечисленных пяти линий, по-видимому, объясняется трансгрессивным характером генетических перестроек или изменений в геноме этих линий, произошедших в процессе их создания и отбора в культуре in vitro, то есть вследствие сомаклональной изменчивости. Не исключено также, что это одна из идентифицированных ранее у Brassicaceae синапинэстераз (42). То же самое можно сказать в отношение изоформ А1 и А2, выявленных у ряда линий как в популяции DH30, так и в популяции DH38. Необходимо отметить, что изоформа А3 присутствует у всех трех родительских форм и отсутствует у линий 28 и 67 из популяции DH30, а также линий 1, 4, 67, 90, 124 и 134 из популяции DH38. Наличие изоформ А1 и А2 у различных линий обеих популяций варьировало. Их присутствие у той или иной линии, по-видимому, объясняется теми же причинами, что и появление изоформы В7.
На основании электрофореграмм для обеих популяций провели QTL-анализ и установили локусы хромосом, определяющие появление каждой изоформы эстеразы, обнаруженной у линий картирующих популяций DH30 и DH38 (табл. 1, см. на сайте журнала http://www.agrobiology.ru).
Использованный нами подход множественного интервального QTL-картирования (composite interval mapping) в сочетании с тестом пермутации (1000 повторений) при уровне значимости p < 0,05 позволил идентифицировать и локализовать на хромосомах локусы (QTL), определяющие появление всех идентифицированных гель-электрофорезом изоформ эстераз (см. рис. 1), за исключением А1 для популяции DH38 и В7 — для DH30. Для этих двух изоформ мы не получили результатов QTL-анализа из-за малого объема соответствующих исходных данных по каждой картирующей популяции.
По результатам QTL-картирования выявлены молекулярные маркеры, генетически сцепленные с идентифицированными локусами, что в перспективе может использоваться для молекулярно-генетического анализа образцов B. rapa, и установлена локализация обнаруженных QTL в группах сцепления (рис. 2, см. на сайте журнала http://www.agrobiology.ru). Так, в популяции DH30 для изоформы А1 были идентифицированы семь локусов хромосом, определяющих ее проявление. Максимальные LOD значения установили для 1-й (3,70) и 5-й (3,76) групп сцепления. Для А2 в популяции DH30 обнаружили три, а в популяции DH38 — шесть локусов, определяющих проявление этой изоформы. Необходимо отметить, что расположение QTL изоформы А2 на 4-й и 9-й хромосомах совпадало в обеих популяциях, указывая на эволюционное закрепление этого признака, имевшего, по-видимому, адаптивное значение. Максимальную величину LOD (2,09) в популяции DH30 установили для 9-й хромосомы, а для DH38 полученные LOD значения варьировали от 2,28 до 4,88 для разных групп сцепления. В противоположность этому величина LOD для изоформы А3 оказалась сравнительно низкой — 1,29 для единственного QTL в популяции DH30 и 1,30-1,79 для QTL, выявленных в DH38, однако на долю фенотипической изменчивости, которую определяли QTL, идентифицированные для изоформы А3, приходилось 23,4 % в популяции DH30 и 14,6-19,5 % в случае DH38.
Изоформа В1 тоже характеризовалась низким значением LOD для обеих популяций — 1,36-1,53 и 1,33-1,75 при доле фенотипической изменчивости 20,2-25,4 и 13,3-17,1 % соответственно для DH30 и DH38. Всего же картировали три QTL у популяции DH30 и две — у DH38. QTL изоформы В3 не были многочисленными и имели невысокие значения LOD. Так, для DH30 они составили соответственно 2,00 и 1,37-2,53, для DH38 — 4,00 и 1,32-1,78. Однако для В3, как и для А2, месторасположение QTL, картированных в 6-й группе сцепления, совпадало, что указывает на эволюционно-адаптивный характер этой изоформы. Число QTL, определяющих экспрессию изоформы В2, также было невелико — четыре для популяции DH30 и две — для DH38. Однако величина LOD (1,36-3,36 и 1,93-2,47 соответственно для DH30 и DH38) и доля контролируемой фенотипической изменчивости (соответственно 23,9-48,0 и 19,9-24,8 %) были выше, чем в случае В3. Практически такие же результаты получили для изоформы В4. Для нее выявили по два QTL в каждой из картирующих популяций. Величина LOD и доля фенотипической изменчивости при этом варьировали в пределах 1,48-1,55 и 46,1-47,8 % для DH30, 1,33-1,49 и 20,3-22,4 % — для DH38. Изоформа В5, хотя и имела невысокие LOD (1,32-1,52 и 1,93-2,07 соответственно для DH30 и DH38) при небольшом проценте фенотипической изменчивости (22,3-25,3 и 18,3-19,5 %), но, как и В3, имела QTL, локализованный у обеих популяций в 6-й группе сцепления в близких позициях, чем подтверждается наше заключение об эволюционно-адаптивном характере наследования этих изоформ эстераз семян у B. rapa. У популяции DH30 мы идентифицировали пять QTL изоформ В6 на 3-й, 6-й, 7-й и 8-й хромосомах, у DH38 — три QTL в 8-й, 9-й и 10-й группах сцепления. LOD при этом были относительно невелики — соответственно 1,31-1,84 и 1,31-1,48, доля фенотипической изменчивости — 23,9-29,7 и 11,9-14,9 % для DH30 и DH38. Интересно отметить, что для популяции DH30 выявленный в 6-й группе сцепления QTL располагался в том же месте, что и QTL изоформы В5, что свидетельствует о важности этого хромосомного локуса у B. rapa. Изоформу В7 идентифицировали и картировали только у популяции DH38. Всего локализовали пять QTL, которые располагались на 1-й, 2-й, 7-й и 9-й хромосомах. Значения LOD варьировали от 1,53 до 2,13, доля фенотипической изменчивости, обусловленной выявленными QTL, составила от 82,8 до 91,4 %.
Для С1 в каждой из картирующих популяций обнаружили всего по одному QTL. При этом значения LOD (1,39 и 1,32 для DH30 и DH38) и процент фенотипической изменчивости, обусловленной соответствующим QTL (32,9 и 18,9 %), были относительно невелики. QTL С2 идентифицировали у DH30 во 2-й, 4-й и 5-й группах сцепления (1,46-2,20 и 31,2-43,0 %), у DH38 — в 3-й и 8-й группах (1,29-2,31 и 18,2-30,7 %). У изоформы С3 картировали три QTL в популяции DH30 и четыре QTL — у DH38. При этом значения LOD идентифицированных QTL варьировали в пределах 1,50-2,77 для DH30 и 1,51-2,51 — для DH38, а доля обусловленной фенотипической изменчивости у DH30 и DH38 составила соответственно 68,5-88,1 и 43,3-65,0 %. QTL изоформы С3, картированный на 2-й хромосоме (как и в случае А2, В3 и В5), идентифицировали у обеих картирующих популяций. Этот факт подтверждает, что указанный локус 2-й хромосомы имеет эволюционное значение и его структура была закреплена в процессе онтогенетической и филогенетической адаптации у вида B. rapa. По всей вероятности, именно с такими адаптивно значимыми локусами хромосом связана стабильность характерных морфологических и биохимических признаков растения, а возможно, и повышенная адаптационная способность к неблагоприятным факторам окружающей среды (43-45).
По данным изоферментного анализа мы рассчитали гетерозиготность популяций по каждому локусу Hlи общую гетерозиготность Hобщ., а также дисперсию гетерозиготности Var(Hl) по одному локусу и дисперсию средней гетерозиготности внутри каждой из популяций Var(Hобщ.) (табл. 2).
Поскольку дисперсия средней гетерозиготности должна учитывать ковариации между гетерозиготностями по разным локусам (l и ![]() ), что обусловлено их зависимостью от частоты двойных гетерозигот
), что обусловлено их зависимостью от частоты двойных гетерозигот ![]() по этим локусам, использовали формулу, предложенную Б. Вейром (41). Выявленная гетерозиготность рассматривалась как средняя порция локусов с двумя различными аллелями в одном локусе у одной особи и могла быть определена как наблюдаемая гетерозиготность, характеризующая часть генов, по которым изучаемая популяция гетерозиготна. Использованные нами формулы (39-41) дают возможность рассчитать любой многочлен в наборе переменных, распределенных мультиноминально, что, в свою очередь, позволяет рассматривать выявляемую гетерозиготность как меру информационного полиморфизма, широко используемого при составлении и реализации генетико-селекционных программ. Формирование любых селекционно значимых свойств зависит от генотипа особи (46, 47). При этом информационный полиморфизм есть не что иное, как отражение фенотипических проявлений распределенных в геноме генетических детерминант (генов, локусов хромосом), определяющих изучаемые признаки (48).
по этим локусам, использовали формулу, предложенную Б. Вейром (41). Выявленная гетерозиготность рассматривалась как средняя порция локусов с двумя различными аллелями в одном локусе у одной особи и могла быть определена как наблюдаемая гетерозиготность, характеризующая часть генов, по которым изучаемая популяция гетерозиготна. Использованные нами формулы (39-41) дают возможность рассчитать любой многочлен в наборе переменных, распределенных мультиноминально, что, в свою очередь, позволяет рассматривать выявляемую гетерозиготность как меру информационного полиморфизма, широко используемого при составлении и реализации генетико-селекционных программ. Формирование любых селекционно значимых свойств зависит от генотипа особи (46, 47). При этом информационный полиморфизм есть не что иное, как отражение фенотипических проявлений распределенных в геноме генетических детерминант (генов, локусов хромосом), определяющих изучаемые признаки (48).
Таким образом, мы впервые картировали QTL, детерминирующие биохимические признаки — спектры изоформ эстераз зрелых семян у картирующих популяций удвоенных гаплоидов B. rapa и выявили молекулярные маркеры, генетически сцепленные с QTL этих признаков, что позволяет проводить эффективный молекулярно-генетический скрининг образцов коллекций и селекционного материала. Механизмы, которые контролируют биосинтез и накопление таких биологически активных веществ, как изоферменты, определяющие биохимическую характеристику морфотипов, складываются в результате эволюционно-адаптивного формирования и наследования. Модификация этих механизмов может приводить к резкому изменению количества и набора изоферментов в различных органах, в частности в семенах, что, в свою очередь, у B. rapa сказывается на морфологических и адаптивных качественных признаках вида. Изучение генетических детерминант изоформ эстераз в перспективе может позволить управлять процессом генетической изменчивости по этим адаптивно значимым признакам посредством объединения в одном организме различных генетических производных от разных родительских форм. Анализ генетической изменчивости очень важен для понимания генетики количественных признаков, играющих ключевую роль в селекционных программах по улучшению экономически значимых свойств у B. rapa. В итоге было бы крайне важно определить функции генов, влияющих на их проявление, молекулярно-генетические механизмы экспрессии и, наконец, охарактеризовать молекулярную изменчивость геномов внутри и за пределами вида. Интересно отметить, что схожие данные ранее нами были получены для пшеницы (29) и редиса (30), у которых также выявлен полиморфизм между образцами перспективного селекционного материала по спектрам изоферментных форм эстераз зрелых семян. Кроме того, эти результаты перекликаются с описанными при анализа эстеразно-липазного генного семейства у Arabidopsis thaliana (49) и Olea europaea (50). Данные, полученные на перечисленных видах (как в настоящем исследовании, так и ранее), в перспективе, как мы полагаем, позволят установить генетико-эволюционные и филогенетические взаимосвязи строения и функционирования геномов у одно- и двудольных растений.
Следует отметить обнаруженное нами совпадение позиций локусов, которые контролируют изоферментные профили эстераз и другие биохимические признаки, исследованные нами ранее, — содержание свободных аминокислот, фенольных соединений, каротиноидов и хлорофиллов. Эти локусы локализованы в нижней части хромосомы А03, где находится ген перехода к цветению BrFLC5, а также в середине и в нижней части группы сцепления А06 (31).
Итак, молекулярно-генетическое картирование и анализ спектров изоформ эстераз зрелых семян у Brassica rapa выявили генетические детерминанты этих признаков. Установлено распределение картированных QTL по геному. Показано, что в изученных популяциях линий удвоенных гаплоидов QTL, детерминирующие комплекс изоферментов эстераз, находятся в основном во 2-й, 4-й, 6-й и 9-й группах сцепления и формируют блоки коадаптированных генов и геномные коадаптированные блоки генов, что подчеркивает важность вклада этих локусов в онтогенез и адаптивность растений B. rapa. В целом совпадение локализации обнаруженных QTL поддерживает наблюдаемые биохимические корреляции (например, расположение изоформ эстераз на электрофореграмме), а блоки генов, объединенные в геномные районы в группах сцепления А02, А03, А04, А06 и А09 и в QTL, идентифицированные в группах сцепления, отражают генетические корреляции между такими признаками, как спектры изоформ эстераз, и иными биохимическими свойствами растений.
2. Показатели гетерозиготности и ее дисперсии у линий картирующих популяций DH30 и DH38 Brassica rapa L. по результатам изоферментного анализа эстераз |
|||||||||||||
Показатель |
Изоформа эстераз |
||||||||||||
A1 |
A2 |
A3 |
B1 |
B2 |
B3 |
B4 |
B5 |
B6 |
B7 |
C1 |
C2 |
C3 |
|
Картирующая популяция DH30 |
|||||||||||||
Hl |
0,352 |
0,616 |
0,139 |
0 |
0,139 |
0,475 |
0,491 |
0 |
0 |
0,073 |
0,475 |
0.391 |
0,391 |
Var(Hl) |
0,008 |
0,009 |
0,004 |
0 |
0,004 |
0,009 |
0,009 |
0 |
0 |
0,002 |
0,009 |
0,009 |
0,009 |
Средняя гетерозиготность Hобщ. = 0,272; дисперсия Var(Hобщ.) = 0,007 |
|||||||||||||
Картирующая популяция DH38 |
|||||||||||||
Hl |
0 |
0,213 |
0,213 |
0,078 |
0,149 |
0,424 |
0,476 |
0 |
0 |
0,149 |
0,465 |
0,465 |
0,368 |
Var(Hl) |
0 |
0,003 |
0,003 |
0,001 |
0,002 |
0,005 |
0,005 |
0 |
0 |
0,002 |
0,005 |
0,005 |
0,005 |
Средняя гетерозиготность Hобщ. = 0,231; дисперсия Var(Hобщ.) = 0,003 |
|||||||||||||
А
Б
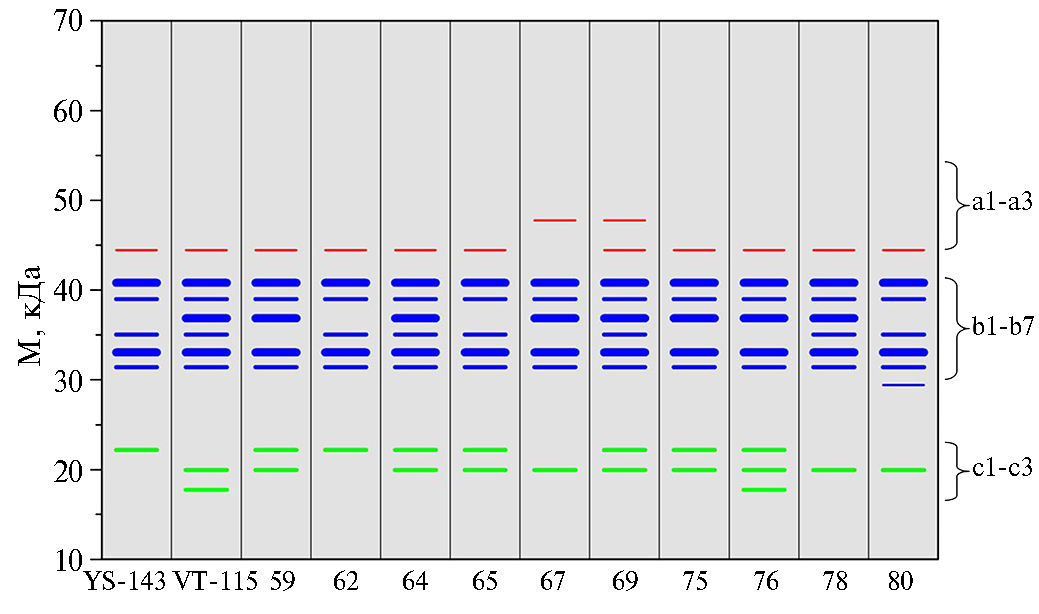
Рис. 1. Схемы электрофореграмм изоформ эстераз в картирующих популяциях DH30 (А) и DH38 (Б) Brassica rapa L.: YS-143 — отцовская форма, VT-115, PC-175 — материнские формы; цифры внизу — номера линий картирующих популяций; А1-А3, В1-В7 и С1-С3 — группы изоформ эстераз соответственно с большой, средней и малой молекулярной массой; М — маркер молекулярных масс Prestained Protein Ladder («Thermo Scientific», США).
ЛИТЕРАТУРА
- Schwartz D., Fuehsman C., McGrath H.K. Allelic isozymes of the pH 7.5 esterases in maize. Genetics, 1965, 52: 1265.
- Solari R.M., Favret E.A. Polymorphism in endosperm proteins of barley and its genetic control. In: Barley genetics II /R. Nilan (ed.). Washington University Press, Pulman WA, 1971: 23-31.
- Artemova N.V. Chromosomal control of the isoenzymes of alcohol dehydrogenase, esterase and amylase in different rye varieties. Genetika, 1982, 18: 661-667.
- Coates P.M., Mestriner M.A., Hopkinson D.A. A preliminary genetic interpretation of the esterase isozymes of human tissues. Ann. Hum. Genet., 1975, 39: 1-20 (doi: 10.1111/j.1469-1809.1975.tb00103.x).
- Lith H.A., Bieman M., Zutphen B.F.M. Purification and molecular properties of rabbit liver esterase ES-1A. Eur. J. Biochem., 1989, 184: 545-551 (doi: 10.1111/j.1432-1033.1989.tb15048.x).
- Deimilling O., Wassmer B. Genetic characterization of esterase-28 (ES-28) of the house mouse. Biochem. Genet., 1991, 29: 55-64 (doi: 10.1007/BF00578240).
- Иваченко Л.Е. Роль множественных форм ферментов сои в процессе биохимической адаптации к условиям выращивания. Успехи современного естествознания, 2010, 9: 96-98.
- Чесноков Ю.В. Молекулярно-генетические маркеры и их использование в предселекционных исследованиях. СПб, 2013.
- Walker C.H., Macknees M.I. Esterases: problems of identification and classification. Biochem. Pharmacol., 1983, 32: 3265-3269.
- Pereira A.J., Lapenta A.S., Vidigal-Filho P.S., Machado M.F.P.S. Differential esterase expression in leaves of Manihot esculenta Crantz infected by Xanthomonas axonopodis pv. manihotis. Biochem. Genet., 2001, 39: 289-296 (doi: 10.1023/A:1012251228986).
- Carvalho V.M., Marques R.M., Lapenta A.S., Machado M.F. Functional classification of esterases from leaves of Aspidosperma polyneuron M. Arg. (Apocynaceae). Genet. Mol. Biol., 2003, 26(2): 195-198 (doi: 10.1590/S1415-47572003000200013).
- MacDonald T., Brewbaker J. Isoenzyme polymorphism in flowering plants. VIII. Genetic control and dimeric nature of transaminase hybrid maize isoenzymes. J. Hered., 1972, 63: 11-14 (doi: 10.1093/oxfordjournals.jhered.a108209).
- Scandalios J.G. Genetic control of multiple molecular forms of enzymes in plants. Biochem. Genet., 1969, 3: 37-79 (doi: 10.1007/BF00485973).
- Desborough S., Peloquin J. Esterases isozymes from Solanum tubers. Phytochemistry, 1967, 6: 1398-1402 (doi: 10.1016/S0031-9422(00)86051-3).
- Cherry J.P., Katterman F.R.H., Endrizzi J.E. Comparative studies of seed proteins of species of Gossypium by gel electrophoresis. Evolution, 1970, 24: 231-447 (doi: 10.2307/2406817).
- Cherry J.P. A comparative study of seed proteins of allopolyploids of Gossypium by gel electrophoresis. Canadian Journal of Genetics and Cytology, 1971, 13: 155-158 (doi: 10.1139/g71-024).
- Marshall D.R., Allard R.W. Isozyme polymorphism in natural populations of Avena fatua and A. barbata. Heredity, 1970, 25: 273-282 (doi: 10.1038/hdy.1970.38).
- Murray B.E., Craig I.L., Rajhathy T. A protein electrophoretic study of three amphidiploids and eight species of Avena. Canadian Journal of Genetics and Cytology, 1970, 12: 651-665 (doi: 10.1139/g70-085).
- Lerch B., Wolf G. Isolation of phosphodiesterase from sugar beet leaves. Biochimica et Biophysica Acta (BBA) — Enzymology, 1972, 258: 200-218 (doi: 10.1016/0005-2744(72)90979-5).
- Barber H.N., Driscoll C.J., Vickery R.S. Enzymic markers for wheat and rye chromosomes. In: Proceedings III International Wheat Genetics Symposium. Canberra, 1968: 116-122.
- May C.E., Vickery R.S., Driscoll C.S. Gene control in hexaploid wheat. In: Proceedings 4th International Wheat Genetics Symposium. Columbia, 1973: 843-849.
- Nakai Y. Isozyme variations in Aegilops and Triticum. 2.Esterase and acid phosphatase isozymes studied by the gel isoelectrofocusing method. Seiken Ziho, 1973, 24: 45-73 (doi: 10.1038/hdy.1977.102).
- Nakai Y. Isozyme variations in Aegilops and Triticum. 3.Chromosomal basis of the esterase isozyme production in different organs of Chinese spring wheat. Bot. Mag. Tokyo, 1976, 89(3): 219-234 (doi: 10.1007/BF02488344).
- Cubadda R., Bozzini A., Quattrucci E. Genetic control of esterases in common wheat. Theor. Appl. Genet., 1975, 45: 290-293 (doi: 10.1007/BF00276681).
- Jaaska V. Electrophoretic survey of seedling esterases in wheats in relation to their phylogeny. Theor. Appl. Genet., 1980, 56: 273-284 (doi: 10.1007/BF00282570).
- Fedak G. Increased homoeologous chromosome pairing in Hordeum vulgare ½ Triticum aestivum hybrids. Nature, 1977, 266: 529-530 (doi: 10.1007/BF00264329).
- Fejer O., Belea A. Cytology and isoenzymes of some Triticum auto- and amphidiploids. Wheat Information Service, 1978, 47/48: 41-44.
- Alexandrescu N., Saulescu N. Esterase variability in kernels of Triticum aestivum cultivars. Revue roumaine de biochimie, 1982, 19: 93-100.
- Рудакова А.С., Рудаков С.В., Давыдова Н.В., Мирская Г.В., Журавлева Е.В., Чесноков Ю.В. Изоферментный анализ эстераз в зрелых семенах гексаплоидной мягкой пшеницы (Triticum aestivum L.). Сельскохозяйственная биология, 2016, (51)3: 327-334 (doi: 10.15389/agrobiology.2016.3.327rus).
- Рудакова А.С., Рудаков С.В., Артемьева А.М., Курина А.Б., Кочерина Н.В., Чесноков Ю.В. Изучение полиморфизма эстеразного состава зрелых семян образцов редиса (Raphanus sativus L.) коллекции ВИР. Овощи России, 2017, 5: 3-8 (doi: 10.18619/2072-9146-2017-5-3-8).
- Артемьева А.М., Соловьева А.Е., Кочерина Н.В., Беренсен Ф.А., Руднева Е.Н., Чесноков Ю.В. Картирование хромосомных локусов, определяющих проявление морфологических и биохимических признаков качества у культур вида Brassica rapa L. Физиология растений, 2016, 63: 275-289 (doi: 10.7868/S0015330316020044).
- Davis B.J. Disc electrophoresis. II. Method and application to human serum proteins. Annals of the NY Academy of Science, 1964, 121: 404-427 (doi: 10.1111/j.1749-6632.1964.tb14213.x).
- Bradford M.M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein dye binding. Analytical Biochemistry, 1976, 72: 248-254 (doi: 10.1016/0003-2697(76)90527-3).
- Meon S. Protein, esterase and peroxidase patterns of phytophtora isolates from Cocoa in Malaysia. Journal of Islamic Academy of Sciences, 1988, 1(2): 154-158.
- Van Ooijen J.W.MapQTL 6. Software for the mapping of quantitative trait loci in experimental populations of diploid species. Wageningen, Netherlands, 2009.
- Kocherina N.V., Artemyeva A.M., Chesnokov Yu.V. Use of LOD-score technology in mapping quantitative trait loci in plants. Russian Agricultural Sciences, 2011, 37: 201-204 (doi: 10.3103/S1068367411030098).
- Ronald P.S., Penner G.A., Brown P.D., Brule-Babel A.Identification of RAPD markers for percent hull in oat. Genome, 1997, 40: 873-878 (doi: 10.1139/g97-813).
- Voorrips R.E.MapChart: Software for the graphical presentation of linkage maps and QTLs. J. Hered., 2002, 93: 77-78 (doi: 10.1093/jhered/93.1.77).
- Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89(3): 583-590.
- Lefèvre F., Charrie A. Isozyme diversity within African Manihot germplasm. Euphytica, 1992, 66: 73-80 (doi: 10.1007/BF00023510).
- Вейр Б. Анализ генетических данных. М., 1995: 132-135.
- Clauss K., Baumert A., Nimtz M., Milkowski C., Strack D. Role of a GDSL lipase-like protein as sinapine esterase in Brassicaceae. Plant J., 2008, 53(5): 802-813 (doi: 10.1111/j.1365-313X.2007.03374.x).
- De Souza A., Hull P.A., Gille S., Pauly M. Identification and functional characterization of the distinct plant pectin esterases PAE8 and PAE9 and their deletion mutants. Planta, 2014, 240(5): 1123-1138 (doi: 10.1007/s00425-014-2139-6).
- Biely P. Microbial carbohydrate esterases deacetylating plant polysaccharides. Biotechnol. Adv., 2012, 30(6): 1575-1588 (doi: 10.1016/j.biotechadv.2012.04.010).
- Gao Z., Qian Q., Liu X., Yan M., Feng Q., Dong G., Liu J. Dwarf 88, a novel putative esterase gene affecting architecture of rice plant. Plant Molecular Biology, 2009, 71(3): 265-276 (doi: 10.1007/s11103-009-9522-x).
- Сандухадзе Б.И., Журавлева Е.В., Кочетыгов Г.В. Озимая пшеница Нечерноземья в решении продовольственной безопасности Российской Федерации. М., 2011.
- Casacuberta J.M., Jackson S., Panaud O., Purugganan M., Wendel J. Evolution of plant phenotypes, from genomes to traits. G3: Genes, Genomes, Genetics, 2016, 6(4): 775-778 (doi: 10.1534/g3.115.025502).
- Yang W., Guo Z., Huang C., Duan L., Chen G., Jiang N., Fang W., Feng H., Xie W., Lian X., Wang G., Luo Q., Zhang Q., Liu Q., Xiong L. Combining high-throughput phenotyping and genome-wide association studies to reveal natural genetic variation in rice. Nature Communications, 2014, 5: 5087 (doi: 10.1038/ncomms6087).
- Lai C.P., Huang L.M., Chen L.O., Chan M.T., Shaw J.F. Genome-wide analysis of GDSL-type esterases/lipases in Arabidopsis. Plant Mol. Biol., 2017, 95(1-2):181-197 (doi: 10.1007/s11103-017-0648-y).
- Rejón J.D., Zienkiewicz A., Rodríguez-García M.I., Castro A.J. Profiling and functional classification of esterases in olive (Olea europaea) pollen during germination. Ann. Bot., 2012, 110(5): 1035-1045 (doi: 10.1093/aob/mcs174).