doi: 10.15389/agrobiology.2022.2.237rus
УДК 579.62:[579.22+579.25
Работа выполнена в рамках Государственного задания Министерства науки и высшего образования Российской Федерации по теме № 0532-2021-0004 «Разработка методологических подходов к мониторингу, контролю и сдерживанию антибиотикорезистентности оппортунистических микроорганизмов в животноводстве».
МОЛЕКУЛЯРНЫЕ МЕХАНИЗМЫ И ГЕНЕТИЧЕСКИЕ ДЕТЕРМИНАНТЫ УСТОЙЧИВОСТИ К АНТИБАКТЕРИАЛЬНЫМ ПРЕПАРАТАМ У МИКРООРГАНИЗМОВ (обзор)
В.Д. ЗУБАРЕВА✉, О.В. СОКОЛОВА, Н.А. БЕЗБОРОДОВА, И.А. ШКУРАТОВА,А.С. КРИВОНОГОВА, М.В. БЫТОВ
Длительное бесконтрольное применение антибактериальных средств в сельском хозяйстве и животноводстве, в особенности с целью стимулировать рост животных, привело к распространению устойчивых форм микроорганизмов, что представляет серьезную проблему для общественного здравоохранения, поскольку такие бактерии могут становиться патогенными для человека, легко передаваться ему через продукцию и сырье животного происхождения или контаминацию окружающей среды отходами агропромышленных предприятий (C. Manyi-Loh с соавт., 2018; A.Н. Панин с соавт., 2017; N. Vidovic с соавт., 2020). Существует ряд механизмов, способствующих развитию устойчивости бактериальной клетки к одному или нескольким антимикробным препаратам: модификация мишени, на которую воздействует лекарственное средство; приобретение метаболических путей, альтернативных существующим (формирование метаболического шунта); избыточное производство фермента-мишени; ферментативная модификация или деградация противомикробного средства и активный эффлюкс антибиотика, то есть уменьшение накопления противомикробного препарата внутри клетки через снижение проницаемости стенки и/или активный эффлюкс антимикробного препарата из бактериальной клетки. Эти механизмы могут быть естественными для одних микроорганизмов или приобретенными — для других (M.F. Varela с соавт., 2021; W.C. Reygaert, 2018; А.Л. Бисекенова с соавт., 2015). Их понимание позволит выбрать наилучший вариант лечения для каждого конкретного инфекционного заболевания и разработать противомикробные препараты, препятствующие развитию резистентных микроорганизмов. Наиболее клинически значимые гены антибиотикорезистентности обычно расположены на разных мобильных генетических элементах (МГЭ), которые могут перемещаться внутриклеточно (между бактериальной хромосомой и плазмидами) или межклеточно (в пределах одного вида или между разными видами или родами) (C.O. Vrancianu с соавт., 2020). В основном передача генов антибиотикорезистентности происходит за счет горизонтального переноса, то есть посредством обмена МГЭ, например плазмидами или транспозонами, несущими гены антибиотикорезистентности, между видами бактерий, даже если они не относятся к близкородственным (S. Redondo-Salvo с соавт., 2020). Горизонтальный перенос генов между устойчивыми к антибиотикам бактериями и облигатной микрофлорой осуществляется с участием трех основных механизмов: конъюгации (с участием плазмиды), трансформации (с участием свободной ДНК) и трансдукции (с участием бактериофагов) (Y. He с соавт., 2020). При горизонтальном переносе трансформация между видами бактерий для передачи генов устойчивости к антибиотикам происходит редко. Конъюгация с участием мобильных генетических элементов служит наиболее эффективным и основным методом распространения антибиотикорезистентности (J.M. Bello-López с соавт., 2019). Целью настоящего обзора стало описание генов антибиотикорезистентности бактерий, персистирующих в условиях агропромышленного комплекса и характерных для микробиоты сельскохозяйственных животных, а также механизмов формирования антибактериальной устойчивости к противомикробным препаратам, применяемым в ветеринарии. Приведена непосредственная локализация некоторых генетических детерминант антибиотикорезистентности, рассмотрены основные меры борьбы с антибиотикорезистентностью, которые включают в себя сокращение использования антибиотиков вследствие улучшения качества жизни и условий содержания животных; организацию и проведение мониторинга и надзора за распространением антибиотикоустойчивых бактерий.
Ключевые слова: антибиотикорезистентность, животноводство, механизмы устойчивости, антибиотики, мобильные генетические элементы, генетические детерминанты, микроорганизмы.
Распространение устойчивых к антибиотикам бактерий — обостряющаяся проблема во всем мире (1). С открытием пенициллина в 1928 году многие опасные для жизни или даже смертельные заболевания стали излечимыми, что принесло очевидную пользу специалистам в области ветеринарии и зоотехнии. Однако с 1960-х годов антибиотики начали широко применяться в качестве стимуляторов роста для сельскохозяйственных животных (2). В 2017 году Всемирная организация здравоохранения (ВОЗ) опубликовала список бактерий, для борьбы с которыми требуется создание новых антибиотиков. По данным ВОЗ, устойчивость к противомикробным препаратам становится причиной 25 000 смертей в год в Европейском Союзе (ЕС) и 700 000 во всем мире. При существующей неблагоприятной тенденции к 2050 году антибиотикорезистентные бактерии могут стать причиной большего количества смертей, чем рак (1, 3).
Целью настоящего обзора стало описание генов антибиотикорезистентности бактерий, персистирующих в условиях агропромышленного комплекса и характерных для микробиоты сельскохозяйственных животных, а также механизмов формирования антибактериальной устойчивости к противомикробным препаратам, применяемых в ветеринарии.
Бесконтрольное использование антибиотиков приводит к накоплению низких субингибирующих концентраций в тканях и кишечнике обработанных животных и в окружающей среде, что способствует отбору антибиотикорезистентных бактерий, усиливает их рост, происходит мутационный процесс и введение мутаций de novo (4). Кроме того, присутствие антибиотиков может стимулировать образование биопленок и горизонтальный перенос генов (ГПГ) у некоторых бактерий. Например, перенос устойчивости к азитромицину, ципрофлоксацину или тигециклину наблюдался у Enterococcus faecalis и Pseudomonas aeruginosa (5).
Основная передача генов антибиотикорезистентности происходит через ГПГ, то есть посредством обмена мобильными генетическими элементами (МГЭ), такими как плазмиды или транспозоны, кодирующие гены антибиотикорезистентности, между видами бактерий, даже если они не относятся к близкородственным (6). Некоторые страны официально ограничили использование антибиотиков в животноводстве с целью применения их только в терапевтических целях (например, ЕС в 2006 году в соответствии с законодательством 1831/2003/EC). Тем не менее антибактериальные препараты все еще чрезмерно используются на территориях с высокой интенсивностью животноводства: в США, России, Индии, Китае и Южной Африке (7). В США на лечение противомикробными средствами животных, используемых для производства пищевых продуктов, приходится примерно 80 % от общего годового объема. При этом подавляющее большинство антибиотиков представляют собой основные лекарственные средства, применяемые для лечения распространенных инфекций или необходимые для проведения операций, трансплантации органов или химиотерапии у людей (8). В Российской Федерации с 1 марта 2022 года вступил в силу приказ об утверждении перечня антибактериальных препаратов, предназначенных для лечения инфекционных и паразитарных болезней животных, вызываемых патогенными и условно-патогенными микроорганизмами, в отношении которых вводятся ограничения на применение в терапевтических целях, в том числе для лечения сельскохозяйственных животных. Этот перечень разделяет антибактериальные средства на группу А (запрещенные для всех животных; запрещенные для продуктивных животных), группу В (лекарственные препараты второго выбора) и группу С (лекарственные препараты первого выбора) (9).
Существует ряд механизмов, способствующих развитию устойчиво-сти бактериальной клетки к одному или нескольким антимикробным препаратам: уменьшение накопления противомикробного препарата внутри клетки через снижение проницаемости стенки и/или активного эффлюкса (выведения) антимикробного препарата из бактериальной клетки; ферментативная модификация или деградация (инактивация) противомикробного средства; приобретение альтернативных метаболических путей к существующим (формирование метаболического шунта); модификация или защита противомикробной мишени; избыточное производство фермента-мишени (11-13).
Уменьшение накопления противомикробного препарата в бактериальной клетке посредством снижения проницаемости клеточной стенки и активного эффлюкса. Эффлюксные насосы — это локализованные в цитоплазматической мембране всех типов клеток белковые переносчики, которым для выполнения своей функции требуется источник химической энергии. Некоторые из них — первичные активные переносчики, использующие гидролиз аденозинтрифосфата как источника энергии, тогда как другие — вторичные активные переносчики (унипортеры, симпортеры или антипортеры), у которых транспорт связан с электрохимической разностью потенциалов, создаваемой выведением ионов водорода или натрия за пределы клетки (14). Изменение проницаемости, возникающее во внешних мембранах бактериальных клеток, обусловливает снижение поступления антибиотика в клетку, одновременно с этим происходит активация эффлюксных насосов, и оставшаяся часть препарата выводится за пределы клетки. Указанные механизмы резистентности коррелируют друг с другом и всегда оказывают сопряженное воздействие на лекарственные препараты (15). Проницаемость клетки подвергается изменениям посредством приобретения мутаций в поринах (белковые каналы, проходящие через клеточную мембрану). Эти мутации включают в себя потерю порина, изменение размера или проводимости поринового канала или более низкую экспрессию порина (16). Что касается эффлюксных насосов, то некоторые из них постоянно экспрессируются, опосредуя врожденную устойчивость бактерий, в то время как другие экспрессируются под воздействием индуктора. Кроме того, сверхэкспрессия эффлюксных насосов может способствовать возникновению более высокой устойчивости. Сверхэкспрессия может носить временный характер и возникать в присутствии эффектора (фенотипическая резистентность) или быть перманентной, когда отбираются мутанты по регуляторным элементам экспрессии эффлюксных насосов (приобретенная, или вторичная, резистентность) (17).
В обзорах S. Hernando-Amado с соавт. (18) и W.C. Reygaert (19) эффлюксные насосы сгруппированы в пять основных структурных суперсемейств — резистентность-нодуляция-деление (resistance-nodulation-division, RND), малая множественная лекарственная устойчивость (small multidrug resistance, SMR), мульти-антимикробные экструзионные белки (multidrug and toxin extrusion, MATE), суперсемейство мембранных транспортеров (major facilitator superfamily, MFS) и АТФ-связывающие кассетные транспортеры (ATP-binding cassette, ABC) (18, 19). В основе этой классификации лежат три критерия: идентичность аминокислотной последовательности, специфичность субстрата и источник энергии, необходимой для управления эффлюксом (20). В статье A.E. Ebbensgaard с соавт. (21) также упоминается семейство транспортеров протеобактериальных антимикробных соединений (proteobacterial antimicrobial compound efflux, PACE). В то время как RND и PACE уникальны для грамотрицательных бактерий, SMR, MATE, MFS и ABC обнаруживаются как у грамположительных, так и у грамотрицательных представителей микробного сообщества (21).
ABC-транспортеры функционально разнообразны и опосредуют АТФ-зависимый импорт или экспорт растворенных веществ. АВС-транспортеры содержат трансмембранные домены (ТМД), которые способны распознавать субстраты и посредством конформационных переключений переносить эти вещества через мембрану, и нуклеотид-связывающие домены (НСД), которые связывают и гидролизуют АТФ, управляя транспортным циклом (22, 23). Механизм переноса веществ указанного семейства действует по принципу переменного доступа, такая структура имеет три состояния: открыт вовнутрь, закрыт или открыт наружу для перемещения субстратов через мембрану.
Группа MFS — самое крупное и разнообразное семейство транспортеров. В нее входят унипортеры (обеспечивают перемещение субстратов через липидный бислой в одном направлении по градиенту концентрации, независимо от других молекул), симпортеры (транспорт вещества и ионов в одном направлении за пределы клетки) и антипортеры (перемещение ионов и веществ в противоположных направлениях). Большинство членов этого семейства функционирует как отдельные мономерные единицы. Их длина составляет от 400 до 600 аминокислотных остатков, и они имеют 12 или 14 трансмембранных спиралей, организованных как два домена, каждый из которых состоит из шести спиральных пучков. Механизм переключения переменного доступа для белков MFS имеет два состояния: открыт внутрь клетки или открыт наружу во время транспортного цикла.
Другие белки семейства MATE для транспорта используют трансмембранные градиенты H+ и/или Na+. Все известные в настоящее время структуры транспортеров MATE зафиксированы во внешнем состоянии (открыты наружу). Вероятно, состояние «открыт внутрь клетки» достигается за счет переноса липидов при помощи флиппазы на цитоплазматическую сторону мембраны (24).
Системы эффлюкса RND-типа состоят из трех компонентов: интегрального мембранного белка (integral membrane protein, IMP), белка слияния периплазматической мембраны (periplasmic membrane fusion protein, MFP) и белка внешней мембраны (outer membrane protein, OMP). Трансмемранный электрохимический потенциал H+ служит движущей силой эффлюкса лекарств, связанного с RND. Все транспортеры RND имеют довольно нетипичную структуру для группы вторичных транспортеров. Большинство систем транспорта суперсемейства RND состоят из больших полипептидных цепей, содержащих 12 трансмембранных доменов. Большой периплазматический домен участвует в распознавании субстрата и образует полость, которая может одновременно принимать несколько лекарств (25).
Белки семейства SMR состоят всего из четырех трансмембранных спиралей, но функционируют как гомодимеры или гетеродимеры. Первые три трансмембранные спирали образуют активную полость для связывания субстрата, тогда как четвертая спираль в основном участвует в димеризации. Во время транспорта чередуется два состояния насоса: обращенный внутрь или наружу посредством конформационного обмена двух протомеров (24).
Транспортные белки семейства PACE формируют устойчивость к ряду биоцидов, используемых в качестве дезинфицирующих и антисептических средств. Диапазон функций и механизмы транспорта, действующие в этих белках, изучены недостаточно. Известно, что белки PACE имеют несколько консервативных аминокислотных мотивов, которые, вероятно, играют роль в транспорте субстрата. У белков PACE также обнаружена консервативная область между N- и C-концевыми аминокислотами. Вероятно, они эволюционировали в результате дупликации предкового белка, состоящего из двух трансмембранных спиралей (26).
Перечисленные механизмы формирования бактериальной устойчивости выявлены в отношении тетрациклинов, макролидов, хинолонов и амфениколов. Около 30 генов резистентности к тетрациклину, например tet (A, B, C, D, E, G, H, J, K), кодируют специфичный для тетрациклина эффлюксный насос, которым обладают как грамположительные, так и грамотрицательные бактерии и который обычно кодируется транспозонами и интегронами. Помимо этого, несколько генов mef кодируют эффлюксный насос, специфичный для макролидов, который снижает их внутриклеточную концентрацию. В отличие от генов erm, гены mef могут защищать только от макролидов, что дает фенотип М. Существуют данные, что, хотя механизм эффлюкса, опосредующий устойчивость к хинолонам, чувствителен к резерпину, этот фенотип представляет собой скорее фенотип множественной лекарственной устойчивости, установленной неспецифическим эффлюксом (11). Гены cml и flo кодируют специфические эффлюксные насосы, обнаруженные в основном у грамотрицательных бактерий.
Ферментативная модификация или деградация (инактивация) противомикробного средства. Бактерии способны синтезировать ферменты, которые химически модифицируют мишень антибиотика, добавляя дополнительные химические группы. Например, гены mph в основном встречаются у грамотрицательных бактерий и опосредуют инактивацию макролидов, что ограничивает их клиническое значение. Гены vat, которые также кодируют инактивацию ферментов, обнаружены у Enterococcus spp. и Staphylococcus spp. (11). Второй тип ферментов действует посредством химической модификации самого антибиотика, что предотвращает связывание антибиотика с его целевым сайтом. Этот механизм антибиотикорезистентности могут обеспечивать ферменты, модифицирующие аминогликозиды, в частности N-ацетилтрансферазы, которые добавляют дополнительную ацетильную группу (CH3CO—) к аминогликозидным антибиотикам, например к канамицину. Связывание с рибосомой нарушается, бактерии приобретают устойчивость (27). Большинство из этих ферментов имеют узкий спектр действия. Например, ANT(2´´)-I способен инактивировать только гентамицин, тобрамицин и канамицин. Бифункциональные фосфотрансферазы и/или ацетилтрансферазы, обнаруженные в грамположительных кокках, инактивируют большинство аминогликозидов. Гены этих ферментов часто находятся в плазмидах и транспозонах и могут быть мобилизованы как генные кассеты между интегронами. Кроме того, некоторые из 1000 различных β-лактамаз, известных на сегодняшний день, способны гидролизовать только несколько субстратов, в то время как другие могут также инактивировать цефалоспорины третьего поколения (β-лактамазы расширенного спектра, БЛРС) и ингибиторы β-лактамаз, например клавулановую кислоту. β-Лактамазы широко рассредоточены по бактериальным группам и могут кодироваться хромосомными или плазмидными генами. Недавно обнаруженный модифицированный фермент устойчивости к аминогликозидам AAC(6´)-lb-cr способен инактивировать ципрофлоксацин. Фермент довольно часто встречается в клинической практике у изолятов кишечных бактерий с пониженной чувствительностью к ципрофлоксацину. Бактерии, продуцирующие хлорамфеникол-ацетилтрансферазу, которая кодируется множеством генов cat и обладает способностью к инактивации левомицетина, приобретают устойчивость к антибиотику. Гены cat были обнаружены как у грамположительных, так и у грамотрицательных бактерий (11). Основные типы ферментов, модифицирующие противомикробные препараты, представлены в таблице 1.
Приобретение альтернативных метаболических путей (формирование метаболического шунта). Этот механизм резистентности достаточно специфичен, чаще всего связан с приобретением бактериями новых генов, позволяющих продуцировать альтернативную мишень (обычно фермент), устойчивую к действию антибиотика. В то же время бактерии синтезируют и первоначальную мишень, которая обладает чувствительностью к антибиотикам. Альтернативная мишень опосредует развитие устойчивости у бактерий, принимая на себя роль исходной мишени, то есть формируется метаболический шунт (28, 29). Яркий пример такого механизма устойчивости к антибиотикам хинолонового ряда — имитация молекулы-мишени белком семейства пентапептидных повторов MfpA. Отмечалось, что при экспрессии MfpA на плазмиде pGADIV в Mycobacterium smegmatis или Mycobacterium bovis минимальная ингибирующая концентрация (МИК) для всех фторхинолонов увеличивалась 2-8-кратно, а когда mfpA был элиминирован из хромосомы M. smegmatis, МИК снижалась в 2-4 раза (30). Белок MfpA мимикрирует под структуру ДНК и взаимодействует с ДНК-гиразой или топоизомеразой IV, тем самым защищая их от ингибирующего действия хинолонов, которые связываются с этими ферментами-мишенями (31).
Другой класс антибиотиков — гликопептиды связываются концевыми остатками D-аланил-D-аланин с клеточной стенкой предшественников пентапептида, блокируя следующие стадии синтеза клеточной стенки (трансгликозилирование и транспептидацию). Гены van изменяют пути синтеза пептидогликана так, что вместо D-аланил-D-аланина образуется D-аланил-D-лактат или D-аланил-D-серин. Для достижения устойчивости к гликопептидам необходимы кластеры генов van (пять и более генов), поэтому весь кластер должен быть перемещен горизонтально, вероятно посредством конъюгации. Некоторые гены van, по всей видимости происходящие от микроорганизмов, продуцирующих ванкомицин, передались представителям рода Streptomyces, а затем грамположительным коккам (32, 33).
Действие β-лактамных антибиотиков основано на подавлении нескольких ферментов, ответственных за синтез стенок бактериальных клеток (пенициллин-связывающие белки, ПСБ). Приобретение альтернативных ферментов способствует развитию устойчивости клетки ко многим или ко всем β-лактамам. Так, Staphylococcus aureus становится устойчивым к большинству β-лактамных антибиотиков, в частности к пенициллину. Метициллинрезистентный золотистый стафилококк (methicillin-resistant Staphylo-coccus aureus, MRSA) приобретает устойчивость к β-лактамным антибиотикам за счет получения дополнительной копии пенициллин-связывающего белка 2a (PBP2a), который служит мишенью для β-лактамных антибиотиков и сохраняет свою функциональность в их присутствии (33). На основании данных B.A. Wall с соавт. (11) и D.M. Boothe (33) известно, что измененные ПСБ лежат в основе резистентности к пенициллину у Streptococcus pneumoniae, приобретенной через трансформацию, накопление повторяющихся точечных мутаций или через рекомбинацию между генами ПСБ у родственных видов стрептококков. Организмы, не имеющие клеточной стенки, например Mycoplasma, по своей природе устойчивы к β-лактамам и ко всем противомикробным препаратам, которые действуют, подавляя синтез клеточной стенки бактерий-мишеней или препятствуя ему (34). Фенотипическая форма устойчивости может возникать при наличии сферопластов (неполная клеточная стенка) или протопластов (отсутствие клеточной стенки). Таким L-формам необходима гиперосмотическая среда (например, мозговое вещество почек), чтобы выжить, в противном случае они будут лизированы (33).
Модификация или защита противомикробной мишени. Одним из распространенных механизмов, используемых бактериями для приобретения устойчивости к антибактериальным препаратам, служит изменение или защита мишени антибиотика. По мере роста и размножения бактерии копируют свой геном. Иногда в процессе копирования в последовательности ДНК происходят ошибки (например, аденин заменяется на цитозин). Сами по себе такие события редки, но большие размеры популяций значительно увеличивают частоту их встречаемости. Если одна из этих мутаций происходит в месте расположения гена, кодирующего белок — мишень антибиотика, то последний больше не может связываться с мишенью. Происходит отбор бактерий, устойчивых к действию противомикробного средства. Таков общий механизм устойчивости к пенициллину у Streptococcus pneumoniae, когда приобретаются мутации в пенициллин-связывающих белках (ПСБ).
В публикации P. Valderrama-Carmona с соавт. (35) сообщалось о метилазах 16S рРНК, которые модифицируют молекулу нуклеиновой кислоты, что изменяет структуру рибосомы, препятствуя связыванию аминогликозидов, у кишечных бактерий Pseudomonas spp. и грамположительных кокков (35). Также рибосомные мутации способны сделать рибосомы нечувствительными к аминогликозидам. Ген erm (семейство генов эритромицин-рибосомной метилазы) обеспечивает устойчивость к макролидным антибиотикам, таким как эритромицин. Метилирование происходит в части рибосомы, которая служит мишенью для эритромицина, эритромицин теряет способность связываться с мишенью, и бактерии могут продолжать развиваться в присутствии антибиотика (27). Подобная модификация защищает рибосому от других химически неродственных противомикробных препаратов, таких как линкозамиды и стрептограмины. Так называемый фенотип макролид-линкозамид-стрептограмин B (macrolide-lincosamide-streptogramin B, MLSB) служит наглядным примером перекрестной резистентности. Гены erm часто локализованы на мобильных генетических элементах, erm(B) и tet(M) располагаются в Tn1545 — конъюгативном транспозоне стрептококков (36). Сульфаниламиды, в свою очередь, способны ингибировать фермент дигидроптероатсинтетазу (dihydropteroate synthetase, DHPS), а триметоприм — дигидрофолат-редуктазу (dihydrofolate reductase, DHFR).
Особое значение имеет ген sul/I, кодирующий DHPS. Он служит частью консервативной области интегронов класса 1. Приобретая (посредством горизонтального переноса) гены ферментов DHPS и/или вариантов DHFR, которые не подавляются этими препаратами, бактерии становятся устойчивыми (11). Мутации в генах gyr и/или par, кодирующих соответственно ДНК-гиразу и топоизомеразу IV, позволяют указанным ферментам завершить трехэтапный процесс суперспирализации ДНК в присутствии хинолонов (37). Единичная мутация может сделать бактериальную клетку устойчивой к налидиксовой кислоте, но необходимы две или более мутации, чтобы добиться устойчивости к фторхинолонам (например, к ципрофлоксацину и энрофлоксацину). Хотя эти мутации рецессивны по своей природе (с предположительно ограниченной способностью к горизонтальной мобилизации), передача через трансформацию была зарегистрирована у стрептококков, поскольку вновь приобретенный ген заменял ген дикого типа посредством рекомбинации.
Ген cfr кодирует РНК-метилтрансферазу, в результате действия которой происходит модификация рибосомы, препятствующая связыванию с ней флорфеникола, что приводит к возникновению устойчивости. В настоящее время в медицине применение левомицетина ограничено, а флорфеникол используется только в ветеринарии. Тем не менее гены cfr имеют значение для здравоохранения, поскольку производимая метилаза также за-щищает бактериальные рибосомы от действия линезолида из класса оксазолидинонов, рассматриваемого как «последнее средство» против инфекций, связанных с золотистым стафилококком и резистентными энтерококками у людей. Гены cfr обнаружены во всем мире у клинических изолятов, устойчивых к линезолиду.
Примерно 10 генов, в число которых входят гены устойчивости к тетрациклину tet (M, O, Q, S, T), кодируют белки, которые взаимодействуют с рибосомами, защищая их от связывания с тетрациклинами. Помимо кишечных бактерий, гены tet, особенно tet(M), обычно обнаруживаются совместно с генами устойчивости к макролидам в том же транспозоне у грамположительных кокков, а также их можно обнаружить у анаэробов.
Группа генов qnr кодирует белок, защищающий ферменты группы топоизомераз от действия хинолонов. Эти гены впервые упоминались как уникальный плазмидоопосредованный механизм устойчивости к хинолонам, обнаруженный у кишечных бактерий. Позднее они были выявлены в хромосомах многих других организмов, наряду с родственными генами mdp схожей природы. Они кодируют низкую устойчивость к хинолонам, часто ниже контрольных точек для развития полной резистентности в клинических условиях (11).
Избыточная продукция фермента-мишени. Бактерии также могут избыточно синтезировать мишени антибиотиков, то есть концентрация белка-мишени превышает концентрацию самого антибиотика (38). Следовательно, целевого белка достаточно, чтобы он продолжал свою роль в клетке в присутствии антибиотиков. Этим обусловлен механизм устойчивости к триметоприму у Escherichia coli и Haemophilus influenzae. Триметоприм обычно используется с сульфаметоксазолом (комбинация, известная как ко-тримоксазол, или SXT). Сверхэкспрессия иногда обнаруживается в сочетании с мутациями, которые снижают способность антибиотика связываться с его мишенью. Мутанты со сверхэкспрессией DHPS и/или DHFR способны преодолеть ингибирующую способность антифолатных препаратов при терапевтических концентрациях и становиться резистентными (32).
Роль мобильных генетических элементов в распространении генов антибиотикорезистентности. Исследователям еще предстоит определить участие мобильных генетических элементов (МГЭ) в распространении антибиотикорезистентности (39).
Плазмиды участвуют в приобретении устойчивости к большинству классов антибиотиков, включая β-лактамы, аминогликозиды, тетрациклины, сульфаниламиды, триметопримы, макролиды, полимиксины и хинолоны, в основном у грамотрицательных бактерий (28, 40). Плазмиды, вызывающие множественную лекарственную устойчивость, обычно конъюгативны, способны инициировать не только свой собственный перенос, но и перенос других плазмид и обладают механизмами для контроля числа их копий в клетке и/или способности к репликации. Плазмиды гарантируют передачу генов антибиотикорезистентности посредством различных механизмов, таких как активные системы разделения, случайная сегрегация или постсегрегационное убийство. S. Nolivos с соавт. (41) выявлено, что тетрациклинустойчивые штаммы кишечной палочки передавали свою устойчивость более чем 70 % изначально тетрациклинчувствительных штаммов Escherichia coli всего лишь за 3 ч посредством конъюгационного переноса трансмиссивной плазмиды — фактора фертильности E. coli (F), несущей инсерцию в транспозоне Tn10. Помимо конъюгативных плазмид, существуют мобилизуемые плазмиды, которые меньше по размеру и не переносятся самостоятельно, но могут транспортировать ДНК конкретному хозяину в присутствии конъюгативных плазмид. Этот перенос происходит как вертикально, так и горизонтально (39).
Инсерционные последовательности (insertion sequences, IS) — самые маленькие (0,7-2,5 т.п.н.) и простейшие мобильные генетические элементы (МГЭ), обнаруженные у бактерий. Они фланкируются короткими, в основном инвертированными, повторами, которые иногда генерируют прямые дупликации мишеней (directed repeats, DR) во время интеграции в ДНК-мишени (42). В настоящее время в специализированной базе данных ISFinder (http://www-is.biotoul.fr) описано более 4500 IS (43). Роль IS в устойчивости к антибиотикам подчеркивалась многократно, особенно в исследованиях, в которых рассматривалась устойчивость к колистину и карбапенему (39). В отличие от сложных транспозонов, которые существуют только в виде единственной копии в специфическом репликоне, IS могут присутствовать в виде множественных копий, тем самым способствуя накоплению генов антибиотикорезистентности (аntibiotic resistance genes, ARG) (32).
Транспозоны (transposons, Tn) — категория МГЭ, несущих гены антибиотикорезистентности. Многие Tn обладают способностью перемещаться в разные участки генома (как интра-, так и интермолекулярно), опосредуя подвижность ARG (44). Бактериальные Tn можно разделить на два типа: составные (два IS-элемента, фланкирующих центральный ген) и сложные (содержащие ген tnpA, кодирующий транспозазу, и ген tnpR, кодирующий резольвазу). Преобладающими ARG-содержащими транспозонами служат Tn5 (кодирует устойчивость к неомицину и канамицину у Acinetobacter baumannii и Pseudomonas aeruginosa), Tn10 (кодирует устойчивость к тетрациклину), Tn9, Tn903, Tn1525 и Tn2350 (40).
Интегроны — это МГЭ, способные накапливать кассеты генов, включая ARG, и распространять их через другие мобильные генетические элементы. Чаще всего их обнаруживают у грамотрицательных бактерий, но они также присутствуют в грамположительных бактериях. Интегроны содержат ген интегразы (integrase gene, intI1) — фермента, осуществляющего сайт-специфическую рекомбинацию, которая приводит к инсерции одной или нескольких генных кассет в интегронную платформу. Интегроны делятся на несколько классов (1-й, 2-й и 3-й) в зависимости от аминокислотной последовательности фермента IntI (36). Интегроны 1-го класса, которые обычно ассоциированы с плазмидами, чаще всего встречаются у больничных клинических изолятов, а также обнаружены у бактериальных патогенов, выявляемых при контроле производства продуктов питания (например, на животноводческих фермах) (45).
Геномные острова — категория интегративных и конъюгативных элементов, способных опосредовать свою собственную эксцизию. Изучение множественных геномных островов выявило несколько общих и существенных характеристик этих хромосомных областей. Они представляют собой фрагменты ДНК размером 10-200 т.п.н., которые встраиваются в гены тРНК (46). Геномные острова содержат повторяющиеся последовательности распознавания и криптические гены, кодирующие факторы, которые участвуют в интеграции, инсерции или переносе (32).
Интегративные и конъюгативные элементы (integrative and conjugative elements, ICE) отвечают за горизонтальный перенос большинства факторов устойчивости и вирулентности. ICE имеют размер 18-600 т.п.н., схожи с геномными островами наличием инсерции в специфическом сайте, ассоциаций с генами фаговой интегразы и фланкированием инвертированными повторами. Вырезание и интеграция ICE осуществляется с помощью рекомбиназы, часто называемой интегразой. Интегразы, ассоциированные с ICE, представляют собой тирозиновые или сериновые рекомбиназы. Интегративные и конъюгативные элементы обладают способностью мобилизовать соседние последовательности, включая геномные островки или составные транспозоны, несущие ARG (39).
Основные виды генетических детерминант устойчивости к антибактериальным препаратам у микроорганизмов представлены в таблице 2.
Контроль и сдерживание распространения антибиотикорезистентности в животноводстве. Распространение устойчивости к противомикробным препаратам среди патогенов человека и животных представляет огромную угрозу для общественного здравоохранения в мировом масштабе. Использование противомикробных препаратов в медицине и ветеринарии, особенно применение больших количеств антибактериальных средств в животноводстве для стимулирования роста животных, усиливает неблагоприятную тенденцию появления и распространения бактерий, устойчивых к противомикробным препаратам (71, 72), что обостряет необходимость рационального применения антибиотиков.
Всемирная организация здравоохранения (WHO) классифицирует фторхинолоны, цефалоспорины 3-го и 4-го поколений, макролиды, гликопептиды и полимиксины как первоочередные и критически важные антибактериальные средства для медицины и ветеринарии (73). Однако антибиотики пенициллиного ряда, макролиды и фторхинолоны используются только для лечения человека, в то время как тетрациклины, пенициллины и сульфаниламиды применяются только для лечения животных.
В первую очередь следует с осторожностью использовать антибиотики резерва в животноводстве (эритромицин, олеандомицин, левомицетин, неомицин, мономицин, канамицин, гентамицин, ванкомицин, ципрофлоксацин) (74). Применение авопарцина в качестве кормовой добавки привело к появлению бактерий, резистентных к ванкомицину — антибиотику резерва для борьбы с опасными для жизни человека инфекциями, вызванными грамположительными бактериями (75). Недавно ветеринарной службой молекулярной генетики (Servei Veterinari de Genètica Molecular, SVGM) Автономного университета Барселоны (Universitat Autònoma de Barcelona, UAB) был проведен анализ и секвенирование образцов фекалий владельца фермы и содержащихся там животных (крупный рогатый скот, свиньи). Результаты показали, что выделенная культура E. coli в исследуемых образцах от телят и свиней, а также от фермера несла гены устойчивости к колистину — антибиотику «последней надежды» в медицине. Эксперты пришли к выводу, что ген устойчивости mcr-1 был передан от животных человеку посредством горизонтального переноса и обмена плазмидами между E. coli, поскольку телята и свиньи получали антибиотик в лечебно-профилактических целях, а владелец фермы никогда не проходил курс терапии этим препаратом (76).
Одна из основных задач заключается в сокращении использования антибиотиков в животноводстве посредством улучшения качества жизни и условий содержания животных. В связи с этим рекомендуется применение надлежащей практики содержания и обращения с животными на предприятиях животноводства и при транспортировке животных; улучшение благополучия животных (например, обеспечение оптимального микроклимата, качественной воды, соответствующей вентиляции и распределения площадей) на всех этапах, включая производство, транспортировку и убой; использование адаптированных к местным условиям пород, более устойчивых к болезням и стрессу, или животных, отбираемых на устойчивость к болезням (устойчивые животные потребуют меньшего количества обработок противомикробными препаратами); соблюдение ветеринарно-санитарных, санитарно-гигиенических правил, мер по биобезопасности на предприятиях АПК, чтобы предотвратить применение лекарственных средств; соблюдение строгих мер борьбы с болезнями (например, проведение вакцинаций); использование кормовых ингредиентов/добавок, повышающих эффективность конверсии корма для исключения применения антибиотиков в качестве стимуляторов роста (кормовые ферменты, пробиотики конкурентного действия, пребиотики, подкислители, растительные экстракты, нутрицевтики, эфирные масла, дрожжи и др.); отказ от ингредиентов корма с антипитательными свойствами (таких как лектины и ингибиторы протеаз); применение современных методов утилизации отходов. Особого внимания требует первичное производство (конкретные производственно-сбытовые этапы) и планирование практических действий, которые можно предпринять для уменьшения потребности в антибактериальных средствах и контроля за распространением устойчивых к противомикробным препаратам микроорганизмов в окружающей среде (11, 77).
Помимо этого, необходимо организовать и проводить мониторинг и надзор за распространением антибиотикоустойчивых бактерий, включающие оценку и определение тенденций и источников устойчивости к противомикробным препаратам у бактерий; обнаружение новых механизмов устойчивости к противомикробным препаратам; предоставление данных, необходимых для анализа рисков в отношении здоровья животных и человека; обеспечение основы для практических рекомендаций по охране здоровья животных и человека; предоставление информации для мониторинга назначений противомикробных препаратов в сельскохозяйственных организациях и разумного использования рекомендаций; оценку и определение эффективности мероприятий по борьбе с антибиотикорезистентностью (78-80). Поскольку устойчивость к антибиотикам возникает как часть необратимого процесса, его можно замедлить, но не остановить. Следовательно, всегда будет существовать необходимость разрабатывать новые антибиотики и диагностические тесты для борьбы с развитием резистентности (81).
Таким образом, разнообразные механизмы антибиотикорезистентности и способы их приобретения бактериями существенно затрудняют процесс подбора эффективной антибиотикотерапии как в условиях агропромышленных организаций, так и в лечебных учреждениях. Механизмы приобретенной и естественной антибиотикорезистентности по своей природе сложны и варьируют от вида к виду, от штамма к штамму микроорганизмов. В основном внутривидовое и межвидовое приобретение генов антибиотикорезистентности осуществляется посредством горизонтального переноса — конъюгации, трансформации, трансдукции. Основные меры борьбы с антибиотикорезистентностью включают сокращение использования антибиотиков посредством улучшения качества жизни и условий содержания животных; организацию и проведение мониторинга и надзора за распространением антибиотикоустойчивых бактерий; разработку новых антибиотиков и тест-систем для диагностики антибиотикорезистентности бактерий.
1. Основные типы ферментов, модифицирующие противомикробные препараты (27) |
||
Тип |
Название |
Инактивируемые антибиотики |
Гидролазы |
β-Лактамазы (пенициллиназы, |
Пенициллины, цефалоспорины, |
Эстеразы |
Макролиды |
|
Эпоксидгидролазы |
Фосфомицин |
|
Трансферазы |
Ацетилтрансферазы |
Аминогликозиды, фениколы, хинолоны, стрептограмин А |
Фосфотрансферазы |
Макролиды, рифамицины, фосфомицин, аминогликозиды |
|
Нуклеотидилтрансферазы |
Аминогликозиды, линкозамиды |
|
Гликозилтрансферазы |
Макролиды, рифамицины |
|
АДФ-рибозил-трансферазы |
Рифамицины |
|
Глутатион-S-трансферазы |
Фосфомицин |
|
Oкислительно-восстановительные |
Монооксигеназы |
Тетрациклины, рифамицины |
Лиазы |
Стрептограмин Б |
|
2. Примеры генетических детерминант устойчивости к антибактериальным препаратам у микроорганизмов |
||||
Ген-маркер резистентности (бактерии — носители генов) |
Локализация гена |
Что кодирует |
Механизм антибиотикорезистентности |
Источник |
аac(6´)-Ib |
Генные кассеты располагаются в первой позиции в интегронах 1-го класса, ассоциированы со слабыми вариантами промотора PC (PCW или PCH1) |
Резистентность к аминогликозидам (гентамицин) |
Ферментативная модификация аминогликозидных антибиотиков, а именно O-фосфорилирование, O-нуклеотидилирование и N-ацетилирование катализируется аминогликозидфосфотрансферазами (APH), аминогликозиднуклеотидилтрансферазами (ANT) и аминогликозидацетилтрансферазой (ААС) |
(47-49) |
aac(6´)-Ib-cr |
Резистентность к аминогликозидам (гентамицин) и фторхинолонам (ципрофлоксацинофлоксацин) |
Ферментативная модификация аминогликозидных антибиотиков, а именно O-фосфорилирование, O-нуклеотидилирование и N-ацетилирование катализируется аминогликозидфосфотрансферазами (APH), аминогликозиднуклеотидилтрансферазами (ANT) и аминогликозидацетилтрансферазой (ААС), мутация генов-мишеней; эффлюксный насос |
(47-49) |
|
strB, или aph(6)-Id, |
На неконъюгативной плазмиде RSF1010 широкого спектра хозяев; в транспозоне Tn5393 с инсерционной последовательностью IS1133 |
Резистентность к аминогликозидам (стрептомицин) |
Ферментативная модификация антибиотиков, катализируют модификацию групп —OH или —NH2 в ядре 2-дезоксистрептамина или сахарных фрагментов |
|
aphA, или aph(3')IIa |
В транспозоне Tn5 |
Резистентность к аминогликозидам (неомицин, канамицин) |
Ферментативная модификация антибиотиков |
|
blaCMY-2 |
Конъюгативная/ неконъюгативная плазмида IncA/C; |
Резистентность к β-лактамам (ампициллин, цефокситин, цефтриаксон, амоксициллин, цефтиофур) |
Продукция плазмидных |
|
blaTEM-1 |
На плазмиде pBR322 |
Резистентность к β-лактамам (ампициллин) |
Модификация мишени |
|
blaCARB-3 |
Мобильные генные кассеты располагаются в интегронах |
Резистентность к β-лактамам (ампициллин) |
Модификация мишени |
|
blaCTX-M |
На плазмидах семейства IncF |
Резистентность к β-лактамам (цефалоспорины 1-го поколения: цефазолин, цефалотин, цефалексин; |
Модификация мишени |
|
AmpC |
Однонуклеотидный полиморфизм, ген, придающий устойчивость к антибиотикам; ферменты AmpC кодируются хромосомными, плазмидными генами и перемещаются между хромосомами и плазмидами |
Резистентность к β-лактамам (цефалоспорины 4-го поколения: цефепим) |
Модификация мишени |
(50) |
blaVIM-1 |
В интегронах 1-го класса, которые являются производными Tn402 (также называемого Tn5090) — транспозона, характеризующегося наличием модуля транспозиции, который включает набор из четырех генов (tniR/tniC, tniQ, tniB и tniA). Некоторые из элементов Tn402 были обнаружены в различных Tn3-подобных транспозонах. У изолятов Enterobacteriaceae blaVIM-1 является частью интегронов, расположенных либо в элементе In2-Tn402, связанном с Tn21, либо в транспозоне Tn402, который ассоциирован соответственно с плазмидой IncHI2 или IncI1 |
Резистентность к β-лактамам (карбапенемы: меропенем, имипенем, дорипенем) |
Производство карбапенемаз, ферментов, способных гидролизовать почти все β-лактамы, или посредством модификаций внешней мембраны клетки, в частности, за счет снижения проницаемости клеточной мембраны в результате модификации порина и/или продукции эффлюксной помпы |
|
blaKPC-2 |
Расположен на родственном Tn3 транспозоне Tn4401, способном к высокой частоте транспозиции |
Резистентность к β-лактамам (карбапенемы: меропенем, имипенем, дорипенем) |
Продукция β-лактамаз (карбапенемаз), наличие эффлюксных насосов и мутаций, которые изменяют экспрессию и/или функцию поринов и пенициллин-связывающих белков (PBP) |
|
tetA, tetR, tetB, tetC, tetG |
В транспозоне Tn10 |
Резистентность к тетрациклинам (тетрациклин) |
Эффлюкс тетрациклино-специфическими насосами: препарат активно откачивается из бактериальной клетки в неизменном виде |
|
tetM |
В транспозонах Tn916 и Tn1545 |
|
Рибосома может функционировать при высоком содержание лекарственного средства внутри бактериальной клетки из-за сложных взаимодействий с другими бактериальными белками; белки препятствуют связыванию тетрациклина с рибосомой и обеспечивают некоторую степень защиты от него |
|
sul1, sul2, sul3 |
Располагаются на небольших конъюгативных плазмидах или больших трансимиссивных плазмидах с множественной резистентностью в интегронах 1-го класса |
Резистентность к сульфаниламидам (сульфаниламид) |
Модификация мишени антибиотика |
|
floR, cmlA |
Мобильные генные кассеты располагаются в интегронах |
Резистентность к фениколам (хлорамфеникол) |
Активный эффлюкс: устойчивость к антибиотикам за счет транспорта антибиотиков за пределы клетки |
|
cat1 |
В транспозоне Tn9 |
Резистентность к фениколам (хлорамфеникол, флорфеникол) |
Ферментативная инактивация антибиотика, вызывающая лекарственную устойчивость |
|
cfr |
В плазмиде pEF-01; в плазмиде pEC-01; |
Резистентность к оксазолидинонам, стрептограмину, линкозамиду, фениколам |
Мутационное изменение или ферментативная модификация мишени антибиотика, приводящая к устойчивости к антибиотикам |
|
fosA1 |
В транспозоне Tn2961 |
Резистентность к |
Ферментативная инактивация антибиотика, вызывающая лекарственную устойчивость |
|
mphA |
Инсерционная последовательность IS 26 в транспозоне Tn6242 в интегронах 1-го класса |
Резистентность к макролидам (азитромицин, эритромицин) |
Ферментативная инактивация антибиотика, вызывающая лекарственную устойчивость |
|
ErmB |
В транспозоне |
Резистентность к макролидам (эритромицин) |
Мутационное изменение или ферментативная модификация мишени антибиотика, приводящая к устойчивости к антибиотикам |
|
arr2 |
Генная кассета, локализующаяся в интегроне 1-го класса — In53, расположенном на составном транс-позоне или плазмиде |
Резистентность к рифампицину |
Ферментативная инактивация антибиотика, вызывающая лекарственную устойчивость |
|
VanA |
В транспозоне Tn5281 на плазмиде pBEM10; в транспозоне Tn1546 на плазмиде pIP816 |
Резистентность к гликолепидам (тейкопланин, ванкомицин) |
Мутационное изменение или ферментативная модификация мишени антибиотика, приводящая к устойчивости к антибиотикам. |
|
GyrA, ParC |
Локализация не выяснена |
Резистентность к фторхинолонам (ципрофлоксацин, офлоксацин, левофлоксацин, цефкином) |
Мутационное изменение или ферментативная модификация мишени антибиотика, приводящая к устойчивости к антибиотикам; активный эффлюкс: устойчивость к антибиотикам за счет транспорта антибиотиков за пределы клетки |
|
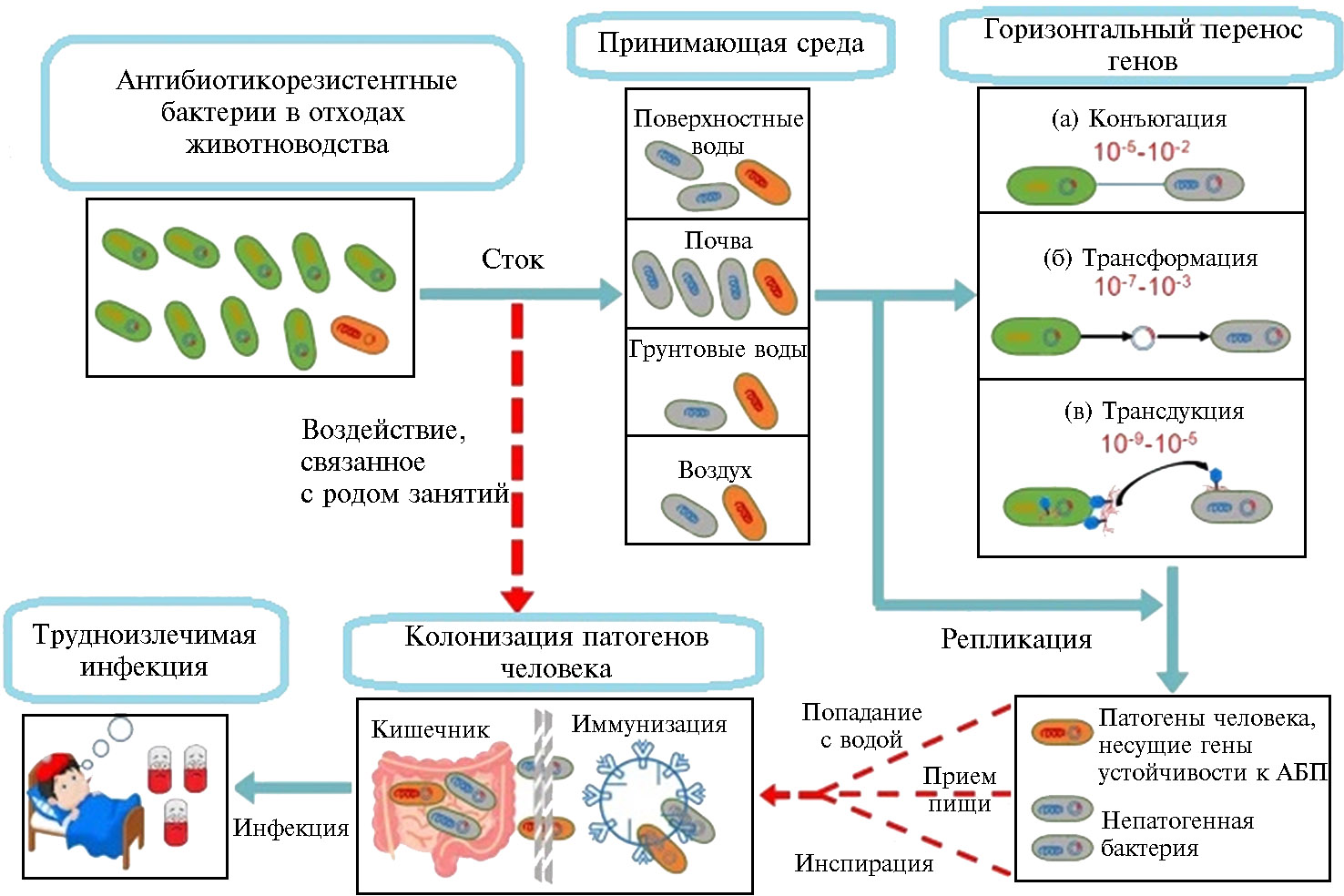
Возможные пути перемещения генов антибиотикорезистентности из отходов животноводства к патогенам человека.Бактерии, содержащие гены антибиотикорезистентности, перемещаются через дренаж, очищенные сточные воды и твердые отходы животноводческих предприятий в различные принимающие среды. Горизонтальный перенос генов между устойчивыми к антибактериальным препаратам (АБП) бактериями и аутохтонной микрофлорой осуществляется с участием трех основных механизмов: конъюгации (с участием плазмиды), трансформации (с участием свободной ДНК) и трансдукции (с участием бактериофагов) с указанными частотами, основанными на данных литературы. Патогенные бактерии способны проникать в организм человека алиментарным путем, а также в результате воздействия, связанного с родом занятий. Затем бактерии размножаются в организме (особенно в кишечнике), вызывая эндогенные или экзогенные инфекции (10).
ЛИТЕРАТУРА
- Communication from the commission to the council and the European parliament. A European one health action plan against antimicrobial resistance (AMR) COM/2017/0339 final. European Commission, 2017.
- Hassan Y.I., Lahaye L., Gong M.M., Peng J., Gong J., Liu S., Gay C.G., Yang C. Innovative drugs, chemicals, and enzymes within the animal production chain. Veterinary Research, 2018, 49: 71 (doi: 10.1186/s13567-018-0559-1).
- Vrancianu C.O., Gheorghe I., Czobor I.B., Chifiriuc M.C. Antibiotic resistance profiles, molecular mechanisms and innovative treatment strategies of Acinetobacter baumannii. Microorganisms, 2020, 8(6): 935 (doi: 10.3390/microorganisms8060935).
- Lim S.-K., Kim D., Moon D.-C., Cho Y., Rho M. Antibiotic resistomes discovered in the gut microbiomes of Korean swine and cattle. GigaScience, 2020, 9(5): giaa043 (doi: 10.1093/gigascience/giaa043).
- Broom L.J. The sub-inhibitory theory for antibiotic growth promoters. Poultry Science, 2017, 96(9): 3104-3108 (doi: 10.3382/ps/pex114).
- Redondo-Salvo S., Fernández-López R., Ruiz R., Vielva L., de Toro M., Rocha E.P.C., Garcillán-Barcia M.P., de la Cruz F. Pathways for horizontal gene transfer in bacteria revealed by a global map of their plasmids. Nature Communications, 2020, 11: 3602 (doi: 10.1038/s41467-020-17278-2).
- Zalewska M., Błażejewska A., Czapko A., Popowska M. Antibiotics and antibiotic resistance genes in animal manure — consequences of its application in agriculture. Frontiers in Microbiology, 2021, 12: 610656 (doi: 10.3389/fmicb.2021.610656).
- Van Boeckel T.P., Brower C., Gilbert M., Grenfell B.T., Levin S.A., Robinson T.P., Teillant A., Laxminarayan R. Global trends in antimicrobial use in food animals. Proceedings of the National Academy of Science,2015, 112(18): 5649-5654 (doi: 10.1073/pnas.1503141112).
- Минсельхоз РФ. Приказ об ограничении использования антибиотиков для лечения животных: проект нормативно-правовых актов, 2021.
- He Y., Yuan Q., Mathieu J., Stadler L., Senehi N., Sun R., Alvarez P.J.J. Antibiotic resistance genes from livestock waste: occurrence, dissemination, and treatment. npj Clean Water, 2020, 3: 4 (doi: 10.1038/s41545-020-0051-0).
- Wall B.A., Mateus A., Marshall L., Pfeiffer D.U., Lubroth J., Ormel H.J., Otto P., Patriarchi A. Drivers, dynamics and epidemiology of antimicrobial resistance in animal production. FAO, 2016.
- Бисекенова А.Л., Рамазанова Б.А., Адамбеков Д.А., Бекболатова К.А. Молекулярные механизмы резистентности грамотрицательных микроорганизмов — возбудителей инфекций к бета-лактамным антибиотикам. Вестник Казахского Национального медицинского университета, 2015, (3): 223-227.
- Закиров И.И., Кадырова Э.Р., Сафина А.И., Каюмов А.Р. Антибиотикорезистентность Staphylococcusaureus и Pseudomonasaeruginosa на модели муковисцидоза как хронического заболевания бронхолегочной системы. Педиатрия, 2018, 97(2): 176-186 (doi: 10.24110/0031-403X-2018-97-2-176-186).
- Pandey A., Agnihotri V. Antimicrobials from medicinal plants: Research initiatives, challenges, and the future prospects. In: Biotechnology of bioactive compounds: sources and applications /V.K. Gupta, M.G. Tuohy (eds.). John Wiley & Sons, 2015.
- Cesur S., Demiröz A.P. Antibiotics and the mechanisms of resistance to antibiotics. Medical Journal of Islamic World Academy of Sciences,2013,21(4): 138-142 (doi: 10.12816/0002645).
- Vergalli J., Bodrenko I.V., Masi M., Moynié L., Acosta-Gutiérrez S., Naismith J.H., Davin-Regli A., Ceccarelli M., van den Berg B., Winterhalter M., Pagès J.M. Porins and small-molecule translocation across the outer membrane of Gram-negative bacteria. Nature Reviews Microbiology, 2020, 18(3): 164-176 (doi: 10.1038/s41579-019-0294-2).
- Alcalde-Rico M., Hernando-Amado S., Blanco P., Martínez J.L. Multidrug efflux pumps at the crossroad between antibiotic resistance and bacterial virulence. Frontiers in Microbiology, 2016, 7: 1483 (doi: 10.3389/fmicb.2016.01483).
- Hernando-Amado S., Blanco P., Alcalde-Rico M., Corona F., Reales-Calderón J.A., Sánchez M.B., Martínez J.L. Multidrug efflux pumps as main players in intrinsic and acquired resistance to antimicrobials. Drug Resistance Updates, 2016, 28: 13-27 (doi: 10.1016/j.drup.2016.06.007).
- Reygaert W.C. An overview of the antimicrobial resistance mechanisms of bacteria. AIMS Microbiology, 2018, 4(3): 482-501 (doi: 10.3934/microbiol.2018.3.482).
- Housseini B., Issa K., Phan G., Broutin I. Functional mechanism of the efflux pumps transcription regulators from Pseudomonas aeruginosa based on 3D structures. Frontiers in Molecular Biosciences, 2018, 5: 57 (doi: 10.3389/fmolb.2018.00057).
- Ebbensgaard A.E., Løbner-Olesen A., Frimodt-Møller J. The role of efflux pumps in the transition from low-level to clinical antibiotic resistance. Antibiotics, 2020, 9(12): 855 (doi: 10.3390/antibiotics9120855).
- Johnson Z.L., Chen J. Structural basis of substrate recognition by the multidrug resistance protein MRP1. Cell, 2017, 168(6): 1075-1085.e9 (doi: 10.1016/j.cell.2017.01.041).
- Verhalen B., Dastvan R., Thangapandian S., Peskova Y., Koteiche H.A., Nakamoto R.K., Tajkhorshid E., Mchaourab H.S. Energy transduction and alternating access of the mammalian ABC transporter P-glycoprotein. Nature, 2017, 543(7647): 738-741 (doi: 10.1038/nature21414).
- Du D., Wang-Kan X., Neuberger A., van Veen H.W., Pos K.M., Piddock L., Luisi B.F. Multidrug efflux pumps: structure, function and regulation. Nature ReviewsMicrobiology, 2018, 16(9): 523-539 (doi: 10.1038/s41579-018-0048-6).
- Toba S., Minato Y., Kondo Y., Hoshikawa K., Minagawa S., Komaki S., Kumagai T., Matoba Y., Morita D., Ogawa W., Gotoh N., Tsuchiya T., Kuroda T. Comprehensive analysis of resistance-nodulation-cell division superfamily (RND) efflux pumps from Serratia marcescens, Db10. Scientific Reports, 2019, 9(1): 4854 (doi: 10.1038/s41598-019-41237-7).
- Hassan K.A., Liu Q., Elbourne L., Ahmad I., Sharples D., Naidu V., Chan C.L., Li L., Harborne ;S., Pokhrel A., Postis V., Goldman A., Henderson P., Paulsen I.T. Pacing across the membrane: the novel PACE family of efflux pumps is widespread in Gram-negative pathogens. Research in Microbiology, 2018, 169(7-8): 450-454 (doi: 10.1016/j.resmic.2018.01.001).
- Egorov A.M., Ulyashova M.M., Rubtsova M.Y. Bacterial enzymes and antibiotic resistance. Acta Naturae, 2018, 10(4): 33-48 (doi: 10.32607/20758251-2018-10-4-33-48).
- Giedraitienė A., Vitkauskienė A., Naginienė R., Pavilonis A. Antibiotic resistance mechanisms of clinically important bacteria. Medicina, 2011, 47(3): 137-146 (doi: 10.3390/medicina47030019).
- Хайтович А.Б. Подходы к решению проблемы лекарственной устойчивости в мире и Российской Федерации. Лекция. Крымскийжурналэкспериментальной и клинической медицины, 2018, 8(2): 81-95.
- Mayer C., Takiff H. The molecular genetics of fluoroquinolone resistance in Mycobacterium tuberculosis. Microbiology Spectrum, 2014, 2(4): MGM2-2013 (doi: 10.1128/microbiolspec.MGM2-0009-2013).
- Bush N.G., Diez-Santos I., Abbott L.R., Maxwell A. Quinolones: mechanism, lethality and their contributions to antibiotic resistance. Molecules, 2020, 25(23): 5662 (doi: 10.3390/molecules25235662).
- Землянко О.М., Рогоза Т.М., Журавлева Г.А. Механизмы множественной устойчивости бактерий к антибиотикам. Экологическаягенетика, 2018, 16(3): 4-17 (doi: 10.17816/ecogen1634-17).
- Boothe D.M. β-Lactam Antibiotics. Pharmacology. MSD Veterinary Manual, 2015. Режим доступа: https://www.msdvetmanual.com/pharmacology/antibacterial-agents/β-lactam-antibiotics. Дата обращения: 01.11.2021.
- Peterson E., Kaur P. Antibiotic resistance mechanisms in bacteria: relationships between resistance determinants of antibiotic producers, environmental bacteria, and clinical pathogens. Frontiers in Microbiology, 2018, 9: 2928 (doi: 10.3389/fmicb.2018.02928).
- Valderrama-Carmona P., Cuartas J.H., Castaño D.C., Corredor M. The role of Pseudomonas aeruginosa RNA methyltransferases in antibiotic resistance. In: Pseudomonas Aeruginosa — an armory within /D. Sriramulu (ed.). IntechOpen, London, 2019 (doi: 10.5772/intechopen.85185).
- Bezborodova N.A., Sokolova O.V., Shkuratova I.A., Lysova Ya.Yu., Isakova M.N., Kozhukhovskaya V.V. Sensitivity and resistance of the microbiota of reproductive organs and mammary gland of cows to anti-microbial agents in cases of inflammation. International Journal of Biology and Biomedical Engineering, 2020, 14: 49-54 (doi: 10.46300/91011.2020.14.8).
- Sultan I., Rahman S., Jan A.T., Siddiqui M.T., Mondal A.H., Haq Q. Antibiotics, resistome and resistance mechanisms: a bacterial perspective. Frontiers in Microbiology, 2018, 9: 2066 (doi: 10.3389/fmicb.2018.02066).
- Varela M.F., Stephen J., Lekshmi M., Ojha M., Wenzel N., Sanford L.M., Hernandez A.J., Parvathi A., Kumar S.H. Bacterial resistance to antimicrobial agents. Antibiotics, 2021, 10(5): 593 (doi: 10.3390/antibiotics10050593).
- Vrancianu C.O., Popa L.I., Bleotu C., Chifiriuc M.C. Targeting plasmids to limit acquisition and transmission of antimicrobial resistance. Frontiers in Microbiology, 2020, 11: 761 (doi: 10.3389/fmicb.2020.00761).
- Bello-López J.M., Cabrero-Martínez O.A., Ibáñez-Cervantes G., Hernández-Cortez C., Pelcastre-Rodríguez L.I., Gonzalez-Avila L.U., Castro-Escarpulli G. Horizontal gene transfer and its association with antibiotic resistance in the genus Aeromonas spp. Microorganisms, 2019, 7(9): 363 (doi: 10.3390/microorganisms7090363).
- Nolivos S., Cayron J., Dedieu A., Page A., Delolme F., Lesterlin C. Role of AcrAB-TolC multidrug efflux pump in drug-resistance acquisition by plasmid transfer. Science, 2019, 364(6442): 778-782 (doi: 10.1126/science.aav6390).
- Vandecraen J., Chandler M., Aertsen A., Van Houdt R. The impact of insertion sequences on bacterial genome plasticity and adaptability. Critical Reviews in Microbiology, 2017, 43(6): 709-730 (doi: 10.1080/1040841X.2017.1303661).
- Siguier P., Perochon J., Lestrade L., Mahillon J., Chandler M. ISfinder: the reference centre for bacterial insertion sequences. Nucleic Acids Research, 2006, 34 (suppl_1): D32-D36 (doi: 10.1093/nar/gkj014).
- Babakhani S., Oloomi M. Transposons: the agents of antibiotic resistance in bacteria. Journal of Basic Microbiology, 2018, 58(11): 905-917 (doi: 10.1002/jobm.201800204).
- Belaynehe K.M., Shin S.W., Yoo H.S. Interrelationship between tetracycline resistance determinants, phylogenetic group affiliation and carriage of class 1 integrons in commensal Escherichia coli isolates from cattle farms. BMC Veterinary Research, 2018, 14(1): 340 (doi: 10.1186/s12917-018-1661-3).
- da Silva Filho A.C., Raittz R.T., Guizelini D., De Pierri C.R., Augusto D.W., Dos Santos-Weiss I., Marchaukoski J.N. Comparative analysis of genomic island prediction tools. Frontiers in Genetics, 2018, 9: 619 (doi: 10.3389/fgene.2018.00619).
- McMillan E.A., Gupta S.K., Williams L.E., Jové T., Hiott L.M., Woodley T.A., Barrett J.B., Jackson C.R., Wasilenko J.L., Simmons M., Tillman G.E., McClelland M., Frye J.G. Antimicrobial resistance genes, cassettes, and plasmids present in Salmonella enterica associated with united states food animals. Frontiers in Microbiology, 2019, 10: 832 (doi: 10.3389/fmicb.2019.00832).
- Sophie R., Thomas J., Margaux G., Emilie P., Aurore T., Carmen T., Marie-Cécile P. Expression of the aac(6′)-Ib-cr gene in class 1 integrons. Antimicrobial Agents and Chemotherapy, 2021, 61(5): e02704-16 (doi: 10.1128/AAC.02704-16).
- Vetting M.W., Park C.H., Hegde S.S., Jacoby G.A., Hooper D.C., Blanchard J.S. Mechanistic and structural analysis of aminoglycoside N-acetyltransferase AAC(6')-Ib and its bifunctional, fluoroquinolone-active AAC(6')-Ib-cr variant. Biochemistry, 2008, 47(37): 9825-9835 (doi: 10.1021/bi800664x).
- Alcock B.P., Raphenya A.R., Lau T.T.Y., Tsang K.K., Bouchard M., Edalatmand A., Huynh W., Nguyen A.-L., Cheng A.A., Liu S., Min S.Y., Miroshnichenko A., Tran H.-K., Werfalli R.E., Nasir J.A., Oloni M., Speicher D.J., Florescu A., Singh B., Faltyn M., Hernandez-Koutoucheva A., Sharma A.N., Bordeleau E., Pawlowski A.C., Zubyk H.L., Dooley D., Griffiths E., Maguire F., Winsor G.L., Beiko R.G., Brinkman F.S.L., Hsiao W.W.L., Dom-selaar G.V., McArthur A.G. CARD 2020: antibiotic resistome surveillance with the Comprehensive Antibiotic Resistance Database. Nucleic Acids Research, 2020, 48(D1): D517-D525.
- Ковтун А.С., Алексеева М.Г., Аверина О.В., Даниленко В.Н. Идентификация аминогликозидфосфотрансфераз клинических штаммов бактерий в микробиоте жителей России. Вестник РГМУ, 2017, 2: 14-19 (doi: 10.24075/brsmu.2017-02-02).
- Goessens W.H., van der Bij A.K., van Boxtel R., Pitout J. D., van Ulsen P., Melles D.C., Tommassen J. Antibiotic trapping by plasmid-encoded CMY-2 β-lactamase combined with reduced outer membrane permeability as a mechanism of carbapenem resistance in Escherichia coli. AntimicrobialAgentsand Сhemotherapy, 2013, 57(8): 3941-3949 (doi: 10.1128/AAC.02459-12).
- Chang P.H., Juhrend B., Olson T.M., Marrs C.F., Wigginton K.R. Degradation of extracellular antibiotic resistance genes with UV254 treatment. Environmental Science & Technology, 2017, 51(11): 6185-6192 (doi: 10.1021/acs.est.7b01120).
- Potron A., Poirel L., Croizé J., Chanteperdrix V., Nordmann P. First ESBL-Derivative CARB-Type beta-lactamase from Acinetobacter baumannii. Antimicrobial Agents and Chemotherapy, 2009, 53(7): 3010-3016 (doi: 10.1128/AAC.01164-08).
- Bevan E.R., Jones A.M., Hawkey P.M. Global epidemiology of CTX-M β-lactamases: temporal and geographical shifts in genotype. The Journal of Antimicrobial Chemotherapy, 2017, 72(8): 2145-2155 (doi: 10.1093/jac/dkx146).
- Falgenhauer L., Ghosh H., Guerra B., Yao Y., Fritzenwanker M., Fischer J., Helmuth R., Imirzalioglu C., Chakraborty T. Comparative genome analysis of IncHI2 VIM-1 carbapenemase-encoding plasmids of Escherichia coli and Salmonella enterica isolated from a livestock farm in Germany. Veterinary Microbiology, 2017, 200: 114-117 (doi: 10.1016/j.vetmic.2015.09.001).
- Tato M., Coque T.M., Baquero F., Cantón R. Dispersal of carbapenemase blaVIM-1 gene associated with different Tn402 variants, mercury transposons, and conjugative plasmids in Enterobacteriaceae and Pseudomonas aeruginosa. Antimicrobial Agents and Chemotherapy, 2010, 54(1): 320-327 (doi: 10.1128/AAC.00783-09).
- Jiansheng H., Xiaolei H., Yunan Z., Yang S., Hui D., Rongzhen W., Zhigang Z., Jiansong J. Comparative analysis of blaKPC expression in Tn4401 transposons and the Tn3-Tn4401 chimera. Antimicrobial Agents and Chemotherapy, 2021, 63(5): e02434-18 (doi: 10.1128/AAC.02434-18).
- Vikram A., Schmidt J.W. Functional blaKPC-2 sequences are present in U.S. beef cattle feces regardless of antibiotic use. Foodborne Pathogens and Disease, 2018, 15(7): 444-448 (doi: 10.1089/fpd.2017.2406).
- Clark D.P., Pazdernik N.J. Transgenic animals. In: Biotechnology, 2nd ed. Elsevier, Amsterdam, 2016: 493-521 (doi: 10.1016/B978-0-12-385015-7.00016-8).
- Jiang H., Cheng H., Liang Y., Yu S., Yu T., Fang J., Zhu C. Diverse mobile genetic elements and conjugal transferability of sulfonamide resistance genes (sul1, sul2, and sul3) in Escherichia coli isolates from Penaeus vannamei and pork from large markets in Zhejiang, China. Frontiers in Microbiology, 2019, 10: 1787 (doi: 10.3389/fmicb.2019.01787).
- Wang Y.-H., Li X.-N., Chen C., Zhang J., Wang G.-Q. Detection of floR gene and active efflux mechanism of Escherichia coli in Ningxia, China. Microbial Pathogenesis, 2018, 117: 310-314 (doi: 10.1016/j.micpath.2018.02.042).
- Shen J., Wang Y., Schwarz S. Presence and dissemination of the multiresistance gene cfr in Gram-positive and Gram-negative bacteria. Journal of Antimicrobial Chemotherapy, 2013, 68(8): 1697-1706 (doi: 10.1093/jac/dkt092).
- Argudín M.A., Deplano A., Meghraoui A., Dodémont M., Heinrichs A., Denis O., Nonhoff C., Roisin S. Bacteria from animals as a pool of antimicrobial resistance genes. Antibiotics, 2017, 6(2): 12 (doi: 10.3390/antibiotics6020012).
- Yang T.-Y., Lu P.-L., Tseng S.-P. Update on fosfomycin-modified genes in Enterobacteriaceae. Journal of Microbiology, Immunology and Infection, 2019, 52(1): 9-21 (doi: 10.1016/j.jmii.2017.10.006).
- Yang S., Deng W., Liu S., Yu X., Mustafa G.R., Chen S., He L., Ao X., Yang Y., Zhou K., Li B., Han X., Xu X., Zou L. Presence of heavy metal resistance genes in Escherichia coli and Salmonella isolates and analysis of resistance gene structure in E. coli E308. Journal of Global Antimicrobial Resistance, 2020, 21: 420-426 (doi: 10.1016/j.jgar.2020.01.009).
- Wan T.W., Hung W.C., Tsai J.C., Lin Y.T., Lee H., Hsueh P.R., Lee T.F., Teng L.J. Novel structure of Enterococcus faecium-originated ermb-positive Tn1546-like element in Staphylococcus aureus. Antimicrobial Agents and Chemotherapy, 2016, 60(10): 6108-6114 (doi: 10.1128/AAC.01096-16).
- Nikibakhsh M., Firoozeh F., Badmasti F., Kabir K., Zibaei M. Molecular study of metallo-β-lactamases and integrons in Acinetobacter baumannii isolates from burn patients. BMC Infectious Diseases, 2021, 21(1): 782 (doi: 10.1186/s12879-021-06513-w).
- Wardal E., Kuch A., Gawryszewska I., Żabicka D., Hryniewicz W., Sadowy E. Diversity of plasmids and Tn1546-type transposons among VanA Enterococcus faecium in Poland. European Journal of Clinical Microbiology & Infectious Diseases, 2017, 36(2): 313-328 (doi: 10.1007/s10096-016-2804-8).
- Kareem S.M., Al-Kadmy I., Kazaal S.S., Mohammed Ali A.N., Aziz S.N., Makharita R.R., Algammal A.M., Al-Rejaie S., Behl T., Batiha G.E., El-Mokhtar M.A., Hetta H.F. Detection of gyrA and parC mutations and prevalence of plasmid-mediated quinolone resistance genes in Klebsiella pneumoniae. Infection and Drug Resistance, 2021, 14: 555-563 (doi: 10.2147/IDR.S275852).
- Vidovic N., Vidovic S. Antimicrobial resistance and food animals: influence of livestock environment on the emergence and dissemination of antimicrobial resistance. Antibiotics, 2020, 9(2): 52 (doi: 10.3390/antibiotics9020052).
- Sharif Z., Peiravian F., Salamzadeh J., Mohammadi N.K., Jalalimanesh A. Irrational use of antibiotics in Iran from the perspective of complex adaptive systems: redefining the challenge. BMCPublicHealth, 2021, 21(1): 778 (doi: 10.1186/s12889-021-10619-w).
- Ma F., Xu S., Tang Z., Li Z., Zhang L. Use of antimicrobials in food animals and impact of transmission of antimicrobial resistance on humans. Biosafety and Health, 2021, 3(1): 32-38 (doi: 10.1016/j.bsheal.2020.09.004).
- Collignon P.C., Conly J.M., Andremont A., McEwen S.A., Aidara-Kane A., WHO-AGISAR, Agerso Y., Andremont A., Collignon P., Conly J., Dang Ninh T., Donado-Godoy P., Fedorka-Cray P., Fernandez H., Galas M., Irwin R., Karp B., Matar G., McDermott P., McEwen S., Mitema E., Reid-Smith R., Scott H.M., Singh R., DeWaal C.S., Stelling J., Toleman M., Watanabe H., Woo G.J. World Health Organization ranking of antimicrobials according to their importance in human medicine: a critical step for developing risk management strategies to control antimicrobial resistance from food animal production. Clinical Infectious Diseases, 2016, 63(8): 1087-1093 (doi: 10.1093/cid/ciw475).
- Wijesekara P.N.K., Kumbukgolla W.W., Jayaweera J.A.A.S., Rawat D. Review on usage of vancomycin in livestock and humans: maintaining its efficacy, prevention of resistance and alternative therapy. Veterinary Sciences, 2017, 4(1): 6 (doi: 10.3390/vetsci4010006).
- Viñes J., Cuscó A., Napp S., Alvarez J., Saez-Llorente J.L., Rosàs-Rodoreda M., Migura-Garcia L. Transmission of similar Mcr-1 carrying plasmids among different Escherichia coli lineages isolated from livestock and the farmer. Antibiotics, 2021, 10(3): 313 (doi: 10.3390/antibiotics10030313).
- Manyi-Loh C., Mamphweli S., Meyer E., Okoh A. Antibiotic use in agriculture and its consequential resistance in environmental sources: potential public health implications. Molecules, 2018, 23(4): 795 (doi: 10.3390/molecules23040795).
- Панин A.H., Комаров А.А., Куликовский А.В., Макаров Д.А. Проблема резистентности к антибиотикам возбудителей болезней, общих для человека и животных. Ветеринария, зоотехния и биотехнология, 2017, 5: 18-24.
- FAO. Monitoring and surveillance of antimicrobial resistance in bacteria from healthy food animals intended for consumption. Regional Antimicrobial Resistance Monitoring and Surveillance Guidelines, 2019, 1: 9-10.
- O’Neill J. Review on antimicrobial resistance: tackling drug-resistant infections globally: final report and recommendations. HM Government and Wellcome Trust, London, 2016.
- Windels E.M., Michiels J.E., Van den Bergh B., Fauvart M., Michiels J. Antibiotics: combatting tolerance to stop resistance. mBio, 2019, 10(5): e02095-19 (doi: 10.1128/mBio.02095-19).