doi: УДК 636.4:636.082:575.22:577.21
УДК 633.1:581.143.6
При выполнении исследований использовалось оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ВИЖ им. академика Л.К. Эрнста. Исследования выполнены в рамках ГЗ 0445-2019-0026, рег. № АААА-А18-118021590138-1.
ПОИСК ГЕНОМНЫХ ОБЛАСТЕЙ, НЕСУЩИХ ЛЕТАЛЬНЫЕ РЕЦЕССИВНЫЕ ВАРИАНТЫ У СВИНЕЙ ПОРОДЫ ДЮРОК
О.В. КОСТЮНИНА1, А.С. АБДЕЛЬМАНОВА1, Е.У. МАРТЫНОВА2, Н.А. ЗИНОВЬЕВА1
Эмбриональные потери у свиней достигают 30 %, и необходимость их снижения не вызывает сомнений. В качестве одного из генетических факторов, обусловливающих эмбриональную смертность, рассматриваются LoF (loss of function) мутации, которые в гомозиготном состоянии приводят к терминации синтеза белка или синтезу нефункциональных белков. Если у крупного рогатого скота проводится интенсивный поиск LoF мутаций, то у свиней такие исследования носят менее масштабный характер. Полногеномные методы анализа с использованием SNP (single nucleotide polymorphism) чипов средней и высокой плотности, равномерно охватывающих весь геном, позволяют применять новые подходы для идентификации позиционных кандидатов для летальных рецессивных аллелей. Один из таких подходов — анализ степени неравновесия по сцеплению (LD) аллелей SNP маркеров. В представленной работе с использованием анализа степени неравновесия по сцеплению мы впервые провели поиск областей в геноме, которые могут быть связаны с летальными рецессивными эффектами у свиней породы дюрок, и выявили ряд наиболее значимых однонуклеотидных полиморфизмов, локализованных в пределах генов и играющих важную роль в различных физиологических процессах. Нашей целью был поиск областей, несущих презумптивно летальные рецессивные варианты у свиней (Sus scrofa) породы дюрок, на основании анализа неравновесия по сцеплению аллелей в SNP локусах. Исследования проводили на 715 хрячках (ООО «Селекционно-гибридный центр», Воронежская обл.) в 2017-2019 годах. Полногеномное генотипирование осуществляли с использованием ДНК-чипов Porcine GGP HD («Neogene/Illumina, Inc.», США), содержащих около 70 тыс. SNP. По результатам контроля качества для анализа был отобран 42981 полиморфный SNP. Поиск референтных последовательностей (reference sequence, rs) и уточнение их локализации проводили в базе данных Ensembl (http://www.ensembl.org). Функциональные аннотации генов выполняли с использованием базы данных GeneCards (http://www.genecards.org). Анализ сохранения генетического равновесия показал наличие 990 SNP с отсутствием одного из гомозиготных генотипов (2,30 % от общего числа полиморфных SNP), которые были распределены между всеми хромосомами свиней, в том числе 205 SNP, находившихся в неравновесии по сцеплению (0,48 %). Наибольшей долей SNP, которые находились в неравновесии по сцеплению, характеризовались хромосомы SSC9 (0,85 %), SSC5 (0,77 %), SSC7 (0,68 %) и SSC2 (0,68 %), наименьшей — хромосомы SSC13 (0,28 %), SSC4 (0,29 %) и SSC10 (0,30 %). Для 52 SNP, 25 из которых были локализованы внутри генов, различия в частоте наблюдаемой и ожидаемой гетерозиготности оказались статистически значимы (p < 0,01). Среди SNP, расположенных в межгенных областях, два (rs81350198 и rs81337222) были ассоциированы с хозяйственно полезными фенотипами (по результатам ранее проведенных GWAS исследований). Для 12 из 25 выявленных позиционных генов-кандидатов (OR4C45, EPHB4, EML4, SLC4A1AP,ZFAT, CELSR2, NEGR1, LRRC32, MYOCD, HUNK, RPH3A и DOCK1) имеется информация о роли в различных процессах в организме млекопитающих, включая развитие нервной системы, ангиогенез, кардиогенез, дифференцировку клеток, апоптоз и многих другие. Интеграция в программы селекционно-племенной работы ДНК маркеров, ассоциированных с летальными фенотипами, в дополнение к ДНК маркерам, идентифицированным по результатам GWAS исследований, существенно повысит эффективность программ маркерной и геномной селекции в свиноводстве.
Ключевые слова: свиньи, неравновесие по сцеплению, летальные варианты, LoF (loss of function), однонуклеотидные полиморфизмы.
Эмбриональные потери оказывают значительное негативное влияние на эффективность и рентабельность отраслей животноводства. В свиноводстве они составляют до 30 % (1). Среди генетических факторов, ассоциированных с эмбриональной смертностью, рассматриваются так называемые LoF (loss of function) мутации, которые в гомозиготном состоянии приводят к терминации синтеза или синтезу нефункциональных белков (2). Наиболее активно поиск LoF мутаций осуществляется у крупного рогатого скота различных пород с использованием соответствующих тест-систем диагностики в практике сельскохозяйственных предприятий (3). В свиноводстве исследования LoF мутаций пока носят менее масштабный характер. Для идентификации мутаций, приводящих к видимым изменениям, часто используется подход, основанный на анализе родословных (4). Однако он непригоден для идентификации LoF мутаций, поскольку их фенотипический эффект проявляется в снижении многоплодия, что может быть обусловлено рядом других причин.
Развитие методов анализа полногеномных данных открыло новые возможности для поиска генетических факторов, ассоциированных с эмбриональной смертностью. Чтобы успешно использовать ДНК маркеры для выявления летальных рецессивных вариантов, вызывающих пренатальную смертность, требуется достаточная степень неравновесия по сцеплению (linkage disequilibrium, LD) между маркерным алеллем (аллелями) и летальным вариантом (вариантами) (5). Два аллеля в различных локусах находятся в неравновесии по сцеплению, если частота состоящего из них гаплотипа значимо отличается от частоты, ожидаемой при случайной сегрегации. Для лучшего выявления редких и нетипичных вариантов, которые не включены в общий массив генотипируемых однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP), применяют гаплотипический подход. Фазирование генотипических данных обеспечивает высокую четкость гетерогенности гаплотипов и позволяет сделать вывод о гаплотипах негенотипированных предков и животных, которых генотипировали с использованием матриц более низкой плотности. Такой подход был применен при выявлении летального рецессивного гаплотипа, связанного с числом мертворожденных поросят (6).
M.F.L. Derks с соавт. (7) искали летальные аллели, сегрегирующие в популяциях свиней пород ландрас (n = 28085) и дюрок (n = 11255), с использованием SNP-чипа средней плотности («Illumina, Inc.», США). Применив метод с перекрывающимся скользящим окном, они обнаружили один сильный кандидатный гаплотип (DU1), несущий летальный рецессивный аллель в популяции свиней породы дюрок, и четыре гаплотипа-кандидата у породы ландрас (LA1-4). Среди гаплотипов DU1, LA1 и LA3 не выявлялись гомозиготы, в то время как их ожидаемое чило составляло соответственно 26, 126 и 16. Для гаплотипов LA2 и LA4 также отмечалось нарушение генетического равновесия, что может свидетельствовать о неполном LD гаплотипов с летальными рецессивными мутациями. Показано влияние всех пяти гаплотипов на значительное снижение общего количества рожденных поросят (total number born, TNB) и числа живорожденных поросят (number born alive, NBA). Вместе с тем существенного увеличения числа мертворожденных или мумифицированных поросят не наблюдали, что свидетельствует о гибели гомозиготных носителей на ранних этапах супоросности (7).
В представленной работе с использованием анализа степени неравновесия по сцеплению мы впервые провели поиск областей в геноме, которые могут быть связаны с летальными рецессивными эффектами у свиней породы дюрок и выявили ряд наиболее значимых однонуклеотидных полиморфизмов, локализованных в пределах генов и играющих важную роль в различных физиологических процессах.
Нашей целью был поиск областей, несущих презумптивно летальные рецессивные варианты у свиней (Sus scrofa) породы дюрок, на основании анализа степени неравновесия по сцеплению аллелей в SNP локусах.
Методика. Исследования проводили на 715 хрячках породы дюрок (ООО «Селекционно-гибридный центр», Воронежская обл., 2017-2019 годы).
Биоматериалом для выделения геномной ДНК с использованием набора реагентов ДНК-Экстран-2 (ООО «Синтол», Россия) были пробы ткани (ушной выщип). Качество и концентрацию ДНК определяли с помощью флуориметра Qubit 2.0 («Invitrogen/Life Technologies», США) и cпектрофотометра NanoDrop8000 («Thermo Fisher Scientific», США).
Полногеномное генотипирование проводили с использованием чипов GGP Porcine («Neogene/Illumina, Inc.», США), содержащих около 70 тыс. SNP. В пакете Plink 1.9 (http://zzz.bwh.harvard.edu/plink/) по параметрам качества генотипирования (выше 90 %), частоты минорных аллелей (не более 0,5 %) и неравновесия по сцеплению (с шагом 50 kb) (8) для анализа отобрали 42981 полиморфный SNP. Поиск референтных последовательностей (reference sequence, rs) и уточнение их локализации осуществляли в базе данных Ensembl (http://www.ensembl.org, дата обращения: август 2019 года). Функциональные аннотации генов выполняли с помощью базы данных GeneCards (http://www.genecards.org, дата обращения: август 2019 года).
Результаты. Анализ сохранения генетического равновесия показал наличие 990 SNP с отсутствием одного из гомозиготных генотипов, из которых 205 SNP находились в неравновесии по сцеплению, что составляло соответственно 2,30 и 0,48 % от общего числа полиморфных SNP (рис.).
Наибольшее число SNP, для которых наблюдалось отсутствие одного из гомозиготных генотипов, было локализовано на 1-й хромосоме (Sus scrofa chromosome 1, SSC1) — 193 SNP, а находящихся в неравновесии по сцеплению — на SSC7 и SSC9 (20 SNP), наименьшее — соответственно на SSC16 (11 SNP) и SSC18 (5 SNP). Наибольшей долей SNP, находящихся в неравновесии по сцеплению, характеризовались хромосомы SSC9 (0,85 %), SSC5 (0,77 %), SSC7 (0,68 %) и SSC2 (0,68 %), наименьшей — SSC13 (0,28 %), SSC4 (0,29 %) и SSC10 (0,30 %).
Наиболее значимые полиморфизмы, для которых отмечалось неравновесие по сцеплению (p ≤ 0,01), представлены в таблице 1.
Всего идентифицировали 52 значимых полиморфизма, для которых различия в наблюдаемой и ожидаемой частоте гетерозиготности были статистически значимы. Эти SNP распределялись между 17 хромосомами (кроме SSC18), в том числе 5 SNP на SSC1, 6 — на SSC2, 6 — на SSC3, 4 — на SSC4, 3 — на SSC5, 3 — на SSC6, 3 — на SSC7, 2 — на SSC8, 3 — на SSC9, 1 — на SSC10, 3 — на SSC11, 3 — на SSC12, 3 — на SSC13, 3 — на SSC14, 1 — на SSC15, 1 — на SSC16, 2 — на SSC17. Два SNP (rs81350198 на SSC1 и rs81337222 на SSC6) по результатам GWAS (genome-wide as-sociation study, полногеномный поиск ассоциаций) идентифицированы в качестве ДНК маркеров, ассоциированных с хозяйственно полезными фенотипами (9, 10). Полиморфизм rs81350198 ассоциирован со вкусом мяса у некастрированных хряков, обусловленным накоплением скатола и андростенона в период половой зрелости (9). Для полиморфизма rs81337222 выявлена умеренная ассоциация (р = 2,4×10-5) с возникновением пупочной грыжи (10). Несмотря на то, что в непосредственной близости от rs81337222 не были идентифицированы QTL (quantitative trait loci), которые связаны с пупочной грыжей, и не локализованы описанные гены, для которых подтверждена связь с этим фенотипическим признаком, его презумптивный QTL может быть расположен в up-stream регионе SSC6 (6:3 814 021-3 870 534), выявленном при анализе CNV (сopy number variation) (11). Следовательно, в начале SSC6 имеются регуляторные области, которые требуют дополнительного исследования.
Анализ геномных регионов, в которых мы обнаружили SNP, показал, что 25 SNP находились внутри генов. По типу локализации большинство SNP представляли собой мутации внутри интронов (всего 15 мутаций). Были также обнаружены замены в 3'-последовательности генов (6 мутаций) и в 3'-нетранслируемой области гена (2 мутации), а также одна миссенс-мутация и одна синонимическая мутация (табл. 2).
Для 12 из 25 выявленных позиционных генов-кандидатов известна информация об их роли в различных процессах в организме млекопитающих. Так, ген OR4C45 кодирует белок обонятельного рецептора (ОР), который важен для поддержания кишечного гомеостаза. ОР экспрессируется в энтерохромаффинных клетках слизистой оболочки. Одорантные лиганды через ОР, присутствующие в клетках энтерохромаффина, вызывают высвобождение серотонина, который контролирует моторику и секрецию кишечника и вовлечен в патологические состояния, такие как рвота, диарея (13). Сообщается о роли ОР лиганда в регуляции проницаемости эпителия и секреции электрогенных анионов в толстой кишке человека (14). EPHB4 играет особую роль в разнообразных биологических процессах, таких как развитие нейронов, гомеостаз костей, ангиогенез (15). Генетически модифицированные эмбрионы мышей, гомозиготные по аллелю EphB4taulacZ, имели сердечно-сосудистые дефекты и характеризовались эмбриональной летальностью с очень высокой пенетрантностью. У таких эмбрионов отмечалось замедление роста, отсутствие кровотока и остановка развития сердца (16). EML4 — плохо охарактеризованный белок, ассоциированный с микротрубочками. Предполагается, что его естественная функция заключается в стабилизации микротрубочек в аксонах и дендритах нейрональных клеток. Химерный EML4-ALK обусловливает развитие рака легких у человека (17).
Канадаптин (SLC4A1AP) — ядерный белок с неизвестной функцией, который широко экспрессируется в тканях млекопитающих. Повсеместное распространение канадаптина у млекопитающих предполагает, что он должен играть важную физиологическую роль (18). ZFAT принимает участие в развитии и периферическом гомеостазе Т-клеток. Имеются сведения, что делеция в гене Zfat у мышей приводит к эмбриональной смерти и нарушает примитивный гематопоэз в кровяных островках желточного мешка (19, 20). У свиней ZFAT связан с подверженностью энтеротоксигенной инфекции, вызываемой Escherichia coli (21). CELSR2 экспрессируется во всех областях мозга и регулирует поддержание и рост дендритов. У гомозиготных по мутации в CELSR2 мышах развивается гидроцефалия из-за снижения количества, размера и ориентации эпендимальных ресничек (22). NEGR1 участвует в регуляции разрастания нейритов в развивающемся мозге (23). В результате поиска QTL, связанных с ожирением у человека и свиней, выявлены три наиболее вероятных гена,в том числе NEGR1, ответственный за генетическую предрасположенность к распространенным формам ожирения, в особенности за толщину подкожного жира (24). LRRC32 функционирует как рецептор для молекул латентного трансформирующего фактора роста, он был обнаружен в регуляторных Т-клетках (25). Отмечается его важная роль в иммунной регуляции. GWAS исследования показали ассоциацию между rs11236909, расположенным примерно на расстоянии 58 kb up-stream от гена LRRC32, и некоторыми параметрами подвижности сперматозоидов человека (26).
MYOCD вносит вклад в развитие сердца и дифференцировку кардиомиоцитов. Отмечается, что у мышей-мутантов с нокаутом гена MYOCD развивалась дилатационная кардиомиопатия, которая сопровождалась на-рушением структурной организации кардиомиоцитов и выраженной депрессией систолической функции (27). Функции гена HUNK еще уточняются. Вероятно, он участвует в переносе фосфорсодержащих групп, проявлении активности трансферазы и тирозинкиназы белка. RPH3A играет важную роль в адгезии нейтрофилов на эндотелиальных клетках во время воспалительных реакций (28). DOCK1 регулирует фагоцитоз, слияние ми-областов и миграцию клеток, участвует в эмбриональном развитии. Выявленное уменьшение развития всех тканей скелетных мышц у эмбрионов с нокаутом по Dock1 позволило идентифицировать DOCK1 как важный регулятор стадии слияния в миогенезе у млекопитающих (29). Информацию о роли гена ENSSSCG00000024474 в открытых базах данных обнаружить не удалось, но при GWAS анализе метилирования ДНК у сальных, постных и миниатюрных пород свиней он был идентифицирован как дифференциально метилированная область (30). Ген C20orf194 (194-я открытая рамка считывания 20-й хромосомы) кодирует неохарактеризованный белок с С-концевой областью спиральной катушки. Ген расположен на хромосоме 20p13 в области 1,8 Mb, связанной с фенотипом спиноцеребеллярной атаксии у человека. Исследования S. Ponsuksili с соавт. (31) по определению регионов, связанных с поведенческими реакциями у свиней породы ландрас, показали наличие в пределах этого гена SNP rs80988530, выявленного нами в регионе с высоким неравновесным сцеплением.
Таким образом, полногеномные исследования, которые мы проведенные с использованием чипов GGP Porcine HD, позволили определить регионы, мутации в которых могут стать причиной возникновения летальных эффектов. Наиболее значимые однонуклеотидные полиморфизмы, находящиеся в неравновесном сцеплении у хрячков породы дюрок, локализованы нами в пределах следующих генов: OR4C45 — обонятельный рецептор, семейство 4, подсемейство C, член 45; EPHB4 — EPH-рецептор B4; EML4 —белок, связанный с избыточно экспрессированной пролиферацией; SLC4A1AP — канадаптин; ZFAT — белок цинкового пальца; CELSR2 — семиповторный рецептор 2 кадгерина G-типа; NEGR1 — регулятор роста нейронов 1; LRRC32 — богатый лейцином повторяющийся белок 32; MYOCD — миокардин; HUNK — гормонально активируемая Neu-ассоциированная киназа; RPH3A — рабфилин 3А; DOCK1 — дедикатор цитокинеза. Практически для каждого идентифицированного гена-кандидата к настоящему времени охарактеризована его важная роль в различных процессах: развитии нервной системы, ангиогенезе, кардиогенезе, дифференцировке клеток, апоптозе и др. У человека многие из этих генов ассоциированы с заболеваниями различных органов и тканей, не исключено их участие в возникновении летальных эффектов у свиней. Понимание процессов, протекающих в период роста и развития эмбрионов, и использование этих знаний при анализе фактических данных зоотехнического учета расширят арсенал средств, позволяющих своевременно предлагать подходы по генетическому совершенствованию племенной продукции. Интеграция в программы селекционно-племенной работы ДНК маркеров, ассоциированных с летальными фенотипами, в дополнение к ДНК маркерам, идентифицированным по результатам GWAS анализа, существенно повысит эффективность программ маркерной и геномной селекции в свиноводстве.
1. Наиболее значимые однонуклеотидные полиморфизмы, находящиеся в неравновесном сцеплении у хрячков |
||||||||
SSC |
RS |
Позиция на хромосоме (сборка 10.2) |
A1A1 |
A1A2 |
A2A2 |
Ho |
He |
р |
1 |
rs81350198 |
226 188 042 |
0 |
163 |
547 |
0,2296 |
0,2032 |
5,1×10-5 |
1 |
rs80795638 |
277 854 630 |
0 |
227 |
440 |
0,3403 |
0,2824 |
1,7×10-10 |
1 |
rs81002425 |
296 461 073 |
0 |
700 |
9 |
0,9873 |
0,4999 |
5,4×10-195 |
1 |
rs334911415 |
312 050 746 |
0 |
662 |
45 |
0,9364 |
0,4980 |
1,2×10-152 |
1 |
rs342062641 |
312 083 991 |
0 |
645 |
63 |
0,9110 |
0,4960 |
1,2×10-137 |
2 |
rs81362641 |
12 082 068 |
0 |
618 |
92 |
0,8704 |
0,4916 |
1,7×10-117 |
2 |
rs81255095 |
14 056 188 |
0 |
345 |
309 |
0,5275 |
0,3884 |
5,2×10-28 |
2 |
rs319913462 |
14 759 005 |
0 |
522 |
126 |
0,8056 |
0,4811 |
4,0×10-84 |
2 |
rs323641934 |
15 384 171 |
0 |
537 |
172 |
0,7574 |
0,4706 |
1,1×10-76 |
2 |
rs343381067 |
15 551 096 |
0 |
665 |
45 |
0,9366 |
0,4980 |
2,0×10-153 |
2 |
rs80911461 |
160 111 983 |
0 |
186 |
506 |
0,2688 |
0,2327 |
7,8×10-7 |
3 |
rs344115015 |
8 107 291 |
0 |
139 |
570 |
0,1961 |
0,1768 |
9,7×10-4 |
3 |
rs323044318 |
104 257 589 |
0 |
120 |
590 |
0,1690 |
0,1547 |
6,4×10-3 |
3 |
rs81375606 |
116 445 052 |
0 |
124 |
586 |
0,1746 |
0,1594 |
4,1×10-3 |
3 |
rs81375903 |
118 615 471 |
0 |
142 |
568 |
0,2000 |
0,1800 |
6,0×10-4 |
3 |
rs80828678 |
132 719 179 |
0 |
348 |
305 |
0,5329 |
0,3909 |
1,9×10-28 |
3 |
rs327044542 |
141265 677 |
0 |
170 |
540 |
0,2394 |
0,2108 |
1,8×10-5 |
4 |
rs331053365 |
4 353 353 |
0 |
156 |
554 |
0,2197 |
0,1956 |
1,4×10-4 |
4 |
rs323787335 |
7 185 799 |
0 |
591 |
114 |
0,8383 |
0,4869 |
1,8×10-103 |
4 |
rs343205058 |
109 273 333 |
0 |
604 |
106 |
0,8507 |
0,4889 |
5,1×10-109 |
4 |
rs80949619 |
121 334 654 |
0 |
123 |
587 |
0,1732 |
0,1582 |
6,7×10-3 |
5 |
rs340620949 |
1 453 085 |
0 |
163 |
546 |
0,2299 |
0,2035 |
5,1×10-5 |
5 |
rs81323749 |
18 046 364 |
0 |
184 |
526 |
0,2592 |
0,2256 |
2,4×10-6 |
5 |
rs80875559 |
99 245 749 |
0 |
119 |
591 |
0,1676 |
0,1536 |
6,5×10-3 |
6 |
rs81337222 |
13 141 |
0 |
709 |
1 |
0,9986 |
0,5000 |
3,3×10-210 |
6 |
rs81476539 |
67 022 132 |
0 |
237 |
471 |
0,3347 |
0,2787 |
6,2×10-11 |
6 |
rs337799081 |
130 798 722 |
0 |
130 |
580 |
0,1831 |
0,1663 |
2,5×10-3 |
7 |
rs319008071 |
27 213 610 |
0 |
237 |
451 |
0,3445 |
0,2851 |
3,0×10-11 |
7 |
rs80944793 |
129 041 116 |
0 |
120 |
590 |
0,1690 |
0,1547 |
6,4×10-3 |
7 |
rs331172717 |
131 889 604 |
0 |
614 |
79 |
0,8860 |
0,4935 |
4,5×10-122 |
8 |
rs81399201 |
31 445 286 |
0 |
134 |
576 |
0,1887 |
0,1709 |
1,6×10-3 |
8 |
rs322099448 |
78 592 741 |
0 |
580 |
127 |
0,8204 |
0,4839 |
5,5×10-97 |
9 |
rs343201786 |
11 913 668 |
0 |
137 |
573 |
0,1930 |
0,1743 |
9,8×10-4 |
9 |
rs346413844 |
12 946 073 |
0 |
638 |
3 |
0,9953 |
0,5000 |
2,7×10-185 |
9 |
rs81337172 |
15 049 063 |
0 |
125 |
585 |
0,1761 |
0,1606 |
4,0×10-3 |
10 |
rs81305281 |
76 905 575 |
0 |
520 |
189 |
0,7334 |
0,4645 |
4,7×10-70 |
11 |
rs80816476 |
939 424 |
0 |
131 |
579 |
0,1845 |
0,1675 |
2,5×10-3 |
11 |
rs325221950 |
21 018 670 |
0 |
145 |
561 |
0,2054 |
0,1843 |
3,7×10-4 |
11 |
rs329067201 |
21 371 677 |
0 |
148 |
562 |
0,2085 |
0,1867 |
3,7×10-4 |
12 |
rs81478101 |
26 712 700 |
0 |
697 |
13 |
0,9817 |
0,4998 |
2,4×10-189 |
12 |
rs81436301 |
50 907 615 |
0 |
342 |
368 |
0,4817 |
0,3657 |
1,9×10-24 |
12 |
rs81228589 |
59 880 454 |
0 |
137 |
573 |
0,1930 |
0,1743 |
9,8×10-4 |
13 |
rs329645817 |
5 325 304 |
0 |
320 |
390 |
0,4507 |
0,3491 |
5,3×10-21 |
13 |
rs322958990 |
205 932 444 |
0 |
276 |
404 |
0,4059 |
0,3235 |
7,0×10-16 |
13 |
rs328137225 |
218 478 357 |
0 |
135 |
575 |
0,1901 |
0,1721 |
1,6×10-3 |
14 |
rs80958173 |
41 518 669 |
0 |
125 |
585 |
0,1761 |
0,1606 |
4,0×10-3 |
14 |
rs80862470 |
95 881 035 |
0 |
624 |
86 |
0,8789 |
0,4927 |
1,6×10-121 |
14 |
rs80993446 |
148 199 735 |
0 |
128 |
582 |
0,1803 |
0,1640 |
4,3×10-3 |
15 |
rs337254355 |
2 154 617 |
0 |
138 |
570 |
0,1949 |
0,1759 |
9,6×10-4 |
16 |
rs334615079 |
81 560 836 |
0 |
381 |
321 |
0,5427 |
0,3955 |
5,0×10-32 |
17 |
rs80988530 |
36 703 557 |
0 |
587 |
68 |
0,8962 |
0,4946 |
4,3×10-120 |
17 |
rs345268841 |
66 549 094 |
0 |
258 |
450 |
0,3644 |
0,2980 |
5,09×10-13 |
Примечание. SSC — номер хромосомы, RS — референтная последовательность, А1 — аллель 1, |
||||||||
2. Однонуклеотидные полиморфизмы, локализованные внутри генов и презумптивно ассоциированные с летальными |
|||
RS (р) |
Название гена |
Ген-кандидат |
Тип мутации |
rs343381067 (2,0×10-153) |
ENSSSCG00000031436 |
OR4C45 |
Замена внутри интрона |
rs344115015 (9,7×10-4) |
ENSSSCG00000007675 |
EPHB4 |
Замена внутри интрона |
rs323044318 (6,4×10-3) |
ENSSSCG00000008467 |
EML4 |
Замена внутри интрона |
rs81375606 (4,1×10-3) |
ENSSSCG00000008533 |
– |
Замена внутри интрона |
rs81375903 (6,0×10-4) |
ENSSSCG00000008549 |
SLC4A1AP |
Замена на 3'-конце гена |
rs327044542 (1,8×10-5) |
ENSSSCG00000049737 |
– |
Замена внутри интрона |
rs323787335 (1,8×10-103) |
ENSSSCG00000030947 |
ZFAT |
Мутация в 3'-UTR |
rs343205058 (5,1×10-109) |
ENSSSCG00000006694 |
– |
Замена внутри интрона |
rs80949619 (6,7×10-3) |
ENSSSCG00000034360 |
CELSR2 |
Замена внутри интрона |
rs340620949 (5,1×10-5) |
ENSSSCG00000024474 |
– |
Замена на 3'-конце гена |
rs81323749 (2,4×10-6) |
ENSSSCG00000024474 |
– |
Замена на 3'-конце гена |
rs81476539 (6,2×10-11) |
ENSSSCG00000003444 |
– |
Замена на 3'-конце гена |
rs337799081 (2,5×10-3) |
ENSSSCG00000025085 |
NEGR1 |
Замена внутри интрона |
rs319008071 (3,0×10-11) |
ENSSSCG00000001395 |
– |
Синонимическая мутация |
rs343201786 (9,8×10-4) |
ENSSSCG00000014869 |
LRRC32 |
Мутация в 3'-UTR |
rs325221950 (3,7×10-4) |
ENSSSCG00000045677 |
– |
Замена на 3'-конце гена |
rs81436301 (1,9×10-24) |
ENSSSCG00000017853 |
– |
Замена на 3'-конце гена |
rs81228589 (9,8×10-4) |
ENSSSCG00000031988 |
MYOCD |
Замена внутри интрона |
rs322958990 (7,0×10-16) |
ENSSSCG00000029392 |
HUNK |
Замена внутри интрона |
rs80958173 (4,0×10-3) |
ENSSSCG00000009883 |
RPH3A |
Замена внутри интрона |
rs80862470 (1,6×10-121) |
ENSSSCG00000043778 |
– |
Замена внутри интрона |
rs80993446 (4,3×10-3) |
ENSSSCG00000035045 |
DOCK1 |
Замена внутри интрона |
rs337254355 (9,6×10-4) |
ENSSSCG00000044919 |
– |
Замена внутри интрона |
rs80988530 (4,3×10-120) |
ENSSSCG00000007155 |
C20orf194 |
Замена внутри интрона |
rs345268841 (5,09×10-13) |
ENSSSCG00000007525 |
– |
Миссенс-мутация |
Примечание. OR4C45 — обонятельный рецептор, семейство 4, подсемейство C, член 45; |
|||
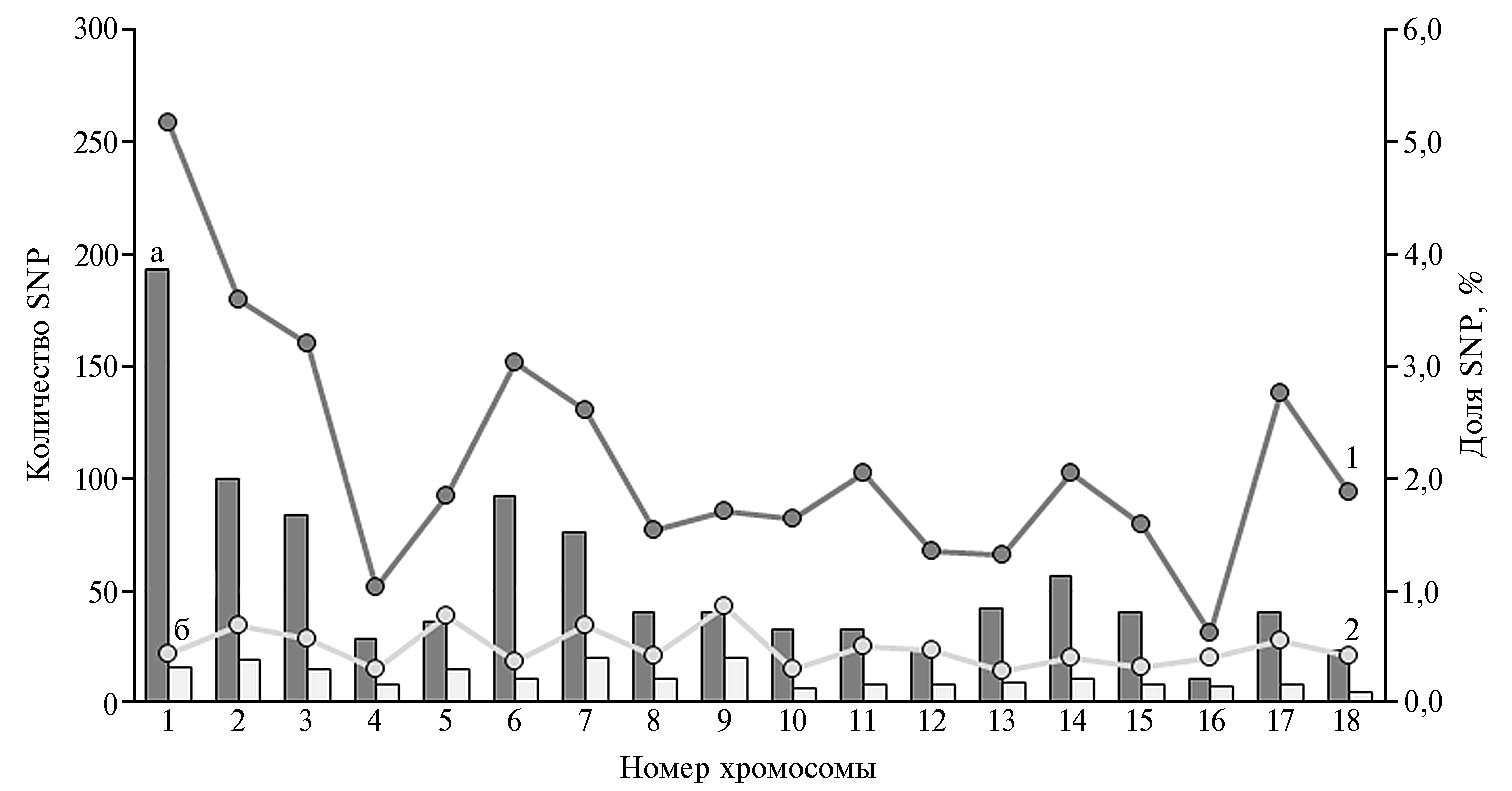
Хромосомальное распределение SNP (single nucleotide polymorphism), по которым зафиксировано отсутствие одного из гомозиготных вариантов и нарушение неравновесия по сцеплению у хрячков (Sus scrofa) породы дюрок: а — число SNP, для которых отсутствует один из гомозиготных генотипов, б — число SNP, находящихся в неравновесии по сцеплению (столбчатая диаграмма); 1 — доля SNP, для которых отсутствует один из гомозиготных генотипов, от общего числа полиморфных SNP на хромосоме, 2 — доля SNP, находящихся в неравновесии по сцеплению, от общего числа полиморфных SNP на хромосоме (линейный график) (n = 715, ООО «Селекционно-гибридный центр», Воронежская обл., 2017-2019 годы).
ЛИТЕРАТУРА
- Pope W.F. Embryonic mortality in swine. In: Embryonic mortality in domestic species /M.T. Zavy, R.D. Geisert (eds.). CRC Press, Boca Raton, 1994: 53-77.
- Bickhart D.M., Hou Y., Schroeder S.G., Alkan C., Cardone M.F., Matukumalli L.K., Song J., Schnabel R.D., Ventura M., Taylor J.F., Garcia J.F., Van Tassell C.P., Sonstegard T.S., Eichler E.E., Liu G.E. Copy number variation of individual cattle genomes using next-generation sequencing. Genome Res., 2012, 22(4): 778-790 (doi: 10.1101/gr.133967.111).
- Зиновьева Н.А. Гаплотипы фертильности голштинского скота. Сельскохозяйственная биология, 2016, 51(4): 423-435 (doi: 10.15389/agrobiology.2016.4.423rus).
- Gorbach D., Mote B., Totir L., Fernando R., Rothschild M. Polydactyl inheritance in the pig. Journal of Heredity, 2010, 101(4): 469-475 (doi: 10.1093/jhered/esq037).
- Howard D.M., Pong-Wong R., Knap P.W., Woolliams J.A. Use of haplotypes to identify regions harbouring lethal recessive variants in pigs. Genetics Selection Evolution, 2017, 49: 57 (doi: 10.1186/s12711-017-0332-3).
- Häggman J., Uimari P. Novel harmful recessive haplotypes for reproductive traits in pigs. J. Anim. Breed. Genet., 2017, 134(2): 129-135 (doi: 10.1111/jbg.12240).
- Derks M.F.L., Gjuvsland A.B., Bosse M., Lopes M.S., van Son M., Harlizius B., Tan B.F., Hamland H., Grindflek E., Groenen M.A.M., Megens H.-J. Loss of function mutations in essential genes cause embryonic lethality in pigs. PLoS Genet., 2019, 15(3): e1008055 (doi: 10.1371/journal.pgen.1008055).
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M.A.R., Bender D., Maller J., Sklar P., de Bakker P.I.W., Daly M.J., Sham P.C. PLINK: A tool set for whole-genome association and population-based linkage analyses. The American Journal of Human Genetics, 2007, 81(3): 559-575 (doi: 10.1086/519795).
- Drag M., Hansen M.B., Kadarmideen H.N. Systems genomics study reveals expression quantitative trait loci, regulator genes and pathways associated with boar taint in pigs. PLoS ONE, 2018, 13(2): e0192673 (doi: 10.1371/journal.pone.0192673).
- Fernandes L.T., Ono R.K., Ibelli A.M.G., Lagos E.B., Morés M.A.Z., Cantão M.E., Lorenzetti W.R., Peixoto J. de O., Pedrosa V.B., Ledur M.C. Novel putative candidate genes associated with umbilical hernia in pigs. Proc. World Congress on Genetics Applied to Livestock Production. Electronic Poster Session. Species. Porcine 2. Auckland, New Zealand, 2018: 743.
- Long Y., Su Y., Ai H., Zhang Z., Yang B., Ruan G., Xiao S., Liao X., Ren J., Huang L., Ding N. A genome-wide association study of copy number variations with umbilical hernia in swine. Anim. Genet., 2016, 47(3): 298-305 (doi: 10.1111/age.12402).
- Борисевич Д.И., Шаталова Л.В., Коростин Д.О., Ильинский В.В. Уточнение статуса некоторых мутаций, считающихся патогенными, с помощью признаков безвредных мутаций. Вестник Российского государственного медицинского университета,2016, 1: 20-24.
- Braun T., Voland P., Kunz L., Prinz C., Gratzl M. Enterochromaffin cells of the human gut: sensors for spices and odorants. Gastroenterology, 2007, 132(5): 1890-1901 (doi: 10.1053/j.gastro.2007.02.036).
- Kaji I., Karaki S., Kuwahara A. Effects of luminal thymol on epithelial transport in human and rat colon. American Journal of Physiology-Gastrointestinal and Liver Physiology, 2011, 300(6): G1132-G1143 (doi: 10.1152/ajpgi.00503.2010).
- Becerikli M., Merwart B., Lam M.C., Suppelna P., Rittig A., Mirmohammedsadegh A., Stricker I., Theiss C., Singer B.B., Jacobsen F., Steinstraesser L. EPHB4 tyrosine-kinase receptor expression and biological significance in soft tissue sarcoma. Int. J. Cancer., 2015, 136(8): 1781-1791 (doi: 10.1002/ijc.29244).
- Gerety S.S., Wang H.U., Chen Z.F., Anderson D.J. Symmetrical mutant phenotypes of the receptor EphB4 and its specific transmembrane ligand ephrin-B2 in cardiovascular development. Molecular Cell, 1999, 4(3): 403-414 (doi: 10.1016/s1097-2765(00)80342-1).
- Fry A.M., O'Regan L., Montgomery J., Adib R., Bayliss R. EML proteins in microtubule regulation and human disease. Biochemical Society Transactions,2016, 44(5): 1281-1288 (doi: 10.1042/BST20160125).
- Xu Q., Deller M.C., Nielsen T.K., Grant J.C., Lesley S.A., Elsliger M.A., Deacon A.M., Wilson I.A. Structural insights into the recognition of phosphopeptide by the FHA domain of kanadaptin. PLoS ONE, 2014, 9(9): e107309 (doi: 10.1371/journal.pone.0107309).
- Tsunoda T., Doi K., Ishikura S., Luo H., Nishi K., Matsuzaki H., Koyanagi M., Tanaka Y., Okamura T., Shirasawa S. Zfat expression in ZsGreen reporter gene knock in mice: Implications for a novel function of Zfat in definitive erythropoiesis. International Journal of Molecular Medicine,2018, 42(5): 2595-2603 (doi: 10.3892/ijmm.2018.3806).
- Tsunoda T., Takashima Y., Tanaka Y., Fujimoto T., Doi K., Hirose Y., Koyanagi M., Yoshida Y., Okamura T., Kuroki M., Sasazuki T., Shirasawa S. Immune-related zinc finger gene ZFAT is an essential transcriptional regulator for hematopoietic differentiation in blood islands. Proceedings of the National Academy of Sciences, 2010, 107(32): 14199-14204 (doi: 10.1073/pnas.1002494107).
- Ji H.Y., Yang B., Zhang Z.Y., Ouyang J., Yang M., Zhang X.F., Zhang W.C., Su Y., Zhao K.W., Xiao S.J., Yan X.M., Ren J., Huang L.S. A genome-wide association analysis for susceptibility of pigs to enterotoxigenic Escherichia coli F41. Animal,2016, 10(10): 1602-1608 (doi: 10.1017/S1751731116000306).
- Tissir F., Qu Y., Montcouquiol M., Zhou L., Komatsu K., Shi D., Fujimori T., Labeau J., Tyteca D., Courtoy P., Poumay Y., Uemura T., Goffinet A.M. Lack of cadherins Celsr2 and Celsr3 impairs ependymal ciliogenesis, leading to fatal hydrocephalus. Nat. Neurosci., 2010, 13: 700-707 (doi: 10.1038/nn.2555).
- Schäfer M., Bräuer A.U., Savaskan N.E., Rathjen F.G., Brümmendorf T. Neurotractin/kilon promotes neurite outgrowth and is expressed on reactive astrocytes after entorhinal cortex lesion. Molecular and Cellular Neuroscience, 2005, 29(4): 580-590 (doi: 10.1016/j.mcn.2005.04.010).
- Lee K.-T., Byun M.-J., Kang K.-S., Park E.-W., Lee S.-H., Cho S., Kim H.Y., Kim K.-W., Lee T.H., Park J.-E., Park W.C., Shin D.H., Park H.-S., Jeon J.-T., Choi B.-H., Jang G.-W., Choi S.-H., Kim D.-W., Lim D., Park H.-S., Park M.-R., Ott J., Schook L. B., Kim T.-H., Kim H. Neuronal genes for subcutaneous fat thickness in human and pig are identified by local genomic sequencing and combined SNP association study. PLoS ONE, 2011, 6(2): e16356 (doi: 10.1371/journal.pone.0016356).
- Stockis J., Colau D., Coulie P.G., Lucas S. Membrane protein GARP is a receptor for latent TGF-b on the surface of activated human Treg. Eur. J. Immunol., 2009, 39(12): 3315-3322 (doi: 10.1002/eji.200939684).
- Kosova G., Scott N.M., Niederberger C., Prins G.S., Ober C. Genome-wide association study identifies candidate genes for male fertility traits in humans. The American Journal of Human Genetics, 2012, 90(6): 950-961 (doi: 10.1016/j.ajhg.2012.04.016).
- Hoofnagle M.H., Neppl R.L., Berzin E.L., Teg Pipes G.C., Olson E.N., Wamhoff B.W., Somlyo A.V., Owens G.K. Myocardin is differentially required for the development of smooth muscle cells and cardiomyocytes. American Journal of Physiology-Heart and Circulatory Physiology, 2011, 300(5): H1707-H1721 (doi: 10.1152/ajpheart.01192.2010).
- Lin C.-C., Huang C.-C., Lin K.-H., Cheng K.-H., Yang D.-M., Tsai Y.-S., Ong R.-Y., Huang Y.N., Kao L-.S. Visualization of Rab3A dissociation during exocytosis: a study by total internal reflection microscopy. J. Cell. Physiol., 2007, 211(2): 316-326 (doi: 10.1002/jcp.20938).
- Laurin M., Fradet N., Blangy A., Hall A., Vuori K., Cóté J.-F. The atypical Rac activator Dock180 (Dock1) regulates myoblast fusion in vivo. Proceedings of the National Academy of Sciences, 2008, 105(40): 15446-15451 (doi: 10.1073/pnas.0805546105).
- Yang Y., Zhou R., Mu Y., Hou X., Tang Z., Li K. Genome-wide analysis of DNA methylation in obese, lean, and miniature pig breeds. Scientific Reports, 2016, 6: 30160 (doi: 10.1038/srep30160).
- Ponsuksili S., Zebunke M., Murani E., Trakooljul N., Krieter J., Puppe B., Schwerin M., Wimmers K. Integrated Genome-wide association and hypothalamus eQTL studies indicate a link between the circadian rhythm-related gene PER1 and coping behavior. Scientific Reports, 2015, 5: 16264 (doi: 10.1038/srep16264).
- Белоус А.А., Сермягин А.А., Костюнина О.В., Brem G., Зиновьева Н.А. Изучение генетической архитектуры конверсии корма у хряков (Sus scrofa) породы дюрок на основе полногеномного анализа SNP. Сельскохозяйственная биология, 2019, 54(4): 705-712 (doi: 10.15389/agrobiology.2019.4.705rus).