doi: 10.15389/agrobiology.2022.1.131rus
УДК 579.64:632.4.01/.08
Исследование выполнено при финансовой поддержке гранта РФФИ № 19-04-00642 А.
ПЕРВОЕ ВЫЯВЛЕНИЕ ГРИБА Fusarium coffeatum НА ТЕРРИТОРИИ РОССИЙСКОЙ ФЕДЕРАЦИИ
Л.П. МИНАЕВА1, Л.В. САМОХВАЛОВА2, С.К. ЗАВРИЕВ2, А.А. СТАХЕЕВ2 ✉
Согласно данным Росстата, доля зерновых и зернобобовых культур в структуре сельскохозяйственного производства в России в 2014-2018 годах колебалась от 17,2 до 20,2 %, а их валовые сборы в тот же период — от 105,2 до 135,5 млн т. При этом острой остается проблема заражения зерна и продуктов его переработки фитопатогенами различной природы, в том числе токсигенными грибами рода Fusarium. В комплексе мер по противодействию распространения фузариозной инфекции важнейшую роль играет оценка видового состава грибов, поражающих сельскохозяйственные культуры в разных регионах. Для надежной видовой идентификации широко используется комплексный подход, включающий, помимо традиционных микробиологических методов, анализ нуклеотидных последовательностей маркерных генов и их сравнение со стандартными последовательностями, представленными в базах данных. Настоящая работа посвящена описанию изолята гриба рода Fusarium (штамм ION-3/4), выделенного из зерна пшеницы в Тульской области Российской Федерации (2014 год). Выделение и очистку ДНК из мицелия исследуемого изолята и референтных культур видов Fusarium equiseti и F. graminearum проводили с использованием набора DNAeasy® Plant Pro Kit («Qiagen», Германия). Секвенирование маркерных фрагментов генов фактора элонгации трансляции 1 альфа (TEF1a, размер фрагмента 587 п.н.) и субъединицы РНК-полимеразы II (RPB2, размер фрагмента 689 п.н.) проводили модифицированным методом Сэнгера с использованием флуоресцентной метки на автоматическом секвенаторе ABI PRISM 3730 («Applied Biosystems», США). Для анализа расшифрованных нуклеотидных последовательностей генов TEF1aи RPB2 использовали алгоритм BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Филогенетический анализ и построение филогенетических деревьев выполняли в программе MEGA-X (https://www.megasoftware.net/) с использованием метода максимального правдоподобия и двухпараметрической модели Кимуры. Макроморфологические характеристики изолята изучали при культивировании на специальных питательных средах, микроморфологические структуры оценивали с использованием микроскопа Olympus CX33 («Olympus Corporation», Япония). Совокупность полученных данных позволила охарактеризовать штамм ION-3/4 как представителя вида Fusariumcoffeatum, недавно выделенного в качестве отдельной таксономической группы, входящей в состав комплекса видов F. incarnatum-equiseti . Филогенетически исследуемый изолят (штамм ION-3/4) формирует отдельную группу с типовым штаммом F. coffeatum 635.76. Ключевые морфологические характеристики (тип мицелия, форма и размер конидий, строение моно- и полифиалид) также соответствовали описанию характерных признаков этого вида. Поскольку вид F. coffeatum входит в комплекс видов F. incarnatum-equiseti, включающий возбудителей болезней растений, многие из которых способны к синтезу микотоксинов, то он также может рассматриваться как потенциальный фитопатоген, требующий пристального внимания и дальнейшего изучения.
Ключевые слова: Fusarium coffeatum, комплекс видов Fusariumin carnatum-equiseti, филогенетический анализ, ДНК-маркеры, морфология.
Видовая идентификация грибов рода Fusarium, особенно при работе с близкородственными видами, зачастую затруднена вследствие высокого сходства ключевых морфологических структур. Подтверждением этому может служить тот факт, что за историю изучения рода Fusarium было предложено около 10 таксономических систем, основанных на анализе морфологических характеристик, в которых число видов колебалось от 9 до 75 (1-4). Существенное значение в разрешении этих проблем на сегодняшний день имеют молекулярно-генетические методы, основанные прежде всего на секвенировании коротких маркерных участков ДНК («штрихкодов») (5, 6), филогенетическом анализе и ПЦР-идентификации. Наиболее надежным и достоверным методом молекулярной таксономии можно считать филогенетическое распознавание вида на основе генеалогического соответствия (Genealogical Concordance Phylogenetic Species Recognition, GCPSR) (7-10). Этот метод применяется для установления границ видов и основан на мультилокусном филогенетическом анализе. В качестве филогенетически информативных ДНК-маркеров для грибов рода Fusarium чаще всего используются гены TEF1a (ген фактора элонгации трансляции 1 альфа) (11) и RPB2 (ген субъединицы РНК-полимеразы II) (12). При этом необходимо отметить, что молекулярно-генетические методы не могут и не должны рассматриваться в качестве замены классическим микробиологическим процедурам, а быть их разумным дополнением, существенно расширяющим арсенал аналитических возможностей специалиста. Примерами удачного комбинирования микробиологических и молекулярных подходов служат работы, посвященные обнаружению на территории России ранее не выявленных видов F. torulosum (13) и F. globosum (14).
С конца 1990-х годов применение мультилокусного анализа позволило провести более глубокую видовую дифференциацию, в результате чего многие морфологически схожие изоляты (штаммы) Fusarium, рассматриваемые до этого как представители одного вида, были ре-классифицированы и отнесены к группе или комплексу видов (species complex) (15). Одним из наиболее крупных и интенсивно изучаемых является комплекс видов Fusarium incarnatum-equiseti (FIESC) (16), на сегодняшний день включающий более 30 филогенетических видов, относящихся к двум кладам — F. equiseti и F. incarnatum (17-20). Интерес, который вызывают у исследователей представители FIESC, обусловлен их высокой генетической вариабельностью, экологической пластичностью, а также способностью к синтезу широкого спектра вторичных метаболитов, в том числе микотоксинов (21).
В 2014 году в рамках ежегодного мониторинга загрязненности зернового сырья микотоксинами, а также изучения их продуцентов, проводимого в ФГБУН ФИЦ питания и биотехнологии, из зерна пшеницы, выращенного в Тульской области, был выделен изолят, получивший рабочее название ION-3/4. Предварительный анализ морфологических особенностей культуры не позволил однозначно определить его видовую принадлежность, что стало основанием для последующих исследований с привлечением молекулярно-генетических инструментов.
Цель настоящей работы — установление видового статуса изолята ION-3/4 c применением комплексного подхода, включающего анализ нуклеотидных последовательностей генов-маркеров, а также расширенное изучение культуральных свойств, макро- и микро-морфологических характеристик.
Методика. Исследуемый изолят грибов рода Fusarium штамм ION-3/4 был выделен из мицелия, выросшего непосредственно из зерновки при микологическом анализе зерна. Далее в результате последовательных пересевов был получен моноспоровый изолят. В настоящее время культура штамма ION-3/4 хранится в коллекции микроорганизмов в лаборатории биобезопасности и анализа нутримикробиома (ФГБУН ФИЦ питания и биобезопасности).
Для проведения молекулярно-генетических исследований и филогенетического анализа, помимо штамма ION-3/4, использовали другие морфологически близкие виды рода Fusariumштаммы: F. equiseti 64803 и 97001 (коллекция ФГБНУ ВИЗР, г. Санкт-Петербург—Пушкин), F. incar-natum F-2681 (коллекция ФИЦ ПНЦБИ, г. Пущино) и F. graminearum F-892 (коллекция НИЦ Курчатовский институт — ГосНИИгенетика, г. Москва), видовая принадлежность которых была подтверждена в предыдущем иссле-довании (22).
ДНК из моноспоровых культур грибов выделяли с использованием набора реагентов DNAeasy® Plant Pro Kit («Qiagen», Германия) в соответствии с протоколом производителя. Концентрации и степень очистки ДНК от белковых и низкомолекулярных примесей определяли с помощью спектрофотометра NanoVue («GE HealthCare», США).
Дизайн универсальных праймеров для секвенирования маркерных участков проводили с помощью выравнивания последовательностей нуклеотидов генов TEF1a и RPB2 грибов рода Fusarium, депонированных в базе данных Национального центра биотехнологической информации (GenBank, http://www.ncbi.nlm.nih.gov/GenBank). Для выравнивания применяли алгоритм ClustalW (23). Расчет температуры отжига и оценку физико-химических свойств олигонуклеотидов проводили с использованием программы Oligo.6.71 (https://www.oligo.net/). В результате проведенного анализа были сконструированы праймеры следующей структуры: TEF30F — 5´-CGTCGTCATCGGCCACGT-3´, TEF650R — 5´-ACCAATGACRGTGACATAGTAGC-3´; RPB2F — 5´-ATGRTCMRCMGAGGYATGGAAGT-3´, RPB2R — 5´- TTGTGATCGGGGAADGGA-3´.
ПЦР проводили в амплификаторе Терцик («ДНК-технология», Россия). Для пары TEF30F-650R использовалась следующая программа амплификации: 93 °С , 90 с (1 цикл); 93 °С, 20 с, 64 °С, 5 с, 67 °С, 5 с (5 циклов); 93 °С, 1 с, 64 °С, 5 с, 67 °С, 5 с (40 циклов); для пары RPB2F-2R — 93 °С, 90 с (1 цикл); 93 °С, 5 с, 60 °С, 10 c, 72 °С, 5 c (45 циклов). Использовали набор реагентов для проведения ПЦР, в том числе Taq полимеразу UP с активностью 5 ед/мкл (ООО «ДНК-технология», Россия). Состав ПЦР-буфера и концентрации компонентов реакционной смеси описаны ранее (24).
Продукты ПЦР клонировали в плазмидный вектор pAL2-T с использованием набора Quick-TA kit (ЗАО «Евроген», Россия) в соответствии с протоколом производителя. Секвенирование проводили модифицированным методом Сэнгера с использованием флуоресцентной метки на автоматическом секвенаторе ABI PRISM 3730 («Applied Biosystems», США; секвенирование выполнено в ЗАО «Евроген»). Для анализа маркерных последовательностей генов TEF1a и RPB2 и их сравнения с базой данных NCBI (https://www.ncbi.nlm.nih.gov/genome/) использовали алгоритм BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Филогенетический анализ выполняли в программе MEGA-X (25) с применением метода максимального правдоподобия (ML) и двухпараметрической модели Кимуры (26). Достоверность топологий филогенетических деревьев подтверждали с помощью бутстреп-анализа с 1000 повторностей. В анализ были включены последовательности генов TEF1a и RPB2 грибов рода Fusarium, депонированных в базе данных NCBI: F. equiseti NRRL 264 919 (GQ505599, GQ505777), F. scirpiNRRL 36478 (GQ505654, GQ505832), F. compactum NRRL 36323 (GQ505648, GQ505826), F. lacertarum NRRL 20434 (GQ505593, GQ505771), F. incarnatum CBS 132.73 (MN170476, MN170409). Также использовались последовательности типового штамма F. coffeatum CBS 635.76 (MN120736, MN120755).
Макро- и микроморфологические характеристики моноспорового изолята ION-3/4 исследовали при выращивании на нескольких видах пи-тательных сред: картофельно-сахарозном агаре (КСА)(1), картофельно-декстрозном агаре (PDA) («Himedia Laboratories Pvt Ltd.», Индия; «Neogen Corporation», США), агаре Чапека-Докса (CDA) («Himedia Laboratories Pvt Ltd.», Индия), овсяном агаре (ОА) (Difco Laboratories, «Fisher Scientific», США), гвоздично-листовом агаре (CLA)(1) и на среде Ниренберга (SNA) (3). Макроморфологические особенности (структура мицелия и пигментация) оценивали на средах КСА, PDA, ОА и CDA на 7-е сут роста, в условиях темноты и переменного освещения (16 ч день/8 ч ночь) при температуре 25 °С, изменение пигментации наблюдали до 3 нед. Скорость горизонтального роста изолята исследовали при культивировании на средах PDA, CDA, OA и SNA в чашках Петри при температурах 20, 25 и 30 °С. Диаметр колоний на 3-и, 5-е и 7-е сут измеряли в двух поперечных направлениях для трех параллельных посевов (n= 3). Рассчитывали средние значения (М), стандартное отклонение по результатам всех измерений составляло 5,3 %, вычисления выполняли с использованием программного обеспечения Microsoft Office Excel. Микроморфологические характеристики (форма и размеры конидий, типы фиалид) исследовали при культивировании на среде CLA при 25 °С на 7-14-е сут с помощью микроскопа Olympus CX33 («Olympus Corporation», Япония). Размеры конидий представлены в виде среднего и диапазонов от наименьшего до наибольшего размера (min-max) в продольном и поперечном направлениях, полученные не менее чем для 10 измерений, выполненных с использование программного обеспечения ToupView http://toup-tek.com/product/showproduct.php?lang=en&id=103.
Морфологические особенности штамма ION-3/4 сопоставляли с описаниями грибов рода Fusarium, представленными в определителях (2, 3), а также в публикациях (20, 27).
Результаты. Анализ нуклеотидных последовательностей участков двух маркерных генов показал 100 % (TEF1a) и 99,45 % (RPB2) сходства с соответствующими последовательностями типового штамма F. coffeatum CBS 635.76. Исследование топологии филогенетических деревьев, представленных на рисунке 1, показало, что штаммы ION-3/4 и CBS 635.76 формируют отдельный кластер с бутстреп-поддержкой 100 % и 99 % соответственно для генов TEF1a и RPB2. Также важно отметить тот факт, что кластер F. cof-featumпринадлежал к филогенетической кладе F. incarnatum, что согласуется с данными, опубликованными ранее (20, 27).
Макроморфологические характеристики штамма ION-3/4 изучали при культивировании на питательных средах КСА, PDA, ОА и CDA. На средах КСА и PDA отмечено формирование обильного плотного невысокого воздушного мицелия молочно-белого цвета с легким кремовым оттенком, реверс имел более насыщенный кремово-персиковый цвет, по мере старения культуры (более 2 нед) появлялся более выраженный желтый цвет, который потом переходил в светло-коричневый — «кофе с молоком» (рис. 2, А). При культивировании на питательных средах на основе картофельного экстракта разных фирм-изготовителей варьировала насыщенность цвета реверса. Более светлый реверс отмечали при росте на среде КСА, приготовленной в лабораторных условиях из картофельного отвара согласно рецептуре (2), наиболее интенсивную яркую желто-оранжевую ок-раску колонии приобретали на среде PDA производства «Himedia Laboratories Pvt Ltd.». Выращивание в условиях переменного освещения (16 ч день/8 ч ночь) на средах КСА и PDA способствовало формированию более насыщенной цветовой палитры реверса в отличие от условий без освещения, воздушный мицелий всегда оставался молочно-белым независимо от освещения. При росте на среде ОA воздушный мицелий был менее плотным, а на среде CDA имел рыхлую, хлопьевидную структуру, менее развитую, чем на PDA. Цвет воздушного мицелия и реверса на средах ОA и CDA оста-вался молочно-белым (см. рис. 2, Б).
Микроморфологические структуры оценивали при культивировании на среде CLA при 25 °С на 7-14-е сут. В воздушном мицелии культура образовывала обильные микро- и мезоконидии (рис. 3), макроконидии встре-чались редко. Размеры конидий приведены в таблице 1. Образования спородохий не наблюдали.
Макроконидии, образуемые на CLA, в основном с двумя перегородками, почти прямые, с небольшим изгибом с дорсальной стороны, концевые клетки имели слабо выраженную форму (в апикальной части в виде сосочка, в базальной — в виде ступни).
В воздушном мицелии наблюдалось обилие веретеновидных мезоконидий без перегородки или с одной перегородкой (см. рис. 3, д), а также микроконидий овальной или обратнояйцевидной формы (см. рис. 3, а-г, е), формируемых в молодых культурах на монофиалидах (см. рис. 3, б, е), а по мере роста — также на полифиалидах (см. рис. 3, а, в, г). Полифиалиды имели два и более локусов для образования конидий. Размер фиалид варьировал от 6 до 60 мкм (см. рис. 3, а-г, е). Хламидоспоры не были обнаружены.
При сравнении с морфологически близким видом F. chlamydosporum Wollenweber & Reinking (3) у изолята F. coffeatum (штамм ION-3/4) отмечается сходство по снижению или отсутствию формирования спородохий и макроконидий в них, по форме конидиогенных клеток (моно- и полифиалиды), обилию и форме микроконидий, образованию белого воздушного мицелия, однако у культур ION-3/4 отсутствуют розовый и бордовый цвет реверса.
По обилию прямых веретенообразных мезоконидий в воздушном мицелии на моно- и полифиалидах изолят F. coffeatum (штамм ION-3/4) схож с F. semitectum Berkeley & Ravenel (син. F. incarnatum) (3), также их объединяет пониженное образование или отсутствии хламидоспор, что, как и образование спородохий, является штамм-специфичным признаком. Однако, в отличие от F. semitectum, изолят F. coffeatum (штамм ION-3/4) обильно образует микроконидии в молодой культуре, тогда как у F. semitectum это отмечают в старых культурах (3), а появление бежевого или коричневого цвета реверса наблюдается только в старой культуре.
Полученные характеристики для изучаемого изолята (штамм ION-3/4) согласуются с описанием типичного штамма F. coffeatum (CBS 635.76), приведенным ранее (27). Это касается в первую очередь микроморфологических характеристик (форма и размеры образуемых в воздушном мицелии конидий, строение моно- и полифиалид, а также отсутствие спородохий на CLA). При этом у указанного штамма F. coffeatum (CBS 635.76) утрачена способность продуцировать пигменты (от бежевого до кофейно-коричневого цвета) на средах PDA и OA, а на CLA отсутствуют спородохии, что, по мнению авторов, является следствием его вырождения. У полученного нами изолята F. coffeatum (штамм ION-3/4), как было описано выше, пигмент образуется на средах PDA, но интенсивность пигментации варьирует на средах разных фирм-изготовителей. В настоящее время в научных публикациях отсутствует более подробное описание F. coffeatum.
Скорость роста культуры штамма ION -3/4 изучали в течение 7 сут при культивировании в чашках Петри на четырех видах питательных сред при 20, 25 и 30 °С (табл. 2).
Набольшую скорость роста наблюдали при 25 °С: на 7-е сут колонии достигали максимального размера (86 мм на средах PDA, CDA, OA, 80 мм — на SNA). При 20 °С и 30 °С культура росла значительно медленнее на средах PDA, OA и SNA, при этом на среде CDA размер колонии при 30 °С был таким же, как при 25 °С, а при 20 °С несколько меньше. Таким образом, выявлено, что температура 25 °С наиболее благоприятна для активного роста изолята F. coffeatum (штамм ION-3/4) на всех видах использованных питательных средах.
Итак, основной результат настоящего исследования — первое обнаружение штамма гриба Fusarium coffeatum на территории России, а также подробное описание его фенотипических характеристик. Согласно данным GenBank (NCBI), до настоящего времени моноспоровые изоляты F. coffeatum были выделены на территории Австралии (FIESC28_10703), Южной Африки (типовой штамм CBS 635.76), Румынии (CBS 430.81). Исходя из полученных данных, можно отметить, что указанный вид является космополитичным и обладает широкой экологической пластичностью, позволяющей ему существовать в различных климатических условиях. Выявление F.coffeatum расширяет представления о видовом разнообразие грибов рода Fusarium на территории Российской Федерации и дополняет новыми знаниями о географических ареалах распространения этого вида. Однако пока не ясно, способен ли вид F. coffeatum заражать сельскохозяйственные культуры и синтезировать микотоксины. Изучение этих аспектов может стать целью будущих исследований.
1. Размеры (мкм) конидий Fusarium coffeatum (штамм ION-3/4), формируемых на гвоздично-листовом агаре (CLA) (n = 10) |
|||
Тип конидий |
Длина (Д), среднее (min-max) |
Ширина (Ш), среднее (min-max) |
Соотношение Д/Ш |
Микроконидии |
6,7 (6,0-7,4) |
1,6 (1,0-2,1) |
4,2 |
Мезоконидии: |
|
|
|
без перегородки: |
15,7 (8,6-16,0) |
2,7 (2,3-3,1) |
5,8 |
с одной перегородкой |
15,2 (9,4-21,0) |
2,4 (2,0-2,7) |
6,3 |
Макроконидии (с двумя перегородками) |
24,5 (24,0-25,0) |
3,8 (3,5-4,0) |
7,3 |
2. Динамика радиального роста колоний Fusarium coffeatum (штамм ION-3/4) (мм) на питательных средах при разных температурах (n = 3, M±5,3 %) |
||||||||||||
Время, сут |
PDA |
CDA |
OA |
SNA |
||||||||
20 °С |
25 °С |
30°С |
20 °С |
20 °С |
30 °С |
20 °С |
25 °С |
30 °С |
20 °С |
25 °С |
30 °С |
|
3-и |
22 |
35 |
23 |
30 |
40 |
35 |
18 |
32 |
23 |
23 |
30 |
17 |
5-е |
50 |
65 |
52 |
60 |
77 |
72 |
45 |
72 |
60 |
46 |
53 |
40 |
7-е |
75 |
86 |
75 |
80 |
86 |
86 |
67 |
86 |
70 |
64 |
80 |
60 |
Примечание. PDA — картофельно-декстрозный агар («Himedia Laboratories Pvt Ltd.», Индия), CDA — агар Чапека-Докса («Himedia Laboratories Pvt Ltd.», Индия), OA — овсяный агар (Difco Laboratories, «Fisher Scientific», США), SNA — среда Ниренберга (3). |
||||||||||||
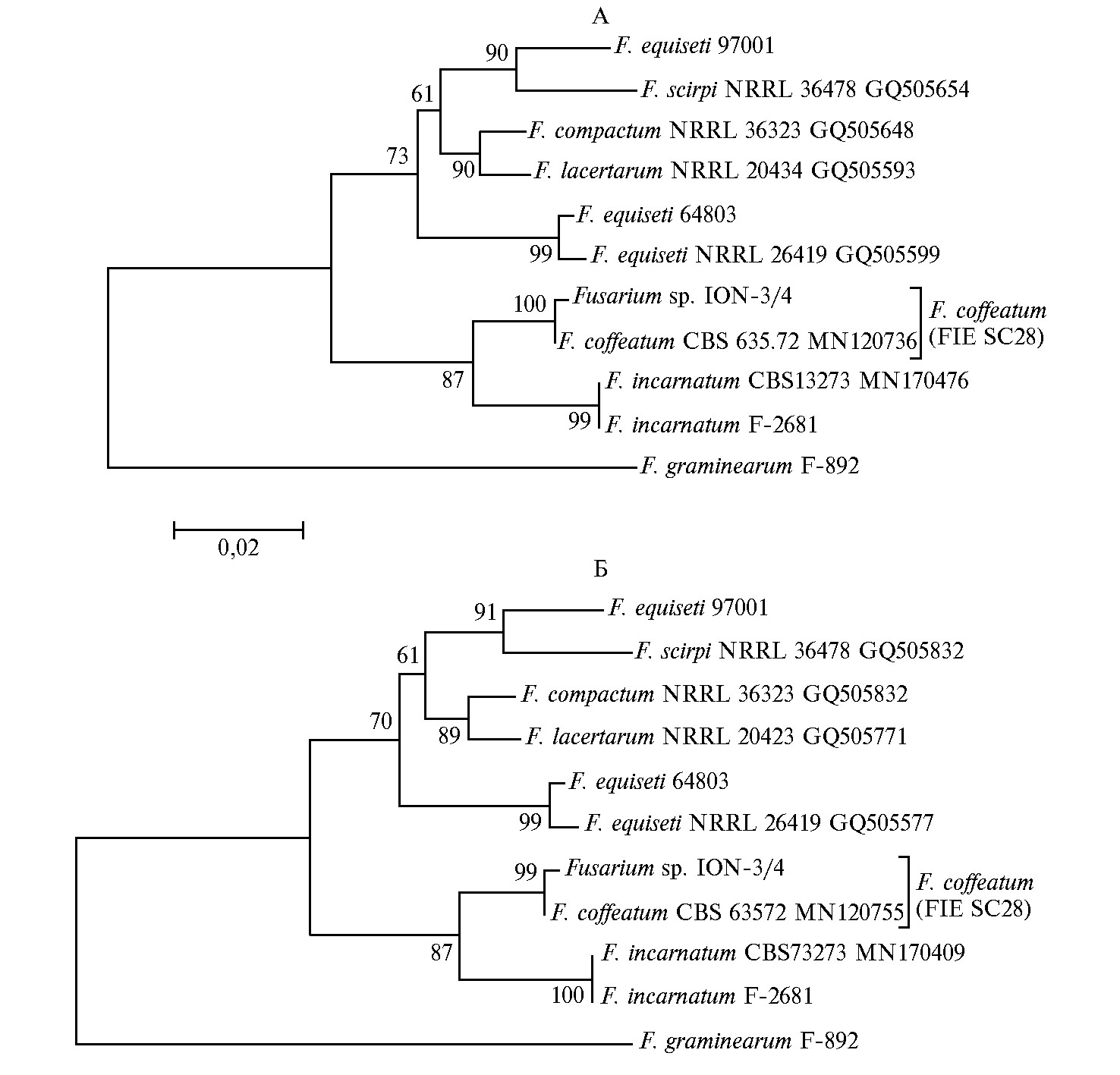
Рис. 1. Филогенетические деревья, построенные на основе сравнения маркерных последовательностей гена TEF1a (А) и RPB2(Б) изолята ION-3/4 и референтных штаммов семи видов грибов рода Fusarium c помощью метода максимального правдоподобия (ML) и двухпараметрической модели Кимуры (26) в программе MEGA-X (25). Показаны только значения бутстрепа более 50 % для 1000 повторностей. Последовательности штамма F. graminearum F-892 использованы в качестве «внешней группы».
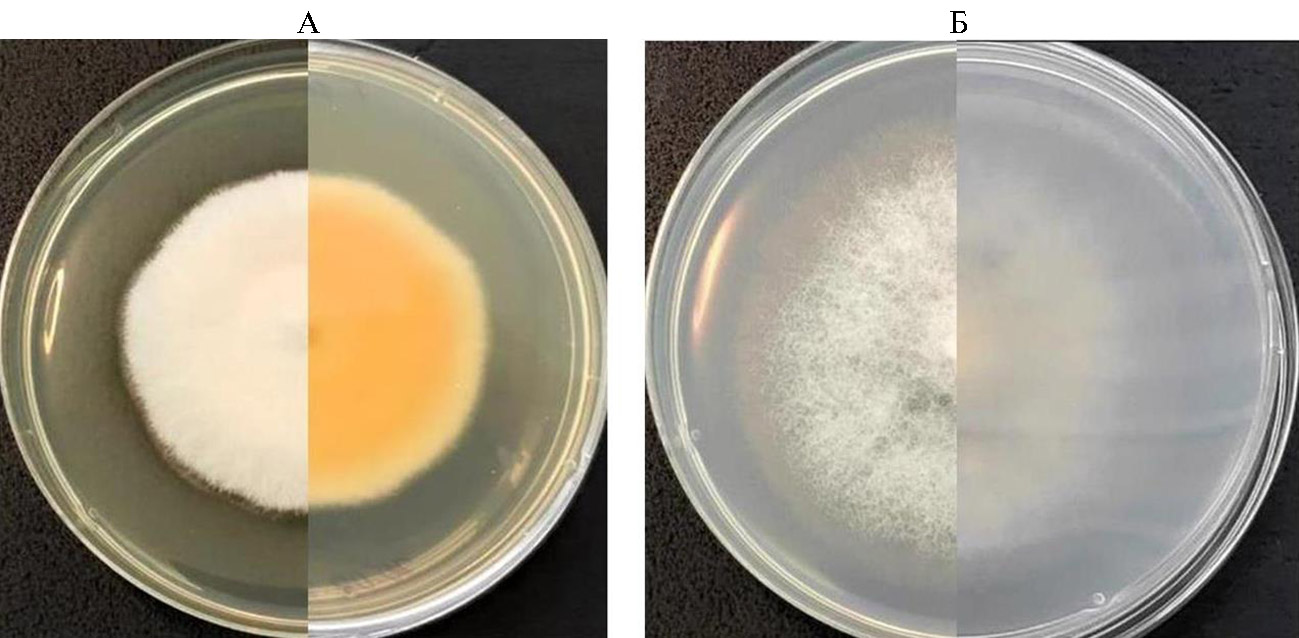
Рис. 2. Fusarium coffeatum (штамм ION-3/4) на 4-е сут культивирования при 25 °С на картофельно-сахарозном агаре (КСА) (А) и агаре Чапека-Докса (CDA) (Б).
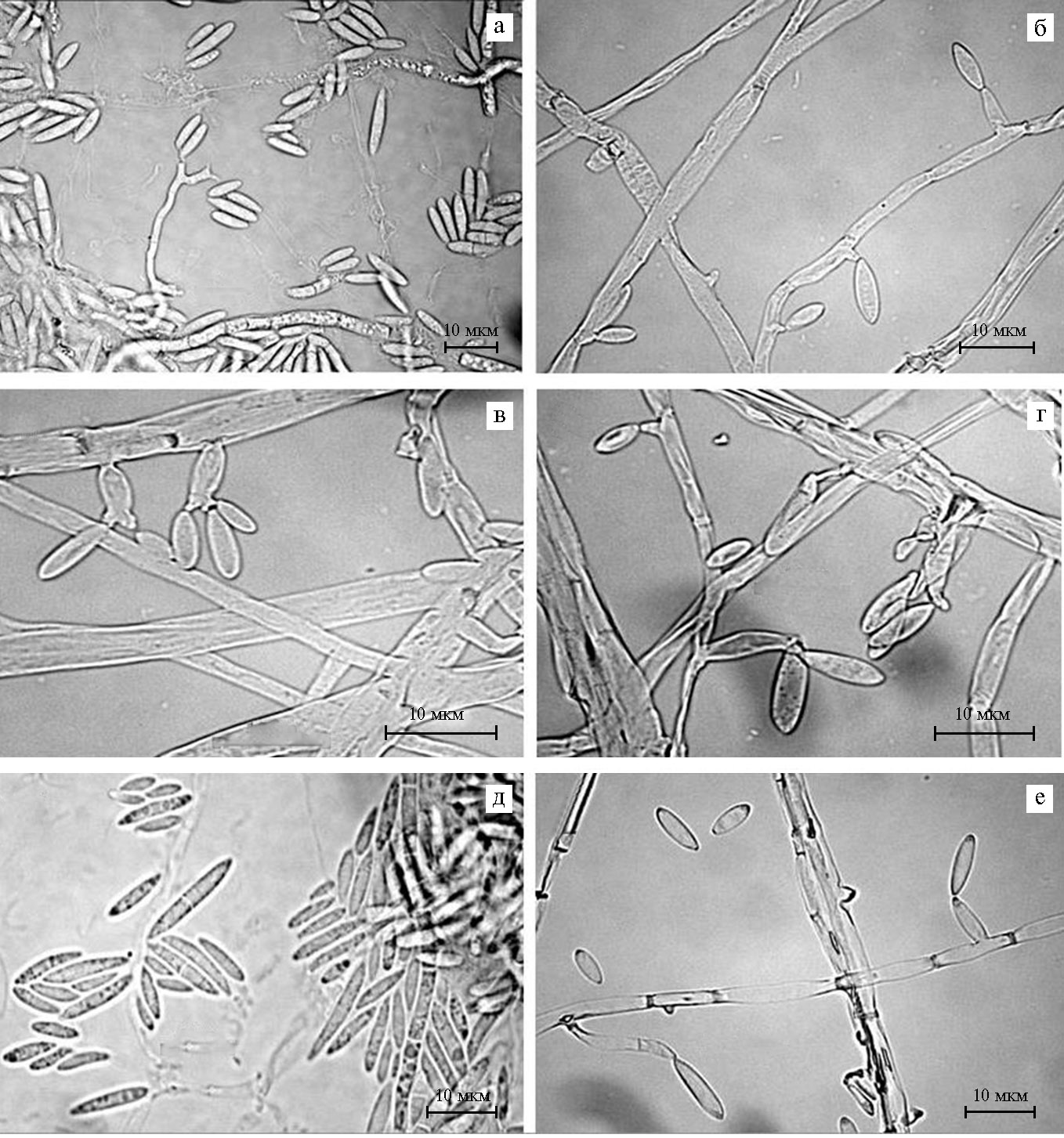
Рис. 3. Fusarium coffeatum (штамм ION-3/4) — микро- и мезоконидии, формируемые в воздушном мицелии на моно- и полифиалидах на гвоздично-листовом агаре (CLA): а, в — микроконидии на полифиалидах; б, е — микроконидии на монофиалидах; г — микроконидии на моно- и полифиалидах; д — микро- и мезоконидии (микроскопа Olympus CX33, «Olympus Corporation», Япония).
ЛИТЕРАТУРА
- Гагкаева Т.Ю., Гаврилова О.П., Левитин М.М., Новожилов К.В. Фузариоз зерновых культур. Защита и карантин растений, 2011, 5: 69-120.
- Gerlach W., Nirenberg H.I. The genus Fusarium — a pictorial atlas. Kommissionsverlag P. Parey, Berlin, 1982.
- The Fusarium laboratory manual /J.F. Leslie, B.A. Summerell. Blackwell Publishing, 2006 (doi: 10.1002/9780470278376).
- O’Donnell K., McCormick S.P., Busman M., Proctor R.H., Ward T.J., Doehring G., Geiser D.M., Alberts J.F., Rheeder J.P. Marasas et al. «Toxigenic Fusarium species: identity and mycotoxicology» revisited. Mycologia, 2018, 110(6): 1058-1080 (doi: 10.1080/00275514.2018.1519773).
- Шнеер В.С. ДНК-штрихкодирование видов животных и растений — способ их молекулярной идентификации и изучения биоразнообразия. Журнал общей биологии, 2009, 70(4): 296-315.
- Щеховцов С.В., Шеховцова И.Н., Пельтек С.Е. ДНК-штрихкодирование: методы и подходы. Успехи современной биологии, 2019, 139(3): 211-220 (doi: 10.1134/S0042132419030074).
- Taylor J.W., Jacobson D.J., Kroken S., Kasuga T., Geiser D.M., Hibbett D.S., Fisher M.C. Phylogenetic species recognition and species concepts in Fungi. Fungal Genetics and Biology, 2000, 31, 21-32 (doi: 10.1006/fgbi.2000.1228).
- O’Donnell K., Humber R.A., Geiser D.M., Kang S., Park B., Robert V.A.R.G., Crous P.W., Johnston P.R., Aoki T., Rooney A.P., Rehner S.A. Phylogenetic diversity of insecticolous fusaria inferred from multilocus DNA sequence data and their molecular identification via FUSARIUM-ID and Fusarium MLST. Mycologia, 2012, 104(2): 427-445 (doi: 10.3852/11-179).
- O’Donnell K., Ward T.J., Robert V.A.R.G., Crous P.W., Geiser D.M., Kang S. DNA sequence-based identification of Fusarium: current status and future directions. Phytoparasitica, 2015, 43: 583-595 (doi: 10.1007/s12600-015-0484-z).
- Стахеев А.А., Самохвалова Л.В., Рязанцев Д.Ю., Завриев С.К. Молекулярно-генетические методы в исследовании таксономии и специфической идентификации токсинпродуцирующих грибов рода Fusarium: успехи и проблемы. Сельскохозяйственная биология, 2016, 51(3): 275-284 (doi: 10.15389/agrobiology.2016.3.275rus).
- Kristensen R., Torp M., Kosiak B., Holst-Jensen A. Phylogeny and toxigenic potential is correlated in Fusarium species as revealed by partial translation elongation factor 1 alpha gene sequences. Mycological Research, 2005, 109: 173-186 (doi: 10.1017/S0953756204002114).
- O’Donnell K., Rooney A.P., Proctor R.H., Brown D.W., McCormick S.P., Ward T.J., Frandsen R.J.N., Lysøe E., Rehner S.A., Aoki T., Robert V.A.R.G., Crous P.W., Groenewald J.Z., Kang S., Geiser D.M. Phylogenetic analyses of RPB1 and RPB2 support a middle Cretaceous origin for a clade comprising all agriculturally and medically important fusaria. Fungal Genetics and Biology, 2013, 52: 20-31 (doi: 10.1016/j.fgb.2012.12.004).
- Гагкаева Т.Ю., Гаврилова О.П., Стахеев А.А., Рязанцев Д.Ю., Завриев С.К. Первое обнаружение в России гриба Fusarium torulosum. Микологияифитопатология, 2012, 46(1): 86-91.
- Gagkaeva T., Gavrilova O., Orina A. First report of Fusarium globosum associated with barley grain in the southwestern part of Siberia. Plant Disease, 2019, 103(3): 588 (doi: 10.1094/PDIS-06-18-1108-PDN).
- O’Donnell K., Kistler H.C., Tacke B.K., Casper H.H. Gene genealogies reveal global phylogeographic structure and reproductive isolation among lineages of Fusarium graminearum, the fungus causing wheat scab. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(14): 7905-7910 (doi: 10.1073/pnas.130193297).
- O’Donnell K., Sutton D.A., Rinaldi M.G., Gueidan C., Crous P.W., Geiser D.M. Novel multilocus sequence typing scheme reveals high genetic diversity of human pathogenic members of the Fusarium incarnatum-F. equiseti and F. chlamydosporum species complexes within the United States. Journal of Clinical Microbiology, 2009, 47(12): 3851-3861 (doi: 10.1128/JCM.01616-09).
- Castellá G., Cabañes F.J. Phylogenetic diversity of Fusarium incarnatum-equiseti species complex isolated from Spanish wheat. Antonie van Leeuwenhoek, 2014, 106(2): 309-317 (doi: 10.1007/s10482-014-0200-x).
- Villani A., Moretti A., De Saeger S., Hab Z., Di Mavungu J.D., Soares C.M.G., Proctor R.H., Venâncio A., Lima N., Stea G., Paciolla C., Logrieco A.F., Susca A. A polyphasic approach for characterization of a collection of cereal isolates of the Fusarium incarnatum-equiseti species complex. International Journal of Food Microbiology, 2016, 234: 24-35 (doi: 10.1016/j.ijfoodmicro.2016.06.023).
- Ramdial H., Latchoo R.K., Hosein F.N., Rampersad S.N. Phylogeny and haplotype analysis of fungi within the Fusarium incarnatum-equiseti species complex. Phytopathology, 2017, 107: 109-210 (doi: 10.1094/PHYTO-05-16-0209-R).
- Xia J.W., Sandoval-Denis M., Crous P.W., Zhang X.G., Lombard L. Numbers to names — restyling the Fusarium incarnatum-equiseti species complex. Persoonia, 2019, 43: 186-221 (doi: 10.3767/persoonia.2019.43.05).
- Villani A., Proctor R.H., Kim H-S., Brown D.W., Logrieco A.F., Amatulli M.T., Moretti A., Susca A. Variation in secondary metabolite production potential in the Fusarium incarnatum-equiseti species complex revealed by comparative analysis of 13 genomes. BMC Genomics, 2019, 20: 314 (doi: 10.1186/s12864-019-5567-7).
- Стахеев А.А., Самохвалова Л.В., Звездина Ю.К., Завриев С.К. Филогенетический анализ и молекулярное типирование трихотеценпродуцирующих грибов рода Fusarium из российских коллекций. Acta Naturae, 2018, 10(2-37): 85-99.
- Saitou N., Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 1987, 4(4): 406-425 (doi: 10.1093/oxfordjournals.molbev.a040454).
- Stakheev A.A., Ryazantsev D.Yu., Gagkaeva T.Yu., Zavriev S.K. PCR detection of Fusarium fungi with similar profiles of the produced mycotoxins. Food Control, 2011, 22(3-4): 462-468 (doi: 10.1016/j.foodcont.2010.09.028).
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549 (doi: 10.1093/molbev/msy096).
- Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution, 1980, 16: 111-120 (doi: 10.1007/BF01731581).
- Lombard L., van Doorn R., Crous P.W. Neotypification of Fusarium chlamydosporum — a reappraisal of clinically important species complex. Fungal Systematics and Evolution, 2019, 4: 183-200 (doi: 10.3114/fuse.2019.04.10).