doi: 10.15389/agrobiology.2020.1.194rus
УДК 634.8:632.08:577.2
Исследования выполнены в рамках государственного задания ВННИИВиВ «Магарач» РАН № 0833-2015-0019.
РАЗРАБОТКА НАБОРА РЕАГЕНТОВ ДЛЯ ДИАГНОСТИКИ ФИТОПЛАЗМЫ Candidatus Phytoplasma solani — ВОЗБУДИТЕЛЯ ПОЧЕРНЕНИЯ ДРЕВЕСИНЫ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ
С.А. БЛИНОВА1, А.А. ШВАРЦЕВ1, С.В. СЫКСИН1, Г.Н. БОНДАРЕНКО2, И.Г. БАШКИРОВА2, С.М. ГОРИСЛАВЕЦ3,
В.И. РИСОВАННАЯ3, Е.П. СТРАНИШЕВСКАЯ3, В.А. ВОЛОДИН3, Я.И. АЛЕКСЕЕВ1, 3, 4
Представители Candidatus Phytoplasma (фитоплазмы) служат возбудителями более 600 заболеваний растений. На виноградных насаждениях Европы наибольший ущерб приносят возбудитель золотистого пожелтения винограда Candidatus Phytoplasma vitis Marzorati et al., 2006 и возбудитель почернения древесины Candidatus Phytoplasma solani Qualino et al., 2013. Сходство симптомов различных фитоплазмозов винограда друг с другом, а также с вирусными и бактериальными заболеваниями делает их визуальное определение до вида невозможным. Широкое распространение и высокая вредоносность фитоплазмозов требуют расширения исследований их эпифитотиологии. Впервые в России нами разработана тест-система для выявления фитопатогена Candidatus Phytoplasma solani и проведено ее сравнение с уже существующими системами диагностики по основным характеристикам. Нашей целью была разработка и апробация набора реагентов для выявления фитоплазмы Candidatus Phytoplasma solani, вызывающей заболевание почернения древесины винограда, методом полимеразной цепной реакции в реальном времени (ПЦР-РВ). В работе использовали образцы ДНК Candidatus Phytoplasma solani, полученные из коллекции Всероссийского центра карантина растений, и зараженный материал винограда. Фрагменты лозы, корней и листьев сортов Шардоне, Пино нуар и Бастардо магарачский с визуальными признаками заражения Candidatus Phytoplasma были отобраны в осенний период 2018 года с виноградных насаждений Южнобережного агроклиматического района полуострова Крым. Пробы состояли из листьев, фрагментов лозы и корней винограда. ДНК фитоплазмы выделяли согласно методике, рекомендованной EPPO, с модификациями, а также с помощью набора реагентов Цитосорб (ООО «Синтол», Россия). Классическую ПЦР и ПЦР-РВ проводили в реакционном буфере, в состав которого входили 3 мM MgCl2, 0,25 мM dNTP, 2,5 ед. SynTaq ДНК-полимеразы с антителами, ингибирующими активность фермента (ООО «Синтол», Россия). Для секвенирования ДНК Candidatus Phytoplasma solani сконструировали пару праймеров SolaSeq_F 5'-AACTTAACCTTTTAACTAGGGC-3' и SolaSeq_R 5'-CATCAAGGCATTTGCC-3'. Для определения аналитической чувствительности набора реагентов была создана векторная конструкция на основе плазмиды Pal2T(«Евроген», Россия) со вставкой целевой нуклеотидной последовательности Candidatus Phytoplasma solani размером 119 п.н. Специфичность набора реагентов проверяли на 37 образцах близкородственных и сопутствующих объектов. Разработанные праймеры на участок гена SecYпозволяли идентифицировать ДНК фитоплазмы Candidatus Phytoplasma solani всех штаммов, последовательности которых находились в базе данных GenBank NCBI Nucleotide на 16.01.2019. Нуклеотидная последовательность гена SecY, в отличие от рекомендованных для диагностики, имела более высокую специфичность и консервативность. Набор реагентов был испытан на разных приборах для ПЦР-РВ. Его аналитическая чувствительность составила не менее 15 копий на реакцию, аналитическая специфичность — 100 %. Ложноположительных результатов при анализе проб в присутствии ДНК других видов фитоплазм, а также сопутствующих микроорганизмов не выявлено. Ложноотрицательных результатов при анализе проб с наличием ДНК целевого организма также не получили. Разработанный набор был апробирован при анализе 194 образцов, предположительно зараженных Candidatus Phytoplasma solani, из шести различных мест произрастания. Во всех случаях специфичность выявления фитоплазмы, вызывающей почернение древесины, подтвердило секвенирование ДНК образцов, положительных по гену SecY. Таким образом, разработанный набор реагентов позволяет быстро, точно и с высокой чувствительностью проводить диагностику инфицированности растений фитоплазмой Candidatus Phytoplasma solani на всех этапах вегетации (в том числе тестировать посадочный материал) и может использоваться для полномасштабных скрининговых исследований.
Ключевые слова: фитоплазма, Candidatus Phytoplasma solani, виноград, ПЦР в реальном времени, диагностика, ПЦР-РВ тест-система, специфичность, чувствительность, воспроизводимость, повторяемость.
В 1967 году одноклеточные патогены растений, которые ранее ошибочно относили к вирусной группе «желтух» из-за схожести симптомов и невозможности культивирования на питательной среде (1-3), были классифицированы как микоплазмоподобные организмы в связи с морфологическим сходством и чувствительностью к тетрациклиновым антибиотикам (4-6). С развитием методов молекулярной биологии, базируясь на нуклеотидных последовательностях генов рибосомальной РНК (7, 8), эти микроорганизмы выделили в отдельную таксономическую группу с родовым названием Candidatus Phytoplasma (9, 10).
Фитоплазмы поражают широкий круг растений, в том числе такую важную сельскохозяйственную культуру, как виноград (11, 12). Наиболее вредоносны для винограда два вида фитоплазм: возбудитель золотистого пожелтения Candidatus Phytoplasma vitis Marzorati et al., 2006 (карантинный объект) (13, 16-18) и возбудитель почернения древесины Candidatus Phytoplasma solaniQualino et al., 2013 (19-21). Основное растение-хозяин для возбудителя золотистого пожелтения Candidatus Phytoplasma vitis — виноград европейско-западноазиатской группы, к которому относятся почти все культивируемые сорта винограда. Потеря урожая восприимчивых сортов может достигать 100 %, через 2-3 года после заражения такие растения отмирают.
Фитоплазма Candidatus Phytoplasma solani поражает также кукурузу сахарную, плодовые культуры и представителей семейства Пасленовые (Solanaceae). Многие дикорастущие растения, в частности крапива двудомная и вьюнок полевой, могут быть резерваторами фитоплазменной инфекции (22, 23). Распределение фитоплазмы в растениях не равномерно. В основном фитоплазма локализуется во флоэме, поэтому эта часть растения считается предпочтительной для выделения нуклеиновых кислот и последующей диагностики. Некоторое виды растений толерантны к заражению фитоплазмами, поэтому при отборе проб для анализа стоит уделять внимание сбору разнообразного материала (фрагментов корня, лозы, листовой пластинки) даже без визуальных признаков инфицирования.
При заражении фитоплазмами снижение урожая варьирует от незначительного до почти полной его потери (1, 12). Поражение винограда приводит к потерям урожая до 25-30 %, при зараженности 70 % виноградники полностью выкорчевывают. Распространенность фитоплазмозов винограда в районах возделывания культуры может достигать 70-80 %, а вредоносность — превышать 40-80 % (23).
По данным EPPO (European and Mediterranean Plant Protection Organization, https://gd.eppo.int), на 2 ноября 2019 года Candidatus Phytoplasma solaniраспространена в Западной и Восточной Европе повсеместно, кроме нескольких стран центральной и северной части Европы (24). При этом чтобы установить реальный объем пораженных виноградников на территории России и других стран бывшего СССР, данных недостаточно (11). Для проведения полноценных скрининговых исследований необходимо разработать специфичный и простой в использовании набор реагентов для идентификации фитоплазмы Candidatus Phytoplasma solani.
Впервые в России нами разработана ПЦР-РВ тест-система для выявления фитопатогена Candidatus Phytoplasma solani и проведено ее сравнение с уже существующими системами диагностики по основным характеристикам (специфичность, чувствительность, воспроизводимость, повторяемость).
Нашей целью была разработка и апробация набора реагентов для выявления фитоплазмы Candidatus Phytoplasma solani, вызывающей заболевание почернения древесины винограда, методом полимеразной цепной реакции в реальном времени (ПЦР-РВ).
Методика. В работе использовали образцы ДНК Candidatus Phytoplasma solani, полученные из коллекции Всероссийского центра карантина растений (ВНИИКР), и зараженный материал винограда, предоставленный Всероссийским национальным научно-исследовательским институтом виноградарства и виноделия «Магарач» (ВННИИВиВ «Магарач»). Фрагменты лозы, корней и листьев сортов Шардоне, Пино нуар и Бастардо магарачский с визуальными признаками заражения Candidatus Phytoplasma были отобраны в осенний период 2018 года с виноградных насаждений Южнобережного агроклиматического района полуострова Крым.
Образцы для выделения ДНК измельчали вручную и при помощи ротационного гомогенизатора Precellys Evolution («Bertin Technologies», Франция). ДНК фитоплазмы выделяли согласно методике, рекомендованной EPPO (24), с модификациями (25), а также с помощью набора реагентов Цитосорб (ООО «Синтол», Россия), принцип действия которого основан на лизисе клеток хаотропным агентом гидрохлоридом гуанидина с последующей сорбцией ДНК на кремниевых частицах согласно описанию в заявке на патент RU № 2019111081/10 (021521).
Классическую ПЦР и ПЦР-РВ проводили в реакционном буфере, в состав которого входили 3 мM MgCl2, 0,25 мM dNTP, 2,5 ед. SynTaq ДНК-полимеразы с антителами, ингибирующими активность фермента (ООО «Синтол», Россия). При подборе праймеров учитывали следующие параметры: присутствие на 3'-конце праймера G или C нуклеотида («GC-зажим»), средняя температура отжига праймеров 65 °C, зонда — на 2-5 °C выше. Наличие или отсутствие вторичных структур и температуру отжига проверяли с помощью онлайн приложений для анализа праймеров Oligo Calc: Oligonucleotide Properties Calculator (http://biotools.nubic.northwestern.edu/OligoCalc.html), ThermoFisher Multiple Primer Analyzer (https://www.thermo-fisher.com), Promega Biomath Calculators (буфер 5×PCR Buffer с концентрацией MgCl2 3 мM) (https://worldwide.promega.com/resources/tools/biomath/tm-calculator/). В качестве флуоресцентной метки в гибридизационных зондах для ПЦР-РВ использовали красители 6FAM и 6ROX, присоединенные к 5'-концу зонда. Гасителями флуоресценции служили красители RTQ1 и BHQ2, присоединенные к 3'-концу зонда. Концентрация праймеров в реакционной смеси составляла 450 нМ, зондов — 200 нМ. ПЦР-РВ проводили на четырех приборах — АНК-32 («ИАП РАН», Россия) (26), CFX-96 («Bio-Rad», США), Rotor-Gene 6000 («QiaGen», США), DTprime 5 («ДНК-Технология», Россия). Использовали следующую программу амплификации: 5 мин при 95 °C; 15 с при 95 °C, 40 с при 60 °C (50 циклов). За положительный результат принимали образцы, интенсивность сигнала флуоресценции которых превышала пороговую линию (устанавливается как 10 % от разницы модулей самого высокого и самого низкого уровня флуоресценции).
Для секвенирования ДНК Candidatus Phytoplasma solani была сконструирована пара праймеров SolaSeq_F 5'-AACTTAACCTTTTAACTAGG-GC-3' и SolaSeq_R 5'-CATCAAGGCATTTGCC-3'. Программа амплификации для проведения классической ПЦР: 5 мин при 95 °C; 20 с при 95 °C, 20 с при 60 °C, 1 мин при 72 °C (36 циклов); 5 мин при 72 °C. Секвенирование ДНК осуществляли на приборе Нанофор 05 («Институт аналитического приборостроения РАН», Россия). Данные анализировали с помощью программного обеспечения ДНК Анализ версия 5.0.3.2 («Институт аналитического приборостроения РАН», Россия).
Биоинформационный анализ и обработку данных проводили с помощью программного обеспечения UGENE («УНИПРО», Россия) и AliView (NBIS, Department of Cell and Molecular Biology, Uppsala University, Швеция). Поиск последовательностей ДНК-мишеней осуществляли в базе данных NCBI GenBank (http://www.ncbi.nlm.nih.gov/BLAST) (27).
ДНК-мишенью для разработанных праймеров и зонда служил ген SecY (длина целевого фрагмента 119 п.н.) под номером KU600099.1 в базе данных NCBI GenBank (http://www.ncbi.nlm.nih.gov/BLAST) (позиции с 513 до 632 п.н.) (27).
Для определения аналитической чувствительности набора реагентов была создана векторная конструкция на основе плазмиды Pal2T («Евроген», Россия) со вставкой целевой нуклеотидной последовательности Candidatus Phytoplasma solani размером 119 п.н. Лигирование проводили 24 ч при 4 °С с использованием фермента Т4 ДНК лигазы («ThermoFisher Scientific», США). Трансформацию бактерии Escherichia coli (Migula 1895) осуществляли методом теплового шока. Наличие вектора проверяли методом ПЦР-колонии с использованием стандартных праймеров М13. Плазмиду выделяли с помощью набора реагентов PlasGen (ООО «Синтол», Россия). Для увеличения эффективности ПЦР кольцевую плазмиду обрабатывали рестриктазой NotI («ThermoFisher Scientific», США). Концентрацию ДНК плазмиды измеряли на флуориметре Quantus («Promega Corporation», США) в 3-кратной повторности. Для определения чувствительности разработанного набора реагентов проводили ПЦР-РВ с серией 10-кратных разведений плазмиды с известной концентрацией в 2-кратной повторности (28).
Специфичность набора реагентов проверяли на 37 образцах близкородственных и сопутствующих объектов: Vitis vinifera, Xanthomonas campestris, Cercospora beticola, Fusarium culmorum, Fusarium sp., Fusarium tricinum, Gibellina cerealis, Botrytis cinerea, Pleospora betae, Phomopsis helianthi, Pseudomonas fluorescens, Alternaria tenuissima, Aspergillus niger (коллекция ООО «Синтол», Россия); Candidatus Phytoplasma vitis (20,9 Ct), Candidatus Phytoplasma rubi (18,9 Ct), Candidatus Phytoplasma pyri (21,4 Ct), Candidatus Phytoplasma mali (18,4 Ct), Candidatus Phytoplasma convolvuli (15,9 Ct), Candidatus Phytoplasma asteris (15,0 Ct), Candidatus Phytoplasma cirsii (33,5 Ct), Candidatus Phytoplasma taraxacum (14,0 Ct), Candidatus Liberibacter solanacearum (23,6 Ct), Xanthomonas oryzae 0227, Cercospora kikuchii (коллекция ФГБУ ВНИИКР, Россия); Xanthomonas euvesicatoria DSM 19128, Xanthomonas gardneri DSM 19127, Xanthomonas perforans DSM 18975, Xanthomonas vesicatoria DSM 22252, Xanthomonas translucens pv. translucens DSM 18974 (Leibniz Institute DSMZ-German Collection of Microorganisms and Cell Cultures GmbH, Германия); Xanthomonas phaseoli CFBP 2534 (CIRM-CFBP, Франция); Fusarium sambucinum F139, Fusarium graminearum F877, Fusarium verticillioides F43, Fusarium avenaceum F623 (Всероссийская коллекция промышленных микроорганизмов, НИЦ Курчатовский институт — ГосНИИгенетика, г. Москва); Fusarium oxysporum F840, Phytophthora cinnamomi F3332, F3333, Phytophthora cactorum 985 (Всероссийская коллекция микроорганизмов, Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина РАН, г. Пущино, Московская обл.).
Для постановки ПЦР-РВ ДНК винограда, бактерий и грибов вносили в концентрации 50 нг на реакцию. Пороговый цикл образцов Candidatus Phytoplasma sp. определяли, используя праймеры UniRT (13), для Candidatus Liberibacter solanacearum применяли праймеры, рекомендованные EPPO (14).
Результаты. В таблице 1 представлены праймеры и зонды, опубликованные E. Angelini с соавторами и C. Pelletier с соавторами и используемые в настоящее время (1, 11, 16, 29, 30), а также разработанные нами праймеры и зонд для ПЦР-РВ (BN).
Для сравнения аналитической чувствительности рекомендованных и разработанных нами систем была проведена ПЦР-РВ с серией 10-кратных разведений ДНК Candidatus Phytoplasma solani. Хотя по пороговому циклу праймеры BNrt не уступали разработанным нами, образец с разведением 1:100 не дал положительного результата, что указывает на меньшую чувствительность BNrt по сравнению с BN. Сигнал флуоресценции положительных образцов при использовании BN и BNrt был соответственно 1500 и 500 относительных единиц флуоресценции (ОЕФ). В обоих случаях концентрация праймеров в реакционной смеси составляла 450 нМ, зондов — 200 нМ. Разница в пороговых циклах у mapBN с BNrt и BN составила +3,0-3,5 Ct. Помимо более низкой чувствительности системы mapBN,при разведении 1:100 наблюдалось полное отсутствие роста сигнала флуоресценции (рис. 1).
Для определения специфичности ПЦР с использованием праймеров BNrt в программе AliView провели выравнивание целевых фрагментов, на которые были подобраны праймеры и зонд для выявления Candidatus Phytoplasma solaniметодом ПЦР-РВ. В базе данных NCBI было найдено более 100 фрагментов ДНК, 69 из которых относились к Candidatus Phytoplasma solani. Их идентичность с целевым фрагментом для праймеров BNrt (16) составляла от 98 до 100 %. Выравнивание показало 100 % совпадение последовательностей посадки праймеров и зондов. Эта система, помимо Candidatus Phytoplasma solani, выявляла и другие фитоплазмы группы 16SrXII, которые вызывают заболевания witches’ broom и potato purple top (31, 32) на картофеле. В соответствии с биоинформационным анализом система праймеров и зонда BNrt для ПЦР-РВ не обеспечивала высокую специфичность выявления ДНК Candidatus Phytoplasma solani
Целевой ген для праймеров mapBN — adk. Выравнивание последовательностей показало высокую специфичность посадки праймеров и зонда, однако в базе данных GenBank NCBI (http://www.ncbi.nlm.nih.gov/BLAST) было найдено всего 10 последовательностей этого гена, 7 из которых принадлежали Candidatus Phytoplasma solani. Из-за малой выборки данных праймеры mapBN также были отвергнуты нами в качестве кандидатов для использования в разрабатываемом наборе реагентов.
В результате в качестве мишени для оценки специфичности выявления ДНК Candidatus Phytoplasma solaniмы выбрали ген SecY, кодирующий белок, который принимает участие в трансмембранном транспорте. Этот ген рекомендован EPPO для диагностики фитоплазмы почернения древесины (24). Анализ последовательностей SecY в GenBank NCBI обнаружил 190 последовательностей, в том числе 110 штаммов Candidatus Phytoplasma solani. Места посадки разработанных нами праймеров и зонда для ПЦР-РВ (BN) оказались строго специфичными и позволяли обнаруживать все штаммы целевого объекта Candidatus Phytoplasma solani, последовательности ДНК которых находились в базе данных GenBank NCBI Nucleotide на 16.01.2019 (27).
Результаты секвенирования ДНК образцов для подтверждения видовой принадлежности Candidatus Phytoplasma solani,проведенного с помощью праймеров на ген SecY для шести образцов винограда, предположительно зараженного Candidatus Phytoplasma solani,представлены на рисунке 2.
Аналитическую специфичность набора реагентов определяли на выборке из 37 образцов близкородственных и сопутствующих организмов и шести образцов винограда, предположительно зараженного Candidatus Phytoplasma solani по данным визуального анализа. При определении аналитической специфичности положительные результаты получили для всех образцов, содержащих ДНК Candidatus Phytoplasma solani, что было подтверждено секвенированием ДНК с помощью праймеров SolaSeq_F и SolaSeq_R. Ложноположительных результатов при внесении ДНК фитоплазм других видов, в том числе с Candidatus phytoplasmaconvolvuli, которая, как и Candidatus Phytoplasma solani, относится к группе 16SrXII, и сопутствующих организмов, мы не выявили.Аналитическая специфичность праймеров и зонда для ПЦР-РВ на ген SecY в исследованной выборке из 37 образцов составила 100 %.
Аналитическую чувствительность набора реагентов оценивали посредством разведения ДНК плазмиды с целевой вставкой Candidatus Phytoplasma solani. Исходная концентрация плазмиды составляла 2×107 копий/мкл. При постановке каждую реакцию дублировали. Рассчитанное значение эффективности ПЦР-РВ для серии из шести 10-кратных разведений плазмиды от 107 до 102 копий в реакции составило E = 98,7 %, наклон кинетической кривой А = -3,355, коэффициент корреляции R2 = 0,999. Начиная с количества 50 копий плазмиды в реакции, ПЦР-РВ проводили в 24 повторах в разведениях 50, 20, 15, 10, 5 и 1 копия плазмиды в реакции. При разведении до 10 копий в реакции результат был нестабильным, а при разведении до 1 копии плазмиды в реакции специфичного сигнала ПЦР-РВ не наблюдали. Чувствительность набора реагентов устанавливали, если минимум 95 % реакций давали положительный результат. Аналитическая чувствительность набора реагентов с использованием праймеров BN составила не менее 15 копий в реакции.
Для апробации набора реагентов для выявления ДНК Candidatus Phytoplasma solaniпроанализировали 194 образца винограда из шести мест произрастания (хозяйства Южнобережного агроклиматического района полуострова Крым). Все исследованные образцы дали положительные результаты на наличие ДНК фитоплазмы CandidatusPhytoplasma solani (рис. 3). Полученные данные подтвердило секвенирование ДНК.
При использовании разработанного набора реагентов результаты были подтверждены на четырех приборах для ПЦР-РВ (табл. 2). Диапазон значений эффективности ПЦР-РВ составил E = 97-100 %, наклон кинетической кривой А = 3,31-3,39, коэффициент корреляции R2 = 1,000. Отличия в пороговых значениях в среднем составили ±2 цикла, что связано с разницей в строении оптических модулей, скорости нагрева и охлаждения реакторов, индивидуальным для каждого прибора программным обеспечением и алгоритмом определения значений порогового цикла.
Для моделирования праймеров и зонда в нашей работе был выбран ген SecY, кодирующий субъединицу белка транслоказы, который характеризуется высокой консервативностью и специфичностью, позволяющей дифференцировать Candidatus Phytoplasma solani от близкородственных видов. По мнению D.L. Davies с соавт. (9) и I.-M. Lee с соавт. (10), этот ген — лучший маркер для видовой дифференциации и классификации штаммов Candidatus Phytoplasma solani. Таргетными генами у существующих и используемых в настоящее время систем диагностики служат участки генов 16SrRNA и adk (15). Ген рибосомальной РНК уступает в специфичности как гену, кодирующему аденозин-киназу (adk), так и SecY. Аналитическая чувствительность разработанного нами набора реагентов оказалась в 10 раз выше по сравнению с рекомендованными системами диагностики и составила не менее 15 копий в реакции.
Успешная апробация предлагаемого набора реагентов на четырех приборах для ПЦР в реальном времени как российского, так и зарубежного производства позволяет рекомендовать эту систему для диагностических и скрининговых анализов в исследовательских лабораториях.
Таким образом, разработан набор реагентов для выявления участка гена SecY фитоплазмы Candidatus Phytoplasma solani, вызывающей заболевание почернения древесины, методом полимеразной цепной реакции в реальном времени (ПЦР-РВ). Анализ полевых образцов листьев, корней и фрагментов лозы винограда, предположительно инфицированных фитоплазмой Candidatus Phytoplasma solani,с помощью предложенного набора показал наличие ДНК этого возбудителя во всех образцах. Данные ПЦР-РВ были подтверждены секвенированием ДНК. ПЦР с использованием разработанного набора реагентов обладает высокой чувствительностью и специфичностью и позволяет проводить эпифитотиологические исследования, а также изучение сортоустойчивости к возбудителю почернения древесины винограда.
1. Последовательности праймеров и зондов, используемые для выявления Candidatus Phytoplasma solani, и соответствующие им режимы амплификации |
||||
Название, |
Мишень |
Режим |
Последовательность |
|
BNrt (16) |
16SrRNA |
5 мин при 95 °C; |
F |
5'-GGTTAAGTCCCGCAACGAG-3' |
R |
5'-CCCACCTTCCTCCAATTATCA-3' |
|||
Pb |
5'-(6FAM)AACCCTTGTTGTTAATTGCCATCATTAAG(RTQ1)-3' |
|||
mapBN (29) |
adk |
5 мин при 95 °C; |
F |
5'-ATTTGATGAAACACGCTGGATTAA-3' |
R |
5'-TCCCTGGAACAATAAAAGTYGCA-3' |
|||
Pb |
5'-(6ROX)AAACCCACAAAATGC(BHQ2)-3' |
|||
BN (собственные данные) |
SecY |
5 мин при 95 °C; |
F |
5'-AATACCAGTACAATACGCTCGC-3' |
R |
5¢-AAAGGTTGCATCAAGGCATTTGC-3¢' |
|||
Pb |
5'-(6FAM)AACACTGCTGGAGTAATGCCTGTAATT(RTQ1)-3' |
|||
Примечание. F — прямой праймер, R — обратный праймер, Pb — зонд. |
||||
2. Воспроизводимость результатов ПЦР в реальном времени на приборах разных производителей при использовании |
||||
Образцы, 10-кратное разведение |
Пороговый цикл, Ct |
|||
CFX96 |
RG 6000 |
DTprime M1 |
АНК 32 |
|
10-1 |
18,61 |
18,37 |
16,2 |
19,21 |
10-1 |
18,73 |
18,26 |
16,2 |
19,27 |
10-2 |
22,05 |
21,57 |
19,3 |
22,61 |
10-2 |
21,94 |
21,72 |
19,3 |
22,75 |
10-3 |
25,51 |
24,98 |
23,0 |
25,77 |
10-3 |
25,45 |
24,92 |
22,8 |
26,05 |
10-4 |
28,75 |
28,37 |
26,0 |
29,24 |
10-4 |
28,66 |
28,18 |
26,1 |
29,10 |
10-5 |
32,05 |
31,34 |
29,4 |
32,53 |
10-5 |
32,46 |
31,46 |
29,1 |
32,57 |
Отрицательный контрольный образец |
N/A |
N/A |
N/A |
N/A |
Отрицательный контрольный образец |
N/A |
N/A |
N/A |
N/A |
Наклон кинетической кривой, A |
3,39 |
3,32 |
3,32 |
3,31 |
Коэффициент корреляции, R2 |
1,000 |
1,000 |
1,000 |
1,000 |
Значение эффективности ПЦР-РВ, Е |
97 |
100 |
100 |
100 |
Примечание. N/Anot available, отсутствует. |
||||
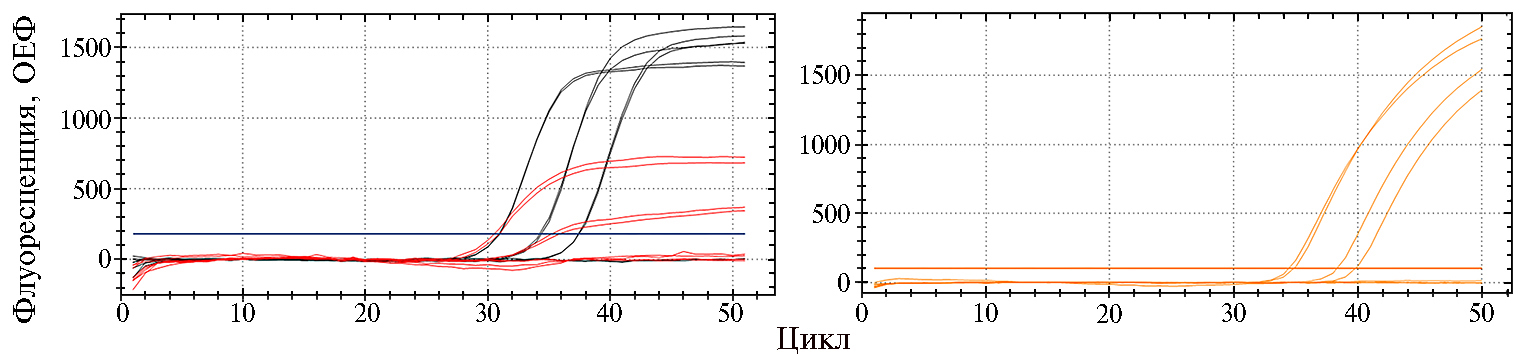
Рис 1. Результаты полимеразной цепной реакции в реальном времени (ПЦР-РВ) для серии из трех 10-кратных разведений ДНК Candidatus Phytoplasma solani с использованием различных систем праймеров и зондов: BN — черный,BNrt — красный, mapBN — оранжевый цвет (ОЕФ — относительные единицы флуоресценции, прибор CFX-96, «Bio-Rad», США).

Рис. 2. Выравнивание последовательностей ДНК Candidatus Phytoplasma solaniиз базы данных GenBank NCBI (http://www.ncbi.nlm.nih.gov/BLAST) с последовательностями, полученными в результате секвенирования ДНК зараженных образцов винограда с помощью праймеров на ген SecY. Выравнивания получены в программе Unipro UGENE (Россия) версия 1.31.1, белой рамкой выделена зона посадки прямого праймера BN_F. Phytoplasma solani 1-6 — образцы, собранные в шести различных местах произрастания. Полностью рисунок см. на сайте http://www.agrobiology.ru.
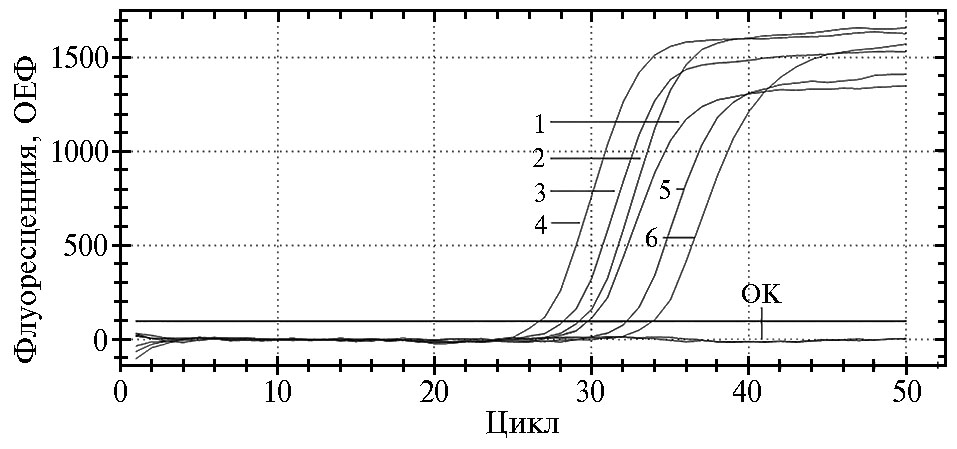
Рис. 3. Результат полимеразной цепной реакции в реальном времени (ПЦР-РВ) при анализе шести образцов винограда, собранных в различных местах произрастания (1-6), на наличие ДНК Candidatus Phytoplasma solaniс использованием разработанного набора реагентов для выявления участка гена SecY(ОК — отрицательный контроль; канал детекции FAM, ОЕФ — относительные единицы флуоресценции, прибор CFX-96, «Bio-Rad», США).
ЛИТЕРАТУРА
- Матяшова Г.Н., Заец В.Г. Исследование метода ПЦР в режиме «реального времени» для обнаружения и идентификации возбудителей фитоплазмозов винограда. Вестник Российского университета дружбы народов. Серия: Агрономия и животноводство, 2015, 4: 7-14.
- Рисованная В.И., Гориславец С.М., Володин В.А. Тестирование латентной стадии фитоплазменной инфекции винограда. Магарач. Виноградарство и виноделие, 2013, 4: 6-8.
- Doi Y., Teranaka M., Yora K., Asuyama H. Mycoplasma- or PLT group-like microorganisms found in the phloem elements of plants infected with mulberry dwarf, potato witches’ broom, aster yellows, or Paulownia witches’ broom. Annals Phytopathological Society, 1967, 33(4): 259-266 (doi: 10.3186/jjphytopath.33.259).
- Duduk B., Bertaccini A. Phytoplasma classification: taxonomy based on 16S ribosomal gene, is it enough? Phytopathogenic Mollicutes,2011, 1(1): 3-13 (doi: 10.5958/j.2249-4669.1.1.001).
- Deng S., Hiruki C. Amplification of 16S rRNA genes from culturable and nonculturable Mollicutes. Journal of Microbiological Methods, 1991, 14(1): 53-61 (doi: 10.1016/0167-7012(91)90007-D).
- Lee I.M., Davis R.E., Hiruki C. Genetic interrelatedness among clover proliferation mycoplasma-like organisms (MLOs) and other MLOs investigated by nucleic acid hybridization and restriction fragment length polymorphism analyses. Applied and Environmental Microbiology, 1991, 57(12): 3565-3569.
- Duduk B., Perić P., Marčić D., Drobnjaković T., Picciau L., Alma A., Bertaccini A. Phytoplasmas in carrots: disease and potential vectors in Serbia. Bulletin of Insectology, 2008, 61(2): 327-331.
- The IRPCM Phytoplasma/Spiroplasma Working Team — Phytoplasma taxonomy group. ‘Candidatus Phytoplasma’, a taxon for the wall-less, non-helical prokaryotes that colonize plant phloem and insects. Internation Journal of Systematic and Evolutionary Microbiology, 2004, 54(4): 1243-1255 (doi: 10.1099/ijs.0.02854-0).
- Davies D.L., Lee I.-M. Cluster-specific polymerase chain reaction amplification of 16S rDNA sequences for detection and identification of mycoplasmalike organisms. Phytopathology, 1993, 83(7): 1008-1011 (doi: 10.1094/Phyto-83-1008).
- Lee I.-M., Gundersen-Rindal D.E., Davis R.E., Bartoszyk I.M. Revised classification scheme of phytoplasmas based on RFLP analyses of 16S rRNA and ribosomal protein gene sequences. Internation Journal of Systematic and Evolutionary Microbiology, 1998, 48(4): 1153-1169 (doi: 10.1099/00207713-48-4-1153).
- Матяшова Г.Н., Заец В.Г. Исследование метода ПЦР в режиме реального времени для обнаружения и идентификации возбудителей фитоплазмозов винограда. Вестник Российского университета дружбы народов. Серия: Агрономия и животноводство, 2015, 4: 7-14.
- PM 7/133 (1) Generic detection of phytoplasmas. EPPO Bulletin, 2018, 48(3): 414-424 (doi: 10.1111/epp.12541).
- Hren M., Boben L., Rotter A., Kralj P., Gruden K., Ravnikar M. Real-time PCR detection systems for Flavescence dorée and Bois noir phytoplasmas in grapevine: comparison with conventional PCR detection and application in diagnostics. Plant Pathology, 2007, 56(5): 785-796 (doi: 10.1111/j.1365-3059.2007.01688.x).
- Li W., Hartung J.S., Levy L. Quantitative real-time PCR for detection and identification of Candidatus Liberibacter species associated with citrus huanglongbing. Journal of Microbiological Methods, 2006, 66(1): 104-115 (doi: 10.1016/j.mimet.2005.10.018).
- PM 7/079 (2) Grapevine flavescence dorée phytoplasma EPPO Bulletin,2016, 46(1): 78-93 (doi: 10.1111/epp.12280).
- Angelini E., Bianchi G.L., Filippin L., Morassutti C., Borgo M. A new TaqMan method for the identification of phytoplasmas associated with grapevine yellows by real-time PCR assay. Journal of Microbiological Methods,2007, 68(3): 613-622 (doi: 10.1016/j.mimet.2006.11.015).
- Berger J., Via J.D., Baric S. Development of a TaqMan allelic discrimination assay for the distinction of two major subtypes of the grapevine yellows phytoplasma Bois noir. European Journal of Plant Pathology, 2009, 124(3): 521-526 (doi: 10.1007/s10658-008-9424-9).
- Единый перечень карантинных объектов Евразийского экономического союза. С изменениями и дополнениями от 30 марта 2018 г. (изменения от 2 мая 2018 г. — Решение Совета Евразийской экономической комиссии от 30 марта 2018 г. N 25). Режим доступа: https://vniikr.ru/edinyij-perechen-karantinnyix-obektov-evrazijskogo-ekonomicheskogo-soyuza. Дата обращения: 14.04.2019.
- Choueiri E., Jreijiri F., El Zammar S., Verdin E., Salar P., Danet J.L., Bové J., Garnier M. First report of grapevine “Bois Noir” disease and a new phytoplasma infecting solanaceous plants in Lebanon. Plant Disease, 2002, 86(6): 697-697 (doi: 10.1094/PDIS.2002.86.6.697A).
- Franova J. Difficulties with conventional phytoplasma diagnostic using PCR/PFLP analysis. Bulletin of Insectology, 2011, 64(Suppl.): S287-S288.
- Galetto L., Bosco D., Merzachi C. Universal and group-specific real-time PCR diagnosis of flavescence dorée (16Sr-V), bois noir (16Sr-XII) and apple proliferation (16Sr-X) phytoplasmas from field-collected plant hosts and insect vectors. Annals of Applied Biology, 2005, 147(2): 191-201 (doi: 10.1111/j.1744-7348.2005.00030.x).
- Plavec J., Križanac I., Budinšćak Ž., Škorić D., Music M.S. A case study of FD and BN phytoplasma variability in Croatia: multigene sequence analysis approach. European Journal of Plant Pathology, 2015, 142(3): 591-601 (doi: 10.1007/s10658-015-0637-4).
- Богоутдинов Д.З., Кастальева Т.Б., Гирсова Н.В. Фитоплазменные болезни — серьезная опасность для растениеводства России. Обзор. Таврический вестник аграрной науки, 2018, 2(14): 15-34 (doi: 10.25637/TVAN.2018.02.02).
- European and Mediterranean Plant Protection Organization. Режим доступа:https://www.eppo.int. Дата обращения: 11.02.2019.
- Doyle J.J., Doyle J.L. Isolation of plant DNA from fresh tissue. Focus, 1990, 12: 13-15.
- Алексеев Я.И., Белов Ю.В., Варламов Д.А., Коновалов С.В., Курочкин В.Е., Маракушин Н.Ф., Петров А.И., Петряков А.О., Румянцев Д.А., Скоблилов Е.Ю., Соколов В.Н., Фесенко В.А., Чернышев А.В. Приборы для диагностики биологических объектов на основе метода полимеразной цепной реакции в реальном времени (ПЦР-РВ). Научное приборостроение, 2006, 16(3): 132-136.
- National Center for Biotechnology Information. Режим доступа: http://www.ncbi.nlm.nih.gov. Дата обращения: 16.01.2019.
- Аляпкина Ю.С., Моисеева М.В., Ксенофонтова О.В., Алексеев Я.И. Разработка и валидация набора для мультиплексного ПЦР-РВ анализа регуляторных элементов (промотора SsuAra и терминатора E9) для обнаружения генетически модифицированных (ГМ) линий рапса, сои, картофеля и других растений. Известия ТСХА, 2008, 3: 5-16 (doi: 10.26897/0021-342X-2018-3-5-16).
- Pelletier C., Salar P., Gillet J., Cloquemin G., Very P., Foissac X., Malembic-Maher S. Triplex real-time PCR assay for sensitive and simultaneous detection of grapevine phytoplasmas of the 16SrV and 16SrXII-A groups with an endogenous analytical control. Vitis, 2009, 48(2): 87-95.
- Quaglino F., Zhao Y., Casati P., Bulgari D., Bianco P.A., Wei W., Davis R.E. ‘Candidatus Phytoplasma solani’, a novel taxon associated with stolbur- and bois noir- related diseases of plants. International Journal of Systematic and Evolutionary Microbiology, 2013, 63: 2879-2894 (doi: )10.1099/ijs.0.044750-0.
- Yu X.M., Ai C.X., Wang J., Fu L., An M., Wang H.R., Sun Q.R. First report of a 16SrXII-A subgroup phytoplasma associated with Artemisia scoparia witches’-broom disease in China. Plant Disease, 2016, 100(7): 1494 (doi: 10.1094/PDIS-11-15-1251-PDN).
- Girsova N.V., Bottner K.D., Mozhaeva K.A., Kastalyeava T.B., Owens R.A., Lee I.M. Molecular detection and identification of Group 16SrI and 16SrXII phytoplasmas associated with diseased potatoes in Russia. Plant Disease, 2008, 92(4): 654 (doi: 10.1094/PDIS-92-4-0654A).